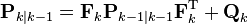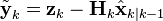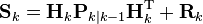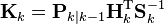R ggplot2 で線形回帰/一般化線形モデルの残差プロット
ggplot2 で出力している他のグラフとデザインを揃えたかったので作ってみた。
インストール
library(devtools) install_github('sinhrks/ggfortify')
サンプル
デフォルトの plot と (ほぼ) 同じ見た目にしたつもり。
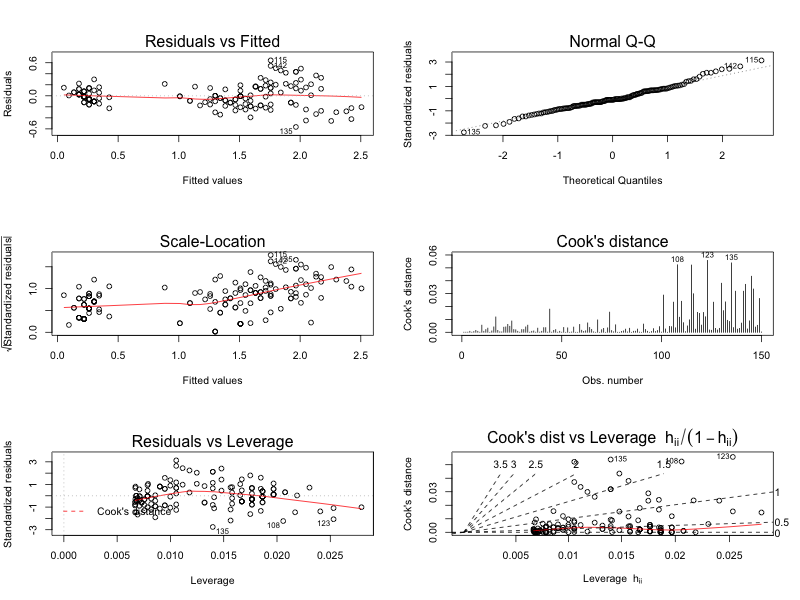
library(ggplot2) library(ggfortify) m <- lm(Petal.Width ~ Petal.Length, data = iris) autoplot(m, which = 1:6)
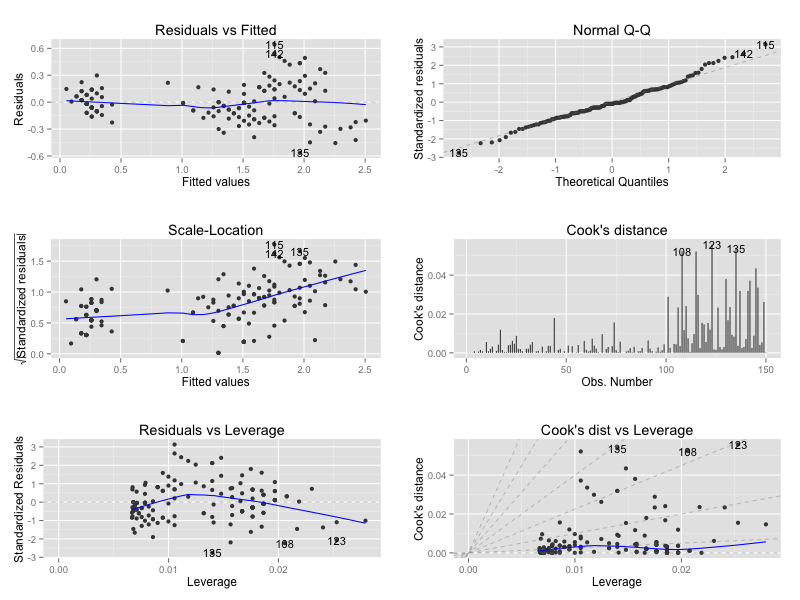
通常の残差プロット
一般化線形モデル
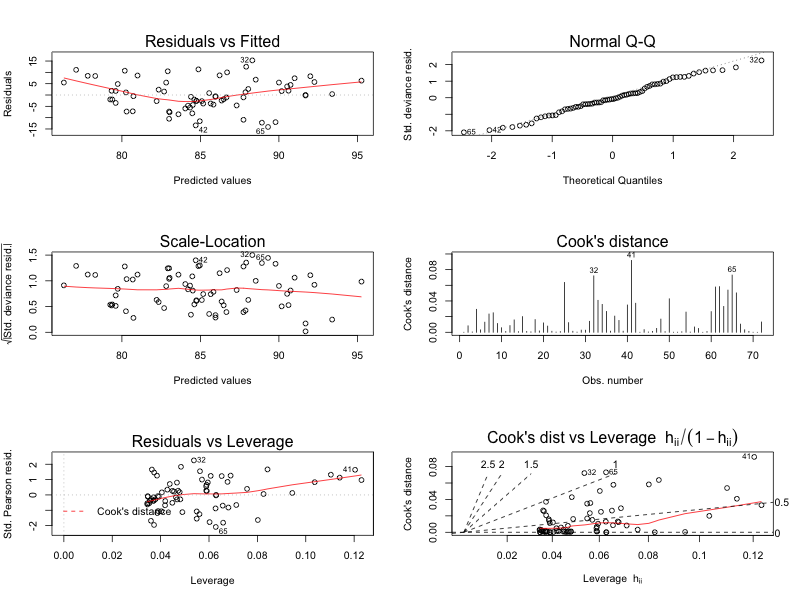
data(anorexia, package = "MASS") m <- glm(Postwt ~ Prewt + Treat + offset(Prewt), family = gaussian, data = anorexia) autoplot(m, which = 1:6)
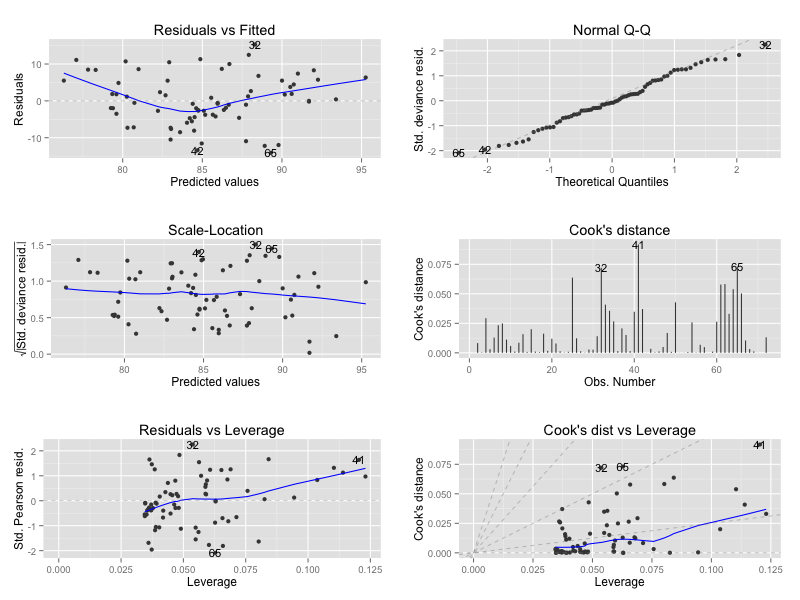
通常の残差プロット
設定できるオプション
which: 描画対象のグラフ (1〜6まで、通常のplotと同じ)colour: データの色label.colour: ラベルの色label.size: ラベルの文字サイズlabel.n: ラベルを表示する数smooth.colour: 平滑線の色smooth.linetype: 平滑線のスタイルad.colour: 補助線の色ad.linetype: 補助線のスタイルad.size: 補助線のサイズnrow: 複数プロットする際の行数ncol: 複数プロットする際の列数

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
R {seasonal} パッケージで X13-ARIMA-SEATSを使う
X13-ARIMA-SEATS とは
米国国勢調査局 (Census Bureau)で開発している 時系列処理/季節調整プログラム。日本の統計データも 前身バージョンの X12-ARIMA を利用して季節調整しているのをよく見かける。X13 では これまでの季節調整 (X12-ARIMA) に加え、欧州でよく使われている季節調整法である TRAMO-SEATS も利用できるようになった。
(手法自体はちゃんと理解できていないのだが) X12-ARIMA は RegARIMA + 移動平均, SEATS は Wienerフィルタを使って季節性を検出しているらしい。
季節調整法に関する最近の動向:X-12-ARIMAからX-13ARIMA-SEATSへ
X13-ARIMA-SEATS のインストール
Winows
リンク先の winx13.zip をダウンロードして解凍するだけ。
Download Win X-13 - X-13ARIMA-SEATS Seasonal Adjustment Program - US Census Bureau
Mac
ソースからコンパイルするには g77 が必要。gretl のサイトに Mac のバイナリへのリンクがあったので、そっち使う。
http://sourceforge.net/projects/gretl/files/x13as/x13as-x86_64-darwin10.dmg/download
まずはふつうに実行
X13-ARIMA-SEATS はかなり硬派なツールで、入力は独自のフォーマットの .spc ファイルとして渡す必要がある。
Windows
ドキュメント参照 (自分は環境もってないので、、)
Win X-13 Documentation - X-13ARIMA-SEATS Seasonal Adjustment Program - US Census Bureau
Mac
下の tar に入っている testairline.spc ファイルで試す。中身は R の AirPassengers データと同じもの。
https://www.census.gov/ts/x13as/unix/x13asall.tar.gz
実行するには、シェルから以下のコマンドを実行。サンプルファイルを指定する際、拡張子の .spc はいらない。
/Applications/x13as/bin/x13as -i /Users/sin/Downloads/x13asall/testairline # X-13ARIMA-SEATS Seasonal Adjustment Program # Version Number 1.1 Build 9 PSP = 24 # Execution began Sat Nov 8 22:35:55 2014 # # Reading input spec file from /Users/sin/Downloads/x13asall/testairline.spc # Storing any program output into /Users/sin/Downloads/x13asall/testairline.out # Storing any program error messages into /Users/sin/Downloads/x13asall/testairline.err # # # WARNING: At least one visually significant trading day peak has been # found in one or more of the estimated spectra. # # Execution complete for /Users/sin/Downloads/x13asall/testairline.spc at Sat Nov 8 22:35:55 2014
実行結果は 同名の .out ファイルに出力される。一部 抜粋するが、モデルのフィッティング結果 / 予測値なんかが入っている。
less /Users/sin/Downloads/x13asall/testairline.out # ... # # ARIMA Model: (0 1 0)(0 1 1) # Nonseasonal differences: 1 # Seasonal differences: 1 # # ... # # Confidence intervals with coverage probability (0.95000) # On the Original Scale Before Prior Adjustments # --------------------------------------- # Date Lower Forecast Upper # --------------------------------------- # 1961.Jan 418.96 442.42 467.19 # 1961.Feb 381.63 412.13 445.06 # 1961.Mar 421.68 463.41 509.26 # 1961.Apr 445.18 496.35 553.40 # 1961.May 439.00 495.82 559.99 # 1961.Jun 501.51 572.95 654.57 # 1961.Jul 579.38 668.98 772.43 # 1961.Aug 558.90 651.83 760.22 # 1961.Sep 471.65 555.17 653.48 # 1961.Oct 411.72 488.94 580.65 # 1961.Nov 351.21 420.60 503.71 # 1961.Dec 395.21 477.08 575.92 # ---------------------------------------
これはいちいち実行 & 確認がめんどくさいぞ、、。ということで R から使えるようにする。
{seasonal} パッケージのインストール
seasonal というパッケージを使うと、 R から X13-ARIMA-SEATS を呼び出して利用することができる。まずはパッケージをインストール。
install.packages('seasonal') library(seasonal) # No path to the binary executable of X-13 specified. # # For installation details, consider Section 2 of the package vignette: # http://cran.r-project.org/web/packages/seasonal/vignettes/seas.pdf
ロードすると上記とおり X13-ARIMA-SEATS のバイナリのパスが指定されていないよ!という警告がでる。このままでは使えないので、 vignette に従ってパスを指定する。
補足 Windows の場合は WinX13.exe ではなく x13as フォルダ (x13as.exeを含むフォルダ) へのパスを指定することに注意。 指定は <ツールのパス>\\winx13\\x13as になるはず。
Sys.setenv(X13_PATH = "/Applications/x13as/bin") checkX13() # Congratulations! 'seasonal' should work fine! # - the X13_PATH is correctly specified # - the binary executable file has been found # - a test run has been successful # # seasonal now supports the HTML version of X13, which offers a more # accessible output via the out() function. For best user experience, # copy the binary executables of x13ashtml to: # # /Applications/x13as/bin
R から X13-ARIMA-SEATSを実行
AirPassengers データでモデルのフィット + 予測実行してみる。
# モデルのフィット m <- seas(AirPassengers) # フィッティング結果は $data プロパティから取得できるが、このプロパティはすでに deprecate されているため使わないほうがよい head(m$data) # final seasonal seasonaladj trend irregular adjustfac # [1,] 122.7133 0.9019909 122.7133 122.9732 0.9978869 0.9126963 # [2,] 124.7657 0.9542175 124.7657 124.3148 1.0036268 0.9457731 # [3,] 125.0734 1.0698054 125.0734 125.5828 0.9959432 1.0553807 # [4,] 127.5286 1.0023238 127.5286 126.7107 1.0064547 1.0115378 # [5,] 127.3584 0.9486743 127.3584 126.9446 1.0032599 0.9500745 # [6,] 126.1729 1.0762915 126.1729 126.2800 0.9991512 1.0699607 # 代わりに、同じデータを $series から作る df <- data.frame(final = m$series$s11, seasonal = m$series$s10, seasonaladj = m$series$s11, trend = m$series$s12, irregular = m$series$s13, adjustfac = m$series$s16) head(df) # final seasonal seasonaladj trend irregular adjustfac # 2 122.7133 0.9019909 122.7133 122.9732 0.9978869 0.9126963 # 3 124.7657 0.9542175 124.7657 124.3148 1.0036268 0.9457731 # 4 125.0734 1.0698054 125.0734 125.5828 0.9959432 1.0553807 # 5 127.5286 1.0023238 127.5286 126.7107 1.0064547 1.0115378 # 6 127.3584 0.9486743 127.3584 126.9446 1.0032599 0.9500745 # 7 126.1729 1.0762915 126.1729 126.2800 0.9991512 1.0699607 # 予測の実行 series(m, c('forecast.forecasts')) # specs have been added to the model: forecast # forecast lowerci upperci # Jan 1961 444.2964 418.1670 472.0585 # Feb 1961 413.5093 381.4051 448.3158 # Mar 1961 465.5498 422.4688 513.0240 # Apr 1961 497.5508 445.3449 555.8766 # May 1961 498.3558 440.6152 563.6630 # ... # Aug 1963 791.9620 502.8166 1247.3808 # Sep 1963 666.3004 417.7790 1062.6581 # Oct 1963 581.5833 360.2171 938.9870 # Nov 1963 510.9198 312.7342 834.6995 # Dec 1963 566.3870 342.6866 936.1157
ツールで実行した結果と違うのは .spc ファイルで行われているオプション指定をやってないからだと思う。
forecast の結果と比べてみる
とりあえずデフォルトの forecast::auto.arima で。
library(forecast) forecast(auto.arima(AirPassengers), 36) # Point Forecast Lo 80 Hi 80 Lo 95 Hi 95 # Jan 1961 446.7582 431.7435 461.7729 423.7953 469.7211 # Feb 1961 420.7582 402.5878 438.9286 392.9690 448.5474 # Mar 1961 448.7582 427.9043 469.6121 416.8649 480.6515 # Apr 1961 490.7582 467.5287 513.9877 455.2318 526.2846 # May 1961 501.7582 476.3745 527.1419 462.9372 540.5792 # ... # Aug 1963 695.2746 574.9455 815.6038 511.2471 879.3022 # Sep 1963 597.2746 473.0909 721.4584 407.3519 787.1973 # Oct 1963 550.2746 422.3523 678.1969 354.6344 745.9149 # Nov 1963 479.2746 347.7200 610.8292 278.0792 680.4700 # Dec 1963 521.2746 386.1853 656.3639 314.6734 727.8759
X13-ARIMA-SEATS と forecast の予測値の差異をプロットしてみた。ズレは手法によるものとオプション既定値によるものがありそうだが、詳細はまだよくわかってない。

まとめ
季節調整にこだわる場合は {seasonal} -> X13-ARIMA-SEATS で。
KalmanFilter の動きを可視化する 二次元版
こちらのつづき。一次元での動きはわかってきたので、今回は二次元でやってみる + KalmanFilter の予測 F を入れてみる。
二次元への拡張
まず以下の条件で考える。
- 二変数は
とし、それぞれ独立に動く
- 観測誤差は それぞれの成分で独立
- 真値の予測, システムの入力はなし (
,
)
- 真値の誤差は時間変化しない (
)
- 真値と観測値は同じ座標系 (
)
※アニメーション部分のコードはちょっと汚いので省略 / gist に添付。
set.seed(1) # 観測系列のサンプルサイズ n <- 100 # 真の値 x <- c(rep(0, n / 4), seq(0, 10, length.out = n / 4), rep(10, n / 4), seq(10, 0, length.out = n / 4)) y <- c(seq(0, 10, length.out = n / 4), rep(10, n / 4), seq(10, 0, length.out = n / 4), rep(0, n / 4)) actual <- cbind(x, y) # 観測系列の次元 dim <- ncol(actual) # 観測される値 (誤差は標準偏差0.1の独立正規分布とする) observed <- actual + matrix(rnorm(n * dim, sd = 0.1), ncol = dim) # 結果保存用のmatrix xhat <- matrix(0, nrow = n, ncol = dim) # 結果保存用の array # P[, , k] K[, , k] に 時刻 k 時点の計算結果を保存 P <- array(c(0.0, 0.1, 0, 0.1), dim = c(dim, dim, n)) K <- array(0, dim = c(dim, dim, n)) # 誤差 Q <- diag(0.01, nrow = dim, ncol = dim) R <- diag(0.01, nrow = dim, ncol = dim) # 単位行列 I <- diag(1, ncol = dim, nrow = dim) for (k in seq(2, n)) { # predict xhat.m <- xhat[k-1, ] P.m <- P[, , k-1] + Q # update S <- R + P.m K[, , k] <- P.m %*% solve(S) xhat[k, ] <- xhat.m + K[, , k] %*% (observed[k, ] - xhat.m) P[, , k] <- (I - K[, , k]) * P.m }
x, y の動きをプロットすると以下のようになる (赤が真の値、黒が観測値、青がKalmanFilterの予測値)。
- 上段のグラフは 二変数 x, y について平面上にプロットしたもの。
- 下段の2つのグラフは、 x, y それぞれが 時間 (time) に対してどのように動くかをプロットしたもの。
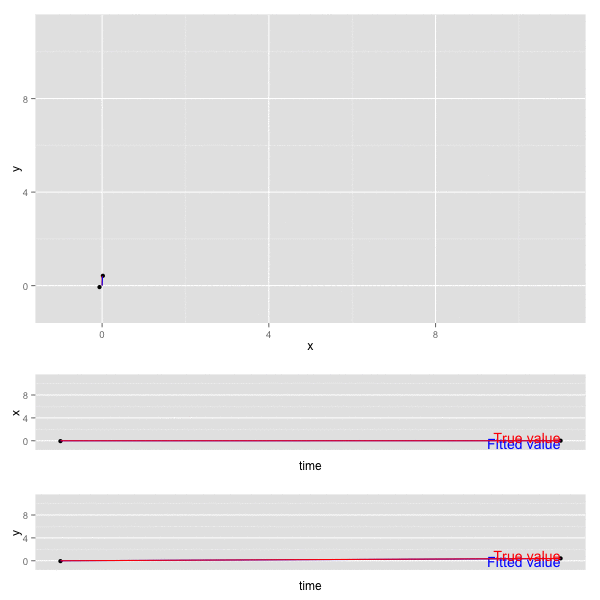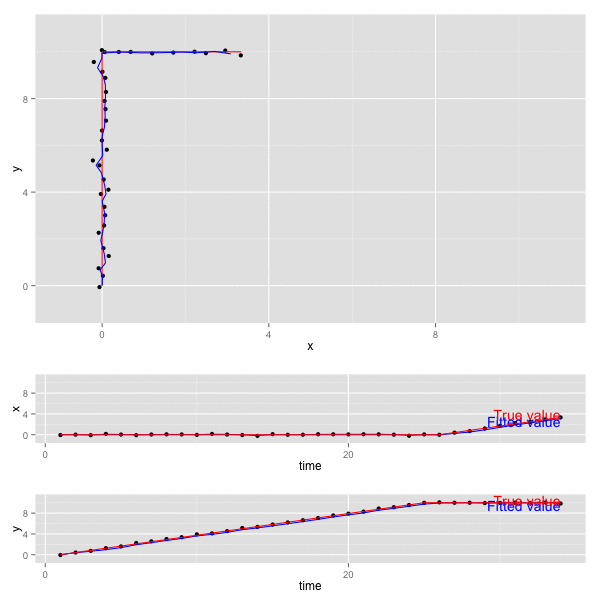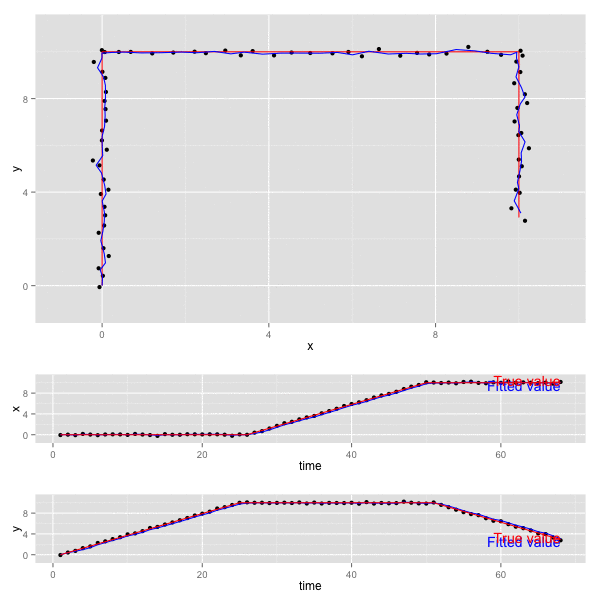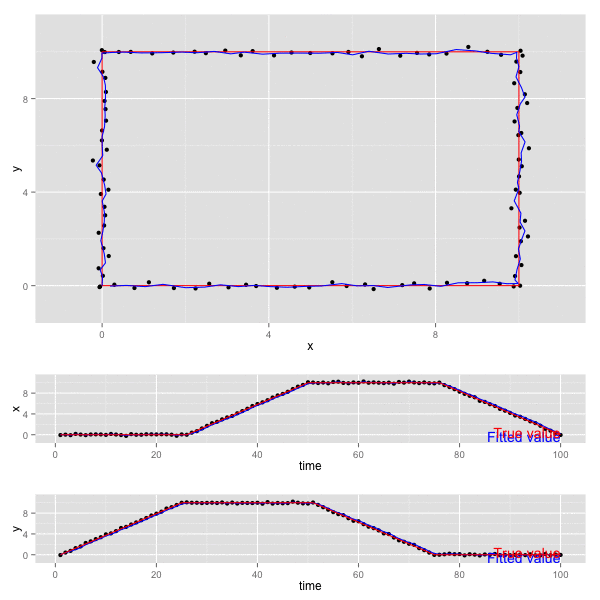
誤差の共分散 P , カルマンゲイン K は最終的に以下の値となっていた。観測誤差が独立なので、共分散成分はでてこない。
P[, , 100] # [,1] [,2] # [1,] 0.00618034 0.00000000 # [2,] 0.00000000 0.00618034 K[, , 100] # [,1] [,2] # [1,] 0.618034 0.000000 # [2,] 0.000000 0.618034
多次元になった場合も、それぞれの系列が独立で相互に干渉しなければ、各系列は 1次元の場合と同じ動きをする (各系列をわけて考えることができる)。
KalmanFilter の予測 F を入れる
これまでの例では と考えていたため、KalmanFilter の予測部分 (以下の式) で 予測値が更新されることはなかった (正確にいうと Q のみ計算されていた)。今度は F による予測ステップでの計算も含めて、各系列がどのように動くのか見てみる。
予測ステップ:
日本語版 wikipedia の "設定例" に記載されているトロッコの例をもとに実装する。
- 加速度 0.5 で等加速度運動するトロッコの位置 x, 速さ v を考える
- 観測誤差は それぞれの成分で独立
- 真値の予測
つまり、k+1時点のx =
, K+1時点のv =
と予測する
- システムの入力はなし (
)
- 真値の誤差は時間変化しない (
)
- 真値と観測値は同じ座標系 (
)
set.seed(1) # 観測系列のサンプルサイズ n <- 30 # 加速度 a <-rep(0.3, n) v <- cumsum(0.5 * a) x <- cumsum(v) # 真の値 actual <- data.frame(x = x, v = v) # 観測系列の次元 dim <- ncol(actual) # 観測される値 (誤差は独立正規分布とする) observed <- cbind(x + 0.5 * rnorm(n, sd = 0.05), v + rnorm(n, sd = 0.05)) # 予測結果保存用のmatrix xhat.m <- matrix(0, nrow = n, ncol = dim) # 補正結果保存用のmatrix xhat <- matrix(0, nrow = n, ncol = dim) # F FM <- matrix(c(1, 0, 1, 1), ncol = 2) # 結果保存用の array # P[, , k] K[, , k] に 時刻 k 時点の計算結果を保存 P <- array(c(0.0, 0.1, 0, 0.1), dim = c(dim, dim, n)) K <- array(0, dim = c(dim, dim, n)) # 誤差 Q <- diag(0.01, nrow = dim, ncol = dim) R <- diag(0.01, nrow = dim, ncol = dim) # 単位行列 I <- diag(1, ncol = dim, nrow = dim) for (k in seq(2, n)) { # predict xhat.m[k, ] <- FM %*% xhat[k-1, ] P.m <- FM %*% P[, , k-1] %*% t(FM) + Q # update S <- R + P.m K[, , k] <- P.m %*% solve(S) xhat[k, ] <- xhat.m[k, ] + K[, , k] %*% (observed[k, ] - xhat.m[k, ]) P[, , k] <- (I - K[, , k]) * P.m }
x, vをそれぞれ x軸, y軸方向にとり、以下のステップを順にプロットしてみた。
- k - 1 の観測値をもとにして 時刻 k の予測値を計算 (青破線) -> 予測ステップに該当
- 時刻 k の値を観測 (黒点)
- 時刻 k の値により、観測値を補正 (青実線) -> 更新ステップに該当
1で計算された予測値が、観測2をうけて補正され、3での更新後は真の値(赤線)に近づいた値となっていることがわかる。
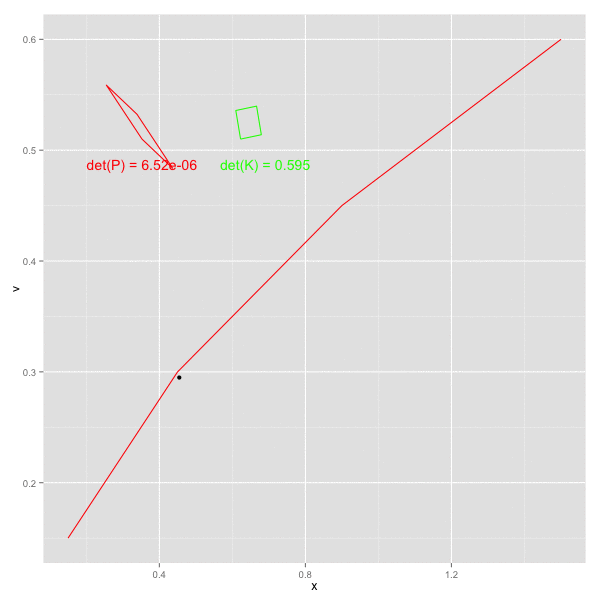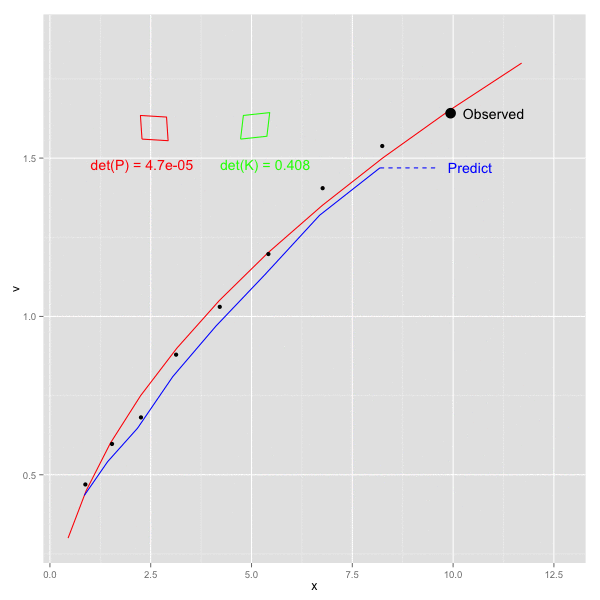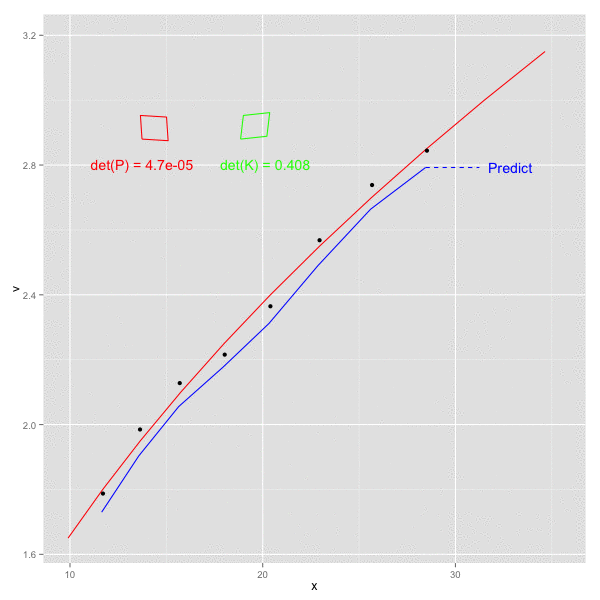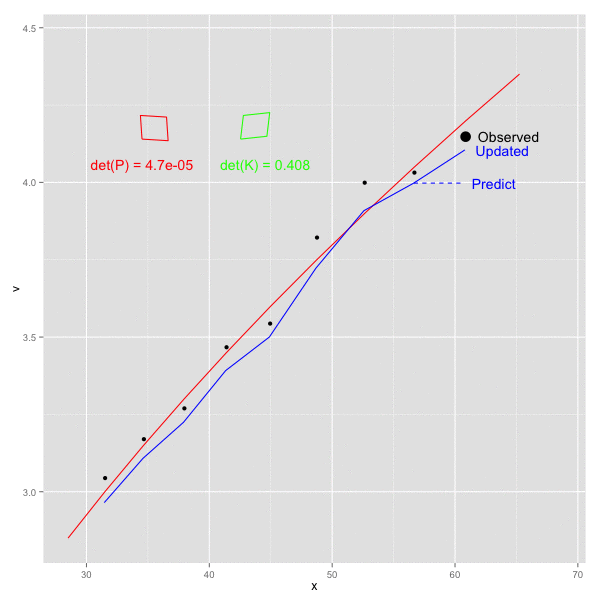
また、画面左側の赤四角、緑の四角はそれぞれ、誤差の共分散 P と カルマンゲイン K の各行列を四角形への回転として図示したもの。四角形は若干傾いており、P, K ともに共分散成分が含まれることを示す ( 実際にデータに乗せている誤差は独立だが、予測ステップで P が F によって更新される際に 共分散があらわれてくる)。
今回で KalmanFilter の予測、更新のステップはだいぶわかってきた (気がする)。が、まだ誤差についての理解が怪しいな、、。
ソースコード
KalmanFilter の動きを可視化する 一次元版
KalmanFilter をきちんと理解したいのだが いまいち 具体的な動作がわからない、、、ということで実装 & 可視化してみた。
KalmanFilter とは
誤差が乗っているであろう観測値の系列について、直前の観測と現在の観測を用いて 真の状態を推定する手法。例えば
- GPSで取得した位置情報から、正しい位置を推定する
- 取得可能な経済指標から 真の景気の状態を推定する
理論
はてなの TeX 記法で うまく数式がかけないところがあるので 英語版 wikipedia の数式を使う。KalmanFilter はある時点で観測を行うたびに 入力値を使って次の状態を予測するとともに、現時点の予測値を補正する処理を繰り返す。
予測:
更新:
k 時点での観測残差 (観測値と"真値の予測値から計算される観測値"の誤差)
k 時点での観測残差の共分散
カルマンゲイン (誤差
を最小化する行列)
k 時点の値を利用して更新した k 時点での"真値の予測値"
k 時点の値を利用して更新した k 時点での「真値と"真値の予測値"の誤差」の共分散
単純化
とりあえず以下のように単純化する。
- 行列だとわけわからなくなるので、1次元で考える
- 真値は時間によって変化しない (
, つまり
) とする
- システムへの入力はなし、つまり
とする
- 真値の誤差は時間変化しない (
) 、つまり定数とする
- 真値と観測値は同じ座標系 (この言葉がいいのかわかりませんが)、、、 つまり
とする
プログラム
元ネタは Python の Scipy cookbook。
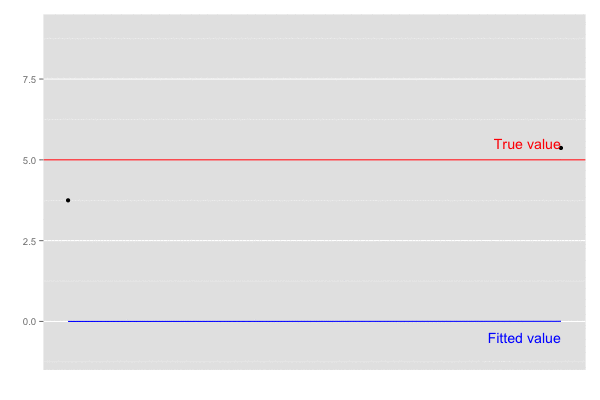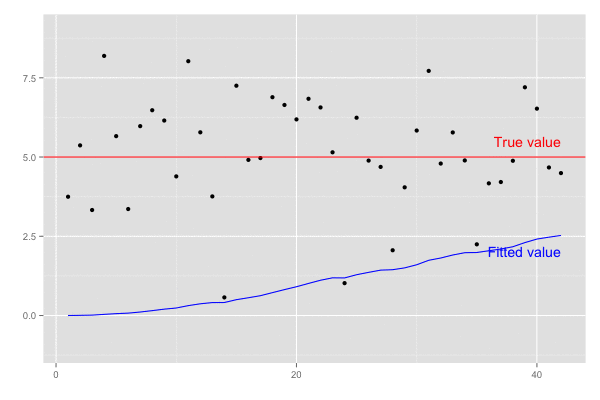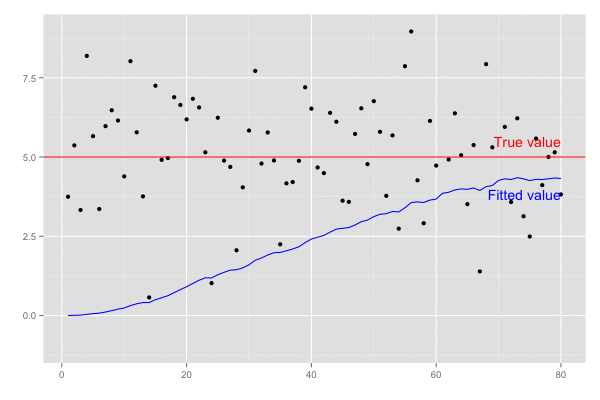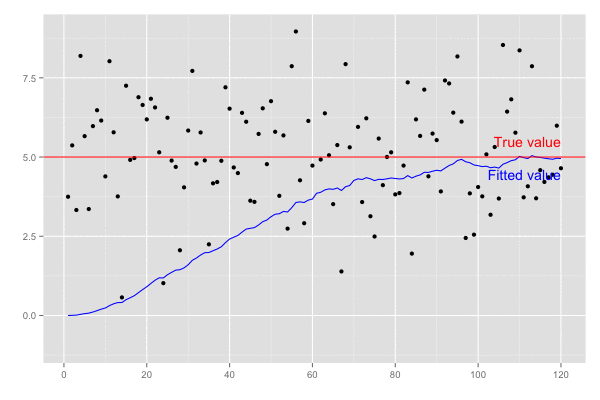
set.seed(1) # 観測系列のサンプルサイズ n <- 120 # 真の値 actual <- 5 # 観測される値 (誤差は標準偏差2の正規分布とする) observed <- rnorm(n, mean = actual, sd = 2) # 結果保存用のvector xhat <- rep(0, length.out = n) P <- rep(0, length.out = n) K <- rep(0, length.out = n) # 誤差 Q = 1e-5 R = 0.1 ** 2 for (k in seq(2, n)) { # predict xhat.m <- xhat[k-1] P.m <- P[k-1] + Q # update S <- R + P.m K[k] <- P.m / S xhat[k] <- xhat.m + K[k] * (observed[k] - xhat.m) P[k] <- (1 - K[k]) * P.m } # 予測値の推移 xhat # [1] 0.000000000 0.005361925 0.011992113 0.036413891 0.058736991 0.075059292 0.109930176 # [8] 0.153625564 0.200419955 0.236967065 0.311962013 0.369479486 0.408063382 0.410052292 # ... # [57] 3.584985506 3.565122418 3.641322662 3.673644005 3.856169061 3.887979061 3.962561981 # [64] 3.995372573 3.980879247 4.022972720 3.943466256 4.064106742 4.101759405 4.260930294 # ... # [106] 4.772249481 4.823831434 4.885835786 4.913247827 5.020455087 4.980316917 4.952240062 # [113] 5.042741558 5.000967888 4.988046417 4.963997727 4.945221799 4.929569956 4.962489912 # [120] 4.952628512
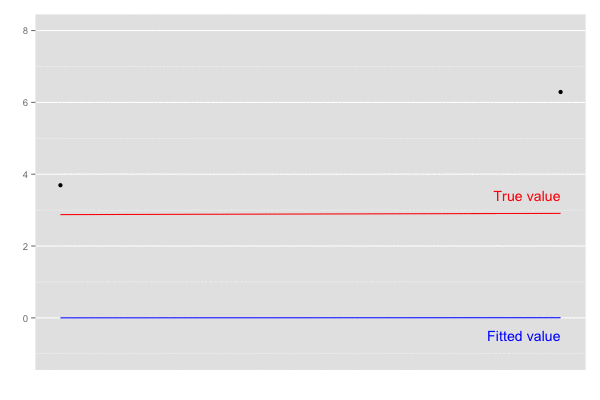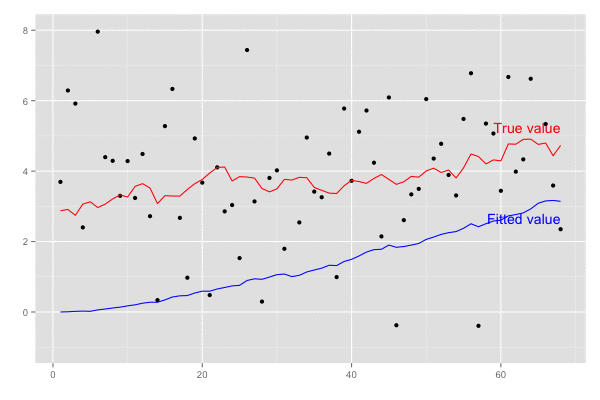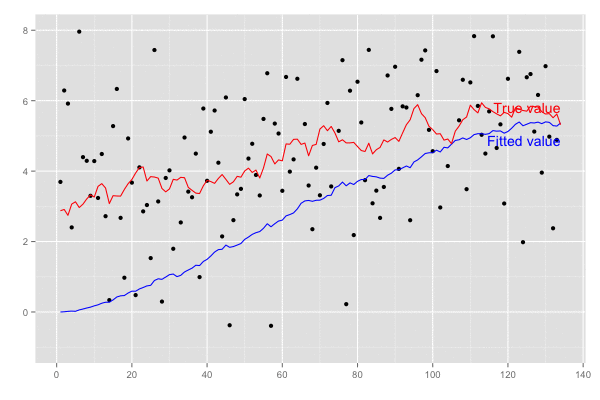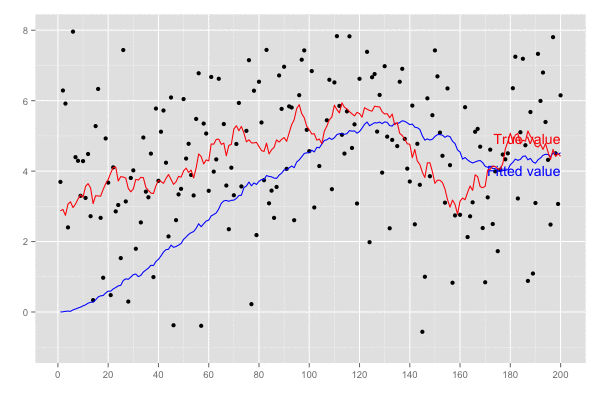
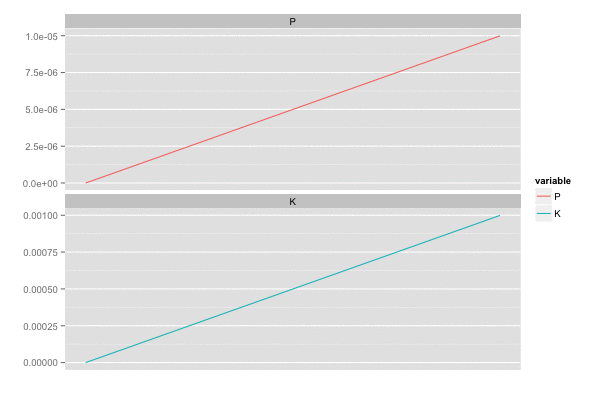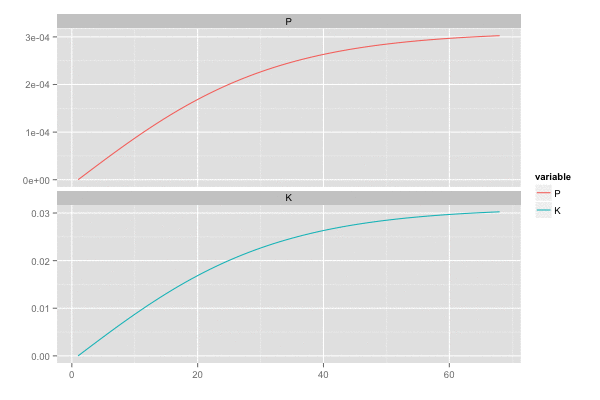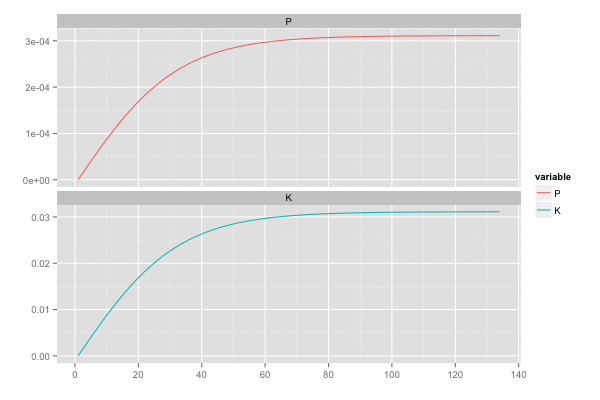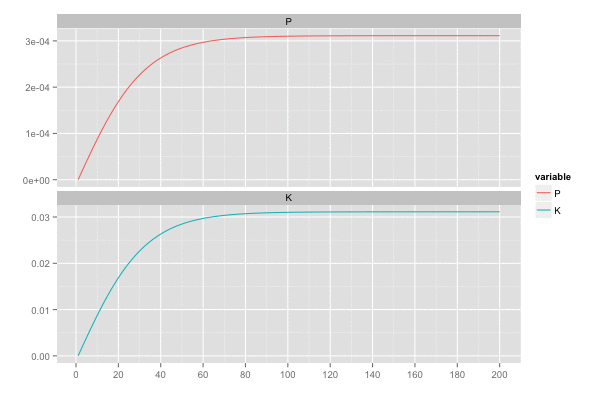
予測値 xhat は徐々に真値 5.0 に近づいていくことがわかる。
可視化
ggplot2 と animation パッケージで KalmanFilter の予測値が真値に近づいていく様子を可視化してみる。
library(animation) library(ggplot2) d <- data.frame(x = seq(1, n), actual = actual, observed = observed, fitted = xhat, P = P, K = K) saveGIF({ for (i in seq(2, n, by = 2)) { tmp <- head(d, i) p <- ggplot(tmp, aes(x = x)) + geom_point(aes(y = observed)) + geom_line(aes(y = fitted), colour = 'blue') + annotate(geom = 'text', x = i, y = tmp$fitted[i] - 0.5, label = 'Fitted value', colour = 'blue', hjust = 1) + geom_line(aes(y = actual), colour = 'red') + annotate(geom = 'text', x = i, y = tmp$actual[i] + 0.5, label = 'True value', colour = 'red', hjust = 1) + ylim(-1, 8) + scale_x_continuous(breaks = seq(0, n, by = 20)) + xlab('') + ylab('') print(p) } }, interval = 0.2, movie.name = "kalmanfilter01_01.gif", ani.width = 600, ani.height = 400)
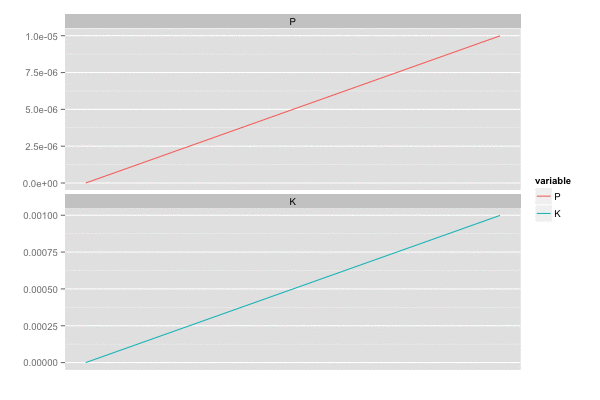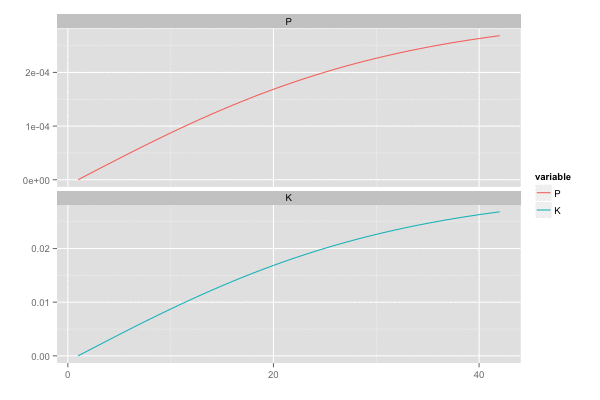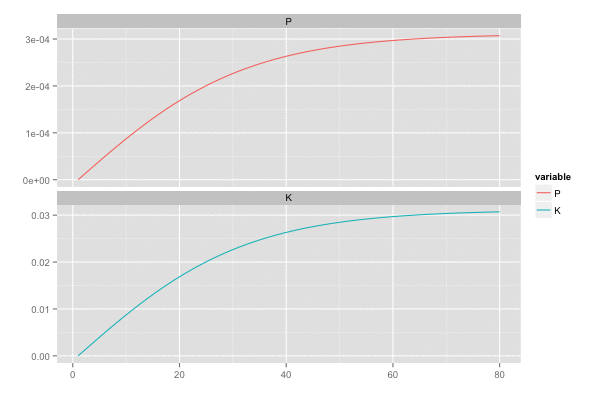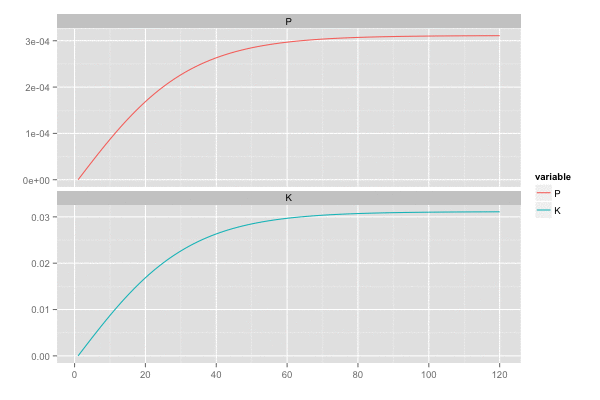
横軸を時間経過とし、観測値(黒点)が観測された際の予測値の推移(青線)を描画。
また、カルマンゲイン は 誤差の分散(一次元なので共分散はない)
を最小化しようとするので、
のとき
,
は同じ動きをする。
library(tidyr) saveGIF({ for (i in seq(2, n, by = 2)) { tmp <- head(d, i) tmp <- gather(tmp, 'variable', 'value', c(P, K)) p <- ggplot(tmp, aes(x = x, y = value, colour = variable)) + geom_line() + facet_wrap(~ variable, ncol = 1, scale = 'free_y') + scale_x_continuous(breaks = seq(0, n, by = 20)) + xlab('') + ylab('') print(p) } }, interval = 0.2, movie.name = "kalmanfilter02.gif", ani.width = 600, ani.height = 400)
真値が時間によって変化する場合
真値を動かしてみる。
- 行列だとわけわからなくなるので、1次元で考える
- 真値の誤差は時間変化しない (
) 、つまり定数とする
- 真値と観測値は同じ座標系、つまり
とする
※真値は動かすが、KalmanFilterで予測はしない ( ,
は変わらず)
set.seed(1) # 観測系列のサンプルサイズ n <- 200 # 真の値が時間変化する actual <- 3.0 + cumsum(rnorm(n, sd = 0.2)) # 観測される値 (誤差は標準偏差2の正規分布とする) observed <- actual + rnorm(n, mean = 0, sd = 2) # 以降同一
乗せている観測誤差の標準偏差は大きいので ある程度ずれてはいるが、トレンドは追えていそう。
次は
2次元で!
ソースコード
R ggplot2 で monthplot
Rで時系列データの周期特性をちゃっと確認したいとき、monthplotという関数を使うとデータを月次に分割してグラフ作成してくれる。
monthplot(AirPassengers)
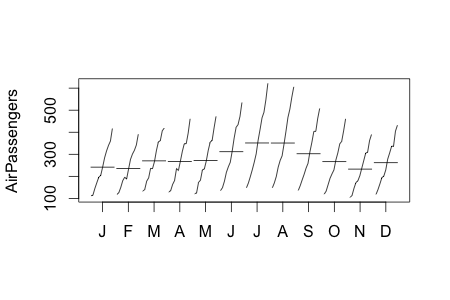
が、出力がちょっとアレなのと 月次以外の周期性をみたい場合があるので、 ggplot2 で作ってみた。
library(devtools) install_github('sinhrks/ggfortify') library(ggfortify) ggfreqplot(AirPassengers)
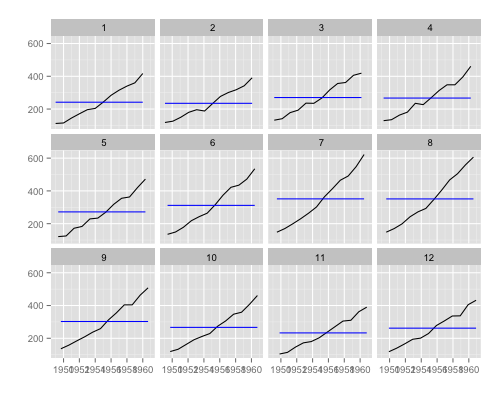
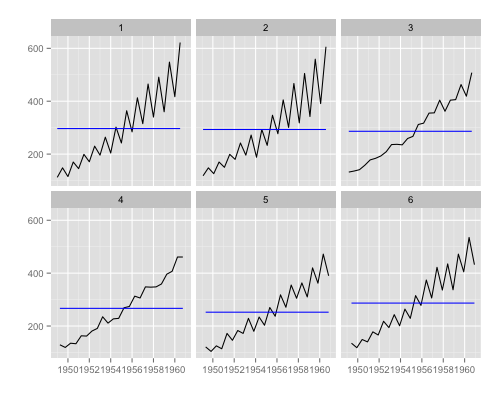
6ヶ月ごとの周期をみたいとき。
ggfreqplot(AirPassengers, freq = 6)
RPubs - Plotting Time Series Statistics with ggplot2 and ggfortify

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
R の {MSwM} パッケージでマルコフ転換モデルをためす
マルコフ転換モデルとは
数式を使わない説明。
サーモンとインターネット広告とマルコフ転換モデル|インターネット広告代理店で働くデータサイエンティストのブログ
マルコフ状態転換モデルのRパッケージ{MSwM}の使い方(異常値検出・ステータス変化検出などに有用) - 銀座で働くData Scientistのブログ
Exampleとしては vignettes がわかりやすい。
http://cran.r-project.org/web/packages/MSwM/vignettes/examples.pdf
まず
手元には下図のような時系列データがあるとする。経験的になんとなく 目的変数 y は説明変数 x に関係していそう、ということがわかっている。が、ぱっと見でも y のレベルが途中でシフトしており、常に一定の関係がなりたっているわけではなさそう (背後に複数の状態がありそう)。
こんなときにマルコフ転換モデルを使うと 背後にある状態変化を踏まえて モデルを作ってくれる。

上のサンプルデータは以下のようにして作った。時点に応じて x, y の関係が切り替わるデータとなっている。
※説明変数は目的変数とまったく関係のないものでもよいが、(一部の状態にでも)関係していそうなものを選んだほうがよい。説明変数が目的変数と関係ないものだと 切片の変化 = ベースラインの変化でしか状態転換を検出できないと思う。その場合マルコフ転換を使う必要がない。
x <- rpois(500, lambda = 10) # x はポアソン分布に従う y1 <- x * 4 + 20 # 状態1のときの x, yの関係 y2 <- x * 2 + 60 # 状態2のときの x, yの関係 # ノイズ付与 noise <- rnorm(1:500, mean = 10, sd = 5) y1 <- y1 + noise y2 <- y2 + noise # 適当なタイミングで状態転換 (1:200までは状態1, 201:400までは状態2, 以降は状態1) y <- c(y1[1:200], y2[201:400], y1[401:500]) # 実際に観測されたデータ observed <- data.frame(x = x, y = y)
x と y1, y2の関係を図示すると下図のようになる。x, yに二つの状態があることがわかっていれば、 x, yはこのように見える。
状態別に分離したデータ

が、観測者にとっては 状態がそもそもいくつあるのかすらわかっていない。そのため x, yは 状態の区別なくひとまとまりで見える。マルコフ転換モデルでは、観測データがいくつかの状態から発生していると捉えて、うまいこと上図のように分離してくれる。
観測されるデータ

マルコフ転換モデルの作成
{MSwM} でマルコフ転換モデルを作るのは非常にカンタンで、
- 目的変数/説明変数の関係を 線形回帰モデル, もしくは一般化線形モデルとして記述する
- 1で作ったモデル式とパラメータを
MSwM::msmFitに渡してマルコフ転換モデルをフィットさせる
だけ。
モデル式作成
ここでは y と x は線形関係にあるものとして、lm 関数でモデル式を作る。この状態 (全体をひとつの状態として線形回帰した場合)で summary 表示して Coefficients をみると、ちょうど y1, y2の間くらいに線を引いていることがわかる。残差が大きいので 調整済みR二乗値も低い。
library(MSwM) m.lm <- lm(y ~ x, data = observed) summary(m.lm) # # Call: # lm(formula = y ~ x, data = observed) # # Residuals: # Min 1Q Median 3Q Max # -24.303 -9.354 -1.914 9.617 29.224 # # Coefficients: # Estimate Std. Error t value Pr(>|t|) # (Intercept) 45.7468 1.7202 26.59 <2e-16 *** # x 3.2262 0.1636 19.71 <2e-16 *** # --- # Signif. codes: # 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 # # Residual standard error: 11.51 on 498 degrees of freedom # Multiple R-squared: 0.4383, Adjusted R-squared: 0.4372 # F-statistic: 388.7 on 1 and 498 DF, p-value: < 2.2e-16
モデルのフィット
上で作ったモデルを msmFit に渡せばよい。パラメータの意味は、
k: マルコフ転換モデルの状態数。 ここでは 背後に2つの状態があるものとして指定。sw: 各パラメータが状態転換時に変化するかどうかを Logical で指定p: ARモデルの係数family: (GLMの場合) 確率分布の family
m.mswm <- msmFit(m.lm, k = 2, sw = c(TRUE, TRUE, TRUE, TRUE), p = 1) summary(m.mswm) # Markov Switching Model # # Call: msmFit(object = m.lm, k = 2, sw = c(TRUE, TRUE, TRUE, TRUE), # p = 1) # # AIC BIC logLik # 3038.846 3101.397 -1513.423 # # Coefficients: # # Regime 1 # --------- # Estimate Std. Error t value Pr(>|t|) # (Intercept)(S) 69.3263 4.0606 17.0729 <2e-16 *** # x(S) 2.1795 0.1187 18.3614 <2e-16 *** # y_1(S) -0.0103 0.0429 -0.2401 0.8103 # --- # Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 # # Residual standard error: 4.99756 # Multiple R-squared: 0.6288 # # Standardized Residuals: # Min Q1 Med Q3 Max # -1.431396e+01 -2.056292e-02 -1.536781e-03 -1.098923e-05 1.584478e+01 # # Regime 2 # --------- # Estimate Std. Error t value Pr(>|t|) # (Intercept)(S) 30.2820 1.7687 17.1210 <2e-16 *** # x(S) 3.9964 0.0913 43.7722 <2e-16 *** # y_1(S) -0.0045 0.0203 -0.2217 0.8245 # --- # Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 # # Residual standard error: 4.836684 # Multiple R-squared: 0.8663 # # Standardized Residuals: # Min Q1 Med Q3 Max # -13.202056966 -0.771854514 0.002211602 1.162769110 12.417873232 # # Transition probabilities: # Regime 1 Regime 2 # Regime 1 0.994973376 0.003347279 # Regime 2 0.005026624 0.996652721
出力中の Regime 1, Regime 2が 背後にある(と考えられる)二つの状態をあらわす。y の真の値と比較すると、出力中の Regime 1が y2 <- x * 2 + 60 に対応しており、
# Regime 1 # --------- # Estimate Std. Error t value Pr(>|t|) # (Intercept)(S) 69.3263 4.0606 17.0729 <2e-16 *** # x(S) 2.1795 0.1187 18.3614 <2e-16 *** # y_1(S) -0.0103 0.0429 -0.2401 0.8103
Regime 2が y1 <- x * 4 + 20 と対応していることがわかる。
切片のずれが少し気になるがノイズのせいかな、、?調整済みR二乗値から全体として改善しているとは言える。
# Regime 2 # --------- # Estimate Std. Error t value Pr(>|t|) # (Intercept)(S) 30.2820 1.7687 17.1210 <2e-16 *** # x(S) 3.9964 0.0913 43.7722 <2e-16 *** # y_1(S) -0.0045 0.0203 -0.2217 0.8245
モデルのプロット
状態 (Regime) ごとに 目的変数 + 指定した説明変数と、その状態にある確率を陰影でプロット
plotReg(m.mswm, c('x'), regime = 1)

各時点がそれぞれどちらの状態 (Regime)にあるかを確率でプロット
plotProb(m.mswm, which = 1)
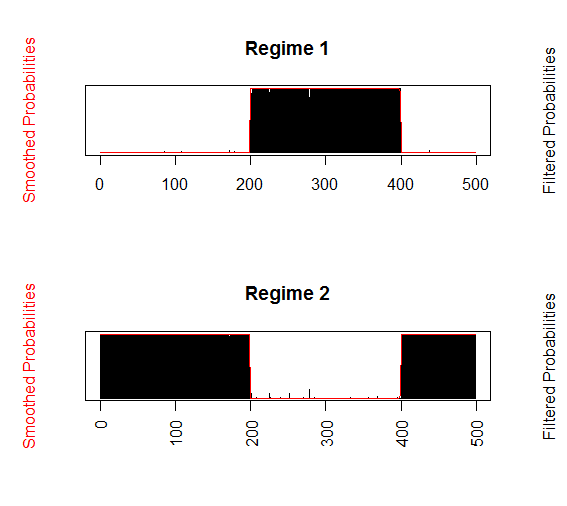
各時点の状態と変化点を取得
ある時点でどちらの状態 (Regime) にいるかを知りたい場合は、その時点で一番 発生している(していた)確率の高い Regimeを拾ってくればよい。
probable <- apply(m.mswm@Fit@smoProb[-1, ], 1, which.max) > probable [1] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 [30] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 ...
異常値 / 変化点は Regime が転換した時点になるので、上で取得したベクトルの値が変化した箇所が TRUE になるようにすると、
c(FALSE, diff(probable) != 0) [1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE [11] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE ... [181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE [191] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE [201] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE ... [381] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE [391] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE [401] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE ... [491] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
ということで 最初のデータ作成時に指定した変化点 (201, 401番目)に近いところが検知できているのがわかる。
ggplot2でよく使うパッケージを autoplot する
パッケージを書いた。
つかいかた
- RPubs - Plotting Time Series with ggplot2 and ggfortify
- RPubs - Plotting Time Series Statistics with ggplot2 and ggfortify
- RPubs - Plotting PCA/clustering results using ggplot2 and ggfortify
- RPubs - Plotting Survival Curves using ggplot2 and ggfortify
- RPubs - Plotting Probability Distributions with ggplot2 and ggfortify

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
R prcomp での主成分分析結果から元データを復元する
普通はこんなことやる必要ないですが、、、主成分分析 prcomp 関数の結果のみを引数にして、元データ込みの処理を行う関数がどうしても書きたかったので。
# 元データ head(iris) # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 1 5.1 3.5 1.4 0.2 setosa # 2 4.9 3.0 1.4 0.2 setosa # 3 4.7 3.2 1.3 0.2 setosa # 4 4.6 3.1 1.5 0.2 setosa # 5 5.0 3.6 1.4 0.2 setosa # 6 5.4 3.9 1.7 0.4 setosa # 数値列のみにフィルタ df <- iris[c(1, 2, 3, 4)]
このデータに対して主成分分析を行い、得られた結果から元データを復元したい。
主成分分析では元データを分散が最大になる方向に回転させたものが主成分得点、回転を定義する行列が主成分(固有ベクトル)になっているため、主成分得点を主成分(固有ベクトル)と逆に回転すれば元の方向に戻る。また、prcomp デフォルトでは center = TRUE が指定されており、元データの平均値を 0 にする標準化が行われているので、こちらも逆の処理をすれば元データに戻せる。
# 主成分分析実行 pca.result <- prcomp(df) str(pca.result) # List of 5 # $ sdev : num [1:4] 2.056 0.493 0.28 0.154 # $ rotation: num [1:4, 1:4] 0.3614 -0.0845 0.8567 0.3583 -0.6566 ... # ..- attr(*, "dimnames")=List of 2 # .. ..$ : chr [1:4] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" # .. ..$ : chr [1:4] "PC1" "PC2" "PC3" "PC4" # $ center : Named num [1:4] 5.84 3.06 3.76 1.2 # ..- attr(*, "names")= chr [1:4] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" # $ scale : logi FALSE # $ x : num [1:150, 1:4] -2.68 -2.71 -2.89 -2.75 -2.73 ... # ..- attr(*, "dimnames")=List of 2 # .. ..$ : NULL # .. ..$ : chr [1:4] "PC1" "PC2" "PC3" "PC4" # - attr(*, "class")= chr "prcomp" # 主成分と逆方向に回転 original <- pca.result$x %*% t(pca.result$rotation) # 平均値の標準化を元に戻す original <- scale(original, center = -pca.result$center, scale = FALSE) head(original) # Sepal.Length Sepal.Width Petal.Length Petal.Width # [1,] 5.1 3.5 1.4 0.2 # [2,] 4.9 3.0 1.4 0.2 # [3,] 4.7 3.2 1.3 0.2 # [4,] 4.6 3.1 1.5 0.2 # [5,] 5.0 3.6 1.4 0.2 # [6,] 5.4 3.9 1.7 0.4
prcomp(scale=TRUE) で標準偏差も標準化して主成分分析した場合は、平均値を戻す前に標準偏差を戻す。
# 主成分分析実行 pca.result <- prcomp(df, scale = TRUE) # 主成分と逆方向に回転 original <- pca.result$x %*% t(pca.result$rotation) # 標準偏差の標準化を元に戻す original <- scale(original, center = FALSE, scale = 1 / pca.result$scale) # 平均値の標準化を元に戻す original <- scale(original, center = -pca.result$center, scale = FALSE) head(original) # Sepal.Length Sepal.Width Petal.Length Petal.Width # [1,] 5.1 3.5 1.4 0.2 # [2,] 4.9 3.0 1.4 0.2 # [3,] 4.7 3.2 1.3 0.2 # [4,] 4.6 3.1 1.5 0.2 # [5,] 5.0 3.6 1.4 0.2 # [6,] 5.4 3.9 1.7 0.4
Python rpy2 で pandas の DataFrame を R の data.frame に変換する
pandas の DataFrame を R へ渡す/また R から Python へデータを戻す方法について、本家のドキュメント が書きかけなのでよくわからない。ということで 以前 下の文書を書いたので訳してみる。
DOC: Complete R interface section by sinhrks · Pull Request #7309 · pydata/pandas · GitHub
rpy2 を使うと pandas (Python) <-> R 間のデータの相互変換を以下 2通りの方法で行うことができる。
何をどちらに読み込ませるかという話なので、1, 2を組み合わせて処理することもできる。
共通
rpy2 のインストール
pip install rpy2
準備
import numpy as np import pandas as pd # 表示する行数を指定 pd.options.display.max_rows = 5
R のデータセットを Pandas に読み込む
pandas.rpy.common.load_data で、R のデータセットが pandas.DataFrame に変換されて読み込まれる。
import pandas.rpy.common as com infert = com.load_data('infert') type(infert) # pandas.core.frame.DataFrame infert.head() # education age parity induced case spontaneous stratum pooled.stratum # 1 0-5yrs 26 6 1 1 2 1 3 # 2 0-5yrs 42 1 1 1 0 2 1 # 3 0-5yrs 39 6 2 1 0 3 4 # 4 0-5yrs 34 4 2 1 0 4 2 # 5 6-11yrs 35 3 1 1 1 5 32
pandas.DataFrame を R に渡せる形式 (rpy2) に変換する
pandas.DataFrame から R に渡せる形式 (rpy2.robjects.DataFrame) への変換は com.convert_to_r_dataframe で行う。
# サンプルの DataFrame 作成 df = pd.DataFrame({'A': [1, 2, 3], 'B': [4, 5, 6], 'C':[7,8,9]}, index=["one", "two", "three"]) df # A B C # one 1 4 7 # two 2 5 8 # three 3 6 9 r_dataframe = com.convert_to_r_dataframe(df) type(r_dataframe) # rpy2.robjects.vectors.DataFrame # ipython でやる場合、print をかまさないと出力がうっとおしい print(r_dataframe) # A B C # one 1 4 7 # two 2 5 8 # three 3 6 9 # rpy2.objects.DataFrame に変換後も属性名の確認/操作はできる ( pandas でやっておけば必要ないが) print(r_dataframe.rownames) # [1] "one" "two" "three" In [20]: print(r_dataframe.colnames) [1] "A" "B" "C"
補足 R へ matrix で渡したい場合は com.convert_to_r_matrix。
r_matrix = com.convert_to_r_matrix(df) type(r_matrix) # rpy2.robjects.vectors.Matrix print(r_matrix) # A B C # one 1 4 7 # two 2 5 8 # three 3 6 9
rpy2 形式のデータ を pandas.DataFrame に変換する
上の逆変換は com.convert_robj。rpy2 オブジェクト (rpy2.robjects.DataFrame) を pandas.DataFrame に戻す。
com.convert_robj(r_dataframe) # A B C # one 1 4 7 # two 2 5 8 # three 3 6 9 com.convert_robj(r_matrix) # A B C # one 1 4 7 # two 2 5 8 # three 3 6 9
1. R の関数を Python に loadし、Python の名前空間上で操作する
rpy2.robjects.r.__getitem__ で、R の名前空間のオブジェクトを Python の名前空間へ load できる。ここでは R の sum 関数をPython 上に読み出して使う。読み込んだ関数へ渡せるのは rpy2 のオブジェクトのみ。
# 処理するデータ print(r_dataframe) # A B C # one 1 4 7 # two 2 5 8 # three 3 6 9 import rpy2.robjects as robjects # R 上の sum 関数を rsum として Python の名前空間に load rsum = robjects.r['sum'] type(rsum) # rpy2.robjects.functions.SignatureTranslatedFunction # r_dataframe に対して関数適用 (Python の関数として使える) rsum_result = rsum(r_dataframe) # R の関数なので、結果は vector になる type(rsum_result) # rpy2.robjects.vectors.IntVector # 要素を取り出す場合はスライス rsum_result[0] # 45
2. Python のデータを R に渡し、 R の名前空間上で操作する
rpy2 のオブジェクトを R の名前空間へ渡す場合は robjects.r.assign。
R で 実行する処理(コマンド)を R に渡す (Rに何か処理をさせる) 場合は robjects.r。
# Python 上の r_dataframe が R 上で rdf という名前で転送される # セミコロンは ipython での出力省略のため robjects.r.assign('rdf', r_dataframe); # R 上で str(rdf) コマンドを実行 robjects.r('str(rdf)'); # 'data.frame': 3 obs. of 3 variables: # $ A:Class 'AsIs' int [1:3] 1 2 3 # $ B:Class 'AsIs' int [1:3] 4 5 6 # $ C:Class 'AsIs' int [1:3] 7 8 9
処理サンプル
これまでの処理の組み合わせで以下のようなことができる。
線形回帰
# データの準備 iris = com.load_data('iris') # setosa のデータをフィルタ setosa = iris[iris['Species'] == 'setosa'] setosa.head() # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 1 5.1 3.5 1.4 0.2 setosa # 2 4.9 3.0 1.4 0.2 setosa # 3 4.7 3.2 1.3 0.2 setosa # 4 4.6 3.1 1.5 0.2 setosa # 5 5.0 3.6 1.4 0.2 setosa # (ほか、R でやるには面倒な処理があれば pandas で実行... ) # rpy2 オブジェクトに変換 r_setosa = com.convert_to_r_dataframe(setosa) # R の名前空間に送る robjects.r.assign('setosa', r_setosa); # R の lm 関数を実行し、結果の summary を表示する robjects.r('result <- lm(Sepal.Length~Sepal.Width, data=setosa)'); print(robjects.r('summary(result)')) # Call: # lm(formula = Sepal.Length ~ Sepal.Width, data = setosa) # # Residuals: # Min 1Q Median 3Q Max # -0.52476 -0.16286 0.02166 0.13833 0.44428 # # Coefficients: # Estimate Std. Error t value Pr(>|t|) # (Intercept) 2.6390 0.3100 8.513 3.74e-11 *** # Sepal.Width 0.6905 0.0899 7.681 6.71e-10 *** # --- # Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 # # Residual standard error: 0.2385 on 48 degrees of freedom # Multiple R-squared: 0.5514, Adjusted R-squared: 0.542 # F-statistic: 58.99 on 1 and 48 DF, p-value: 6.71e-10 # R 上の result オブジェクトを Python の名前空間に戻す result = robjects.r['result'] print(result.names) # [1] "coefficients" "residuals" "effects" "rank" # [5] "fitted.values" "assign" "qr" "df.residual" # [9] "xlevels" "call" "terms" "model" # 結果は名前付きリストになっている。各要素には .rx でアクセスできる print(result.rx('coefficients')) # $coefficients # (Intercept) Sepal.Width # 2.6390012 0.6904897 # 回帰式の切片, 係数を取得 intercept, coef1 = result.rx('coefficients')[0] intercept # 2.6390012498579694 coef1 # 0.6904897170776046
時系列処理
時系列の場合に気をつけるのは、
convert_to_r_dataframeはSeriesに対応していないので、rpy2で直接 ベクトル (ここではrpy2.FloatVector) を作って渡す。詳細は rpy2 documentation: Vectors and arrays 。tsオブジェクトへの変換は R で明示的に行う- R から
pandas.DataFrameへ結果を戻した際に、日時の index を再度 付与する
# 2013年1月〜 月次 4年分のランダムデータを作成 idx = pd.date_range(start='2013-01-01', freq='M', periods=48) vts = pd.Series(np.random.randn(48), index=idx).cumsum() vts # 2013-01-31 0.801791 # ... # 2016-12-31 -3.391142 # Freq: M, Length: 48 # R へ渡す rpy2 オブジェクトを準備 r_values = robjects.FloatVector(vts.values) # R の名前空間へ転送 robjects.r.assign('values', r_values); # R の ts 関数で timeseries に変換 robjects.r('vts <- ts(values, start=c(2013, 1, 1), frequency=12)'); print(robjects.r['vts']) # Jan Feb Mar Apr May Jun # 2013 0.80179068 -0.04157987 -0.15779190 -0.02982779 -2.03214239 -0.09868078 # 2014 5.97378179 6.27023875 4.85958760 6.50728371 5.14595583 5.29780411 # 2015 1.04548632 0.60093762 0.13941486 0.56116450 -0.20040731 1.19696178 # 2016 -0.09101317 -0.79038658 -0.13305769 0.61016756 -0.13059757 -1.28190161 # Jul Aug Sep Oct Nov Dec # 2013 2.84555901 3.96259305 4.45565104 2.86998914 4.52347928 4.38237841 # 2014 5.16001952 3.44611678 3.49705824 2.37352719 0.75428874 1.62569642 # 2015 -0.03488274 0.13323226 -0.78262492 -0.75325348 -0.65414439 0.40700944 # 2016 -2.31702656 -1.78120320 -1.92904062 -0.83488094 -2.31609640 -3.39114197 # R の stl 関数を実行し、 時系列をトレンド/季節性/残差に分解 robjects.r('result <- stl(vts, s.window=12)'); # 結果を Python の名前空間に戻す result = robjects.r['result'] print(result.names) # [1] "time.series" "weights" "call" "win" "deg" # [6] "jump" "inner" "outer" # 結果の時系列を取得し、pandas.DataFrame へ変換 (index は数値型になってしまう) result_ts = result.rx('time.series')[0] converted = com.convert_robj(result_ts) converted.head() # seasonal trend remainder # 2013.000000 0.716947 -1.123112 1.207956 # 2013.083333 0.264772 -0.603006 0.296655 # 2013.166667 -0.165811 -0.082900 0.090919 # 2013.250000 0.528043 0.437206 -0.995077 # 2013.333333 -0.721440 0.938796 -2.249498 # index を再設定 converted.index = idx converted.head() # seasonal trend remainder # 2013-01-31 0.716947 -1.123112 1.207956 # 2013-02-28 0.264772 -0.603006 0.296655 # 2013-03-31 -0.165811 -0.082900 0.090919 # 2013-04-30 0.528043 0.437206 -0.995077 # 2013-05-31 -0.721440 0.938796 -2.249498
結果をプロットしてみる。
import matplotlib.pyplot as plt fig, axes = plt.subplots(4, 1) axes[0].set_ylabel('Original'); ax = vts.plot(ax=axes[0]); axes[1].set_ylabel('Trend'); ax = converted['trend'].plot(ax=axes[1]); axes[2].set_ylabel('Seasonal'); ax = converted['seasonal'].plot(ax=axes[2]); axes[3].set_ylabel('Residuals'); converted['remainder'].plot(ax=axes[3]) plt.show()
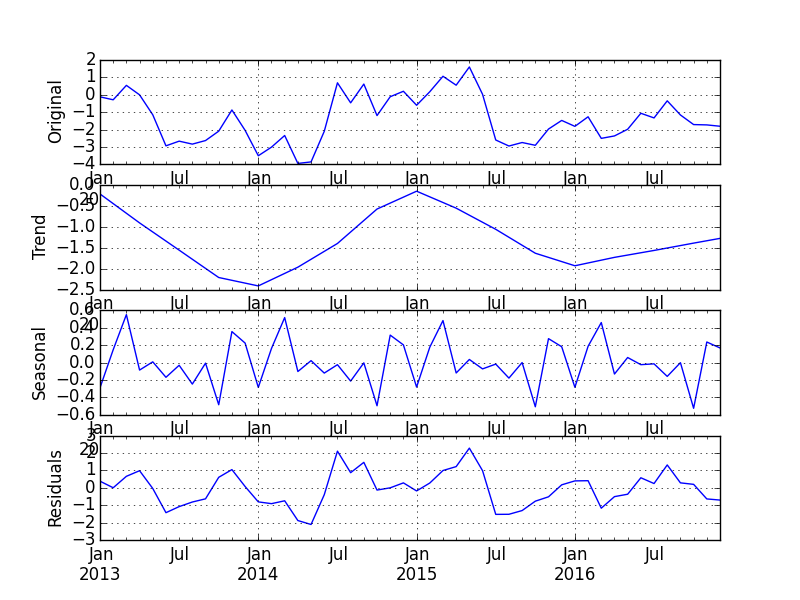
補足
とはいえ、いちいち pandas -> rpy2 形式へ変換するのは面倒なので、robjects.r で直接 pandas.DataFrame を受け渡しできるとうれしい。やり方は、
ENH: automatic rpy2 instance conversion by sinhrks · Pull Request #7385 · pydata/pandas · GitHub

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る
R dplyr, tidyr でのグルーピング/集約/変換処理まとめ
これの続き。よく使う集約/変換処理もまとめておく。
準備
library(dplyr) library(tidyr) (df <- dplyr::tbl_df(iris)) # Source: local data frame [150 x 5] # # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 1 5.1 3.5 1.4 0.2 setosa # 2 4.9 3.0 1.4 0.2 setosa # 3 4.7 3.2 1.3 0.2 setosa # .. ... ... ... ... ...
グルーピング/集約
ある列の値ごとに集計
Species 列ごとに Sepal.Length 列の合計を算出する場合、
df %>% dplyr::group_by(Species) %>% dplyr::summarise(Sepal.Length.Total = sum(Sepal.Length)) # Source: local data frame [3 x 2] # # Species Sepal.Length.Total # 1 setosa 250.3 # 2 versicolor 296.8 # 3 virginica 329.4 # Standard Evaluation df %>% dplyr::group_by_('Species') %>% dplyr::summarise_('Sepal.Length.Total' = 'sum(Sepal.Length)') # 略
同一の集計を 複数列に対して行う場合は dplyr::summarise_eachが便利。全列の合計を取得するなら、
df %>% dplyr::group_by(Species) %>% dplyr::summarise_each(dplyr::funs(sum)) # Source: local data frame [3 x 5] # # Species Sepal.Length Sepal.Width Petal.Length Petal.Width # 1 setosa 250.3 171.4 73.1 12.3 # 2 versicolor 296.8 138.5 213.0 66.3 # 3 virginica 329.4 148.7 277.6 101.3
vars 引数で集計対象列を指定することもできる。通常版 (Non-standard evaluation/NSE) と Standard evaluation で列名のつき方が違う、、、。
df %>% dplyr::group_by(Species) %>% dplyr::summarise_each(dplyr::funs(sum), vars = c(Petal.Width = Petal.Width, Petal.Length = Petal.Length)) # Source: local data frame [3 x 3] # # Species vars.Petal.Width vars.Petal.Length # 1 setosa 12.3 73.1 # 2 versicolor 66.3 213.0 # 3 virginica 101.3 277.6 # Standard evaluation df %>% dplyr::group_by_('Species') %>% dplyr::summarise_each_(dplyr::funs(sum), vars = c('Petal.Width', 'Petal.Length')) # Source: local data frame [3 x 5] # # Species Petal.Width_sum Petal.Length_sum Petal.Width_mean Petal.Length_mean # 1 setosa 12.3 73.1 0.246 1.462 # 2 versicolor 66.3 213.0 1.326 4.260 # 3 virginica 101.3 277.6 2.026 5.552
関数名を文字列渡しする場合は dplyr::funs_。
df %>% dplyr::group_by(Species) %>% dplyr::summarise_each(dplyr::funs_('sum'), vars=c(Petal.Width = Petal.Width, Petal.Length = Petal.Length)) # 略 # Standard evaluation df %>% dplyr::group_by_('Species') %>% dplyr::summarise_each_(dplyr::funs_('sum'), vars=c('Petal.Width', 'Petal.Length')) # 略
また、集約関数は複数渡すこともできる。
df %>% dplyr::group_by(Species) %>% dplyr::summarise_each(funs = c(dplyr::funs(sum), dplyr::funs(mean)), vars=c(Petal.Width = Petal.Width, Petal.Length = Petal.Length)) # Source: local data frame [3 x 5] # # Species vars.Petal.Width_sum vars.Petal.Length_sum vars.Petal.Width_mean vars.Petal.Length_mean # 1 setosa 12.3 73.1 0.246 1.462 # 2 versicolor 66.3 213.0 1.326 4.260 # 3 virginica 101.3 277.6 2.026 5.552 # Standard evaluation df %>% dplyr::group_by_('Species') %>% dplyr::summarise_each_(funs = c(dplyr::funs(sum), dplyr::funs(mean)), vars=c('Petal.Width', 'Petal.Length')) # Source: local data frame [3 x 5] # # Species Petal.Width_sum Petal.Length_sum Petal.Width_mean Petal.Length_mean # 1 setosa 12.3 73.1 0.246 1.462 # 2 versicolor 66.3 213.0 1.326 4.260 # 3 virginica 101.3 277.6 2.026 5.552
NSEの dplyr::groupby に文字列を渡した場合、特にエラーが出ず全集約されるので注意。
# NG! df %>% dplyr::group_by('Species') %>% dplyr::summarise_each_(funs = c(dplyr::funs(sum), dplyr::funs(mean)), vars=c('Petal.Width', 'Petal.Length')) # Source: local data frame [1 x 5] # # "Species" Petal.Width_sum Petal.Length_sum Petal.Width_mean Petal.Length_mean # 1 Species 179.9 563.7 1.199333 3.758
行持ち / 列持ち変換
複数列持ちの値を行持ちに展開 (unpivot / melt)
複数列で持っている値を行持ちに展開する処理は、reshape2::melt から名前が変わって tidyr::gather になった。この処理の名前として一般的なのは unpivot ではないのか、、。
tidyr でも Standard evaluationが使える。
df %>% tidyr::gather(variable, value, c(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width)) # Source: local data frame [600 x 3] # # Species variable value # 1 setosa Sepal.Length 5.1 # 2 setosa Sepal.Length 4.9 # 3 setosa Sepal.Length 4.7 # .. ... ... ... # Standard evaluation df %>% tidyr::gather_('variable', 'value', c('Sepal.Length', 'Sepal.Width', 'Petal.Length', 'Petal.Width')) # 略
複数行持ちの値を列持ちに変換 (pivot)
tidyr::spread。spreadでは集計処理は入らない (行列の入替のみ) ので、まず 行/列として展開する 2列がユニークとなるサンプルデータを用意する。
# spreadへの入力データ。Species (列にする値) と variable (行にする値) の組がユニークでないとダメ。 (unpivot <- df %>% tidyr::gather(variable, value, c(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width)) %>% dplyr::group_by(Species, variable) %>% dplyr::summarise(Total = sum(value))) # Source: local data frame [12 x 3] # Groups: Species # # Species variable Total # 1 setosa Sepal.Length 250.3 # 2 setosa Sepal.Width 171.4 # 3 setosa Petal.Length 73.1 # 4 setosa Petal.Width 12.3 # 5 versicolor Sepal.Length 296.8 # 6 versicolor Sepal.Width 138.5 # 7 versicolor Petal.Length 213.0 # 8 versicolor Petal.Width 66.3 # 9 virginica Sepal.Length 329.4 # 10 virginica Sepal.Width 148.7 # 11 virginica Petal.Length 277.6 # 12 virginica Petal.Width 101.3 unpivot %>% data.frame %>% tidyr::spread(Species, Total) # variable setosa versicolor virginica # 1 Sepal.Length 250.3 296.8 329.4 # 2 Sepal.Width 171.4 138.5 148.7 # 3 Petal.Length 73.1 213.0 277.6 # 4 Petal.Width 12.3 66.3 101.3 # Standard evaluation unpivot %>% data.frame %>% tidyr::spread_('Species', 'Total') # 略
(たぶんバグだと思うが) grouped_df を spread に渡すと以下の通りエラーになるので、上では spread の前に data.frame への変換を挟んでいる。
→ 10/16 追記: バグでした。dplyr 3.1 で修正予定。
class(unpivot) # [1] "grouped_df" "tbl_df" "tbl" "data.frame" # NG! unpivot %>% tidyr::spread(Species, Total) # エラー: index out of bounds
列の分割 / 結合
列の値を複数列に分割
上で作ったデータから variable 列の値をドットで区切って二列に分けたい、なんてときには tidyr::separate。NSE の場合も into に渡す値は文字列 vector でないとダメっぽい (新しい列名を作る処理になるからかなあ、 dplyr::rename では作成する列名もシンボルで渡せるのに)。
df %>% tidyr::gather(variable, value, c(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width)) %>% tidyr::separate(variable, into = c('Parts', 'Scale'), sep='\\.') # Source: local data frame [600 x 4] # # Species Parts Scale value # 1 setosa Sepal Length 5.1 # 2 setosa Sepal Length 4.9 # 3 setosa Sepal Length 4.7 # .. ... ... ... ... # Standard evaluation df %>% tidyr::gather_('variable', 'value', c('Sepal.Length', 'Sepal.Width', 'Petal.Length', 'Petal.Width')) %>% tidyr::separate_('variable', into = c('Parts', 'Scale'), sep='\\.') # 略
正規表現でグルーピングした値で列分割したい場合は tidyr::extract。
df %>% tidyr::gather(variable, value, c(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width)) %>% tidyr::extract(variable, into = c('Parts', 'Scale'), regex='(.+)\\.(.+)') # Source: local data frame [600 x 4] # # Species Parts Scale value # 1 setosa Sepal Length 5.1 # 2 setosa Sepal Length 4.9 # 3 setosa Sepal Length 4.7 # .. ... ... ... ... # Standard evaluation df %>% tidyr::gather_('variable', 'value', c('Sepal.Length', 'Sepal.Width', 'Petal.Length', 'Petal.Width')) %>% tidyr::extract_('variable', into = c('Parts', 'Scale'), regex='(.+)\\.(.+)') # 略
複数列の値を一列に結合
tidyr::separateと逆の操作はtidyr::uniteでできる。
df %>% tidyr::gather(variable, value, c(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width)) %>% tidyr::separate(variable, into = c('Parts', 'Scale'), sep='\\.') %>% tidyr::unite('variable', c(Parts, Scale), sep='.') # Source: local data frame [600 x 3] # # Species variable value # 1 setosa Sepal.Length 5.1 # 2 setosa Sepal.Length 4.9 # 3 setosa Sepal.Length 4.7 # .. ... ... ... # Standard evaluation df %>% tidyr::gather_('variable', 'value', c('Sepal.Length', 'Sepal.Width', 'Petal.Length', 'Petal.Width')) %>% tidyr::separate_('variable', into = c('Parts', 'Scale'), sep='\\.') %>% tidyr::unite_('variable', c('Parts', 'Scale'), sep='.') # 略