R {ggplot2} で 少し複雑なサブプロットが描きたい
この記事は R Advent Calendar 2015 4 日目の記事です。
{ggplot2} でのサブプロット
{ggplot2} でサブプロットを描画したいことがある。同じ種類のプロットを水準別に描画するなど、単純なものであれば facet で描ける。例えば 適当な散布図を Species 別に描きたい場合、
library(dplyr) library(ggplot2) ggplot(iris, aes(Sepal.Width, Sepal.Length)) + geom_point() + facet_wrap(~ Species)

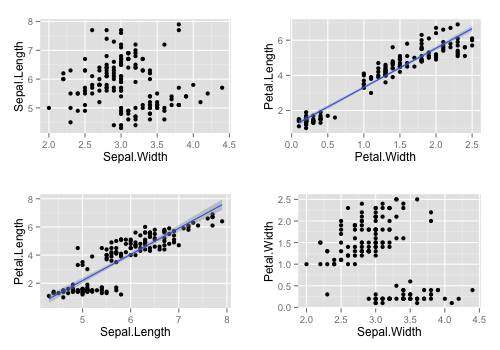
facet では異なる種類のプロットをサブプロットとすることはできない。例えば散布図の縦軸/横軸をそれぞれ変えたい、といった場合には gridExtra::grid.arrange を使う。
library(gridExtra) p1 <- ggplot(iris, aes(Sepal.Width, Sepal.Length)) + geom_point() p2 <- ggplot(iris, aes(Petal.Width, Petal.Length)) + geom_point() p3 <- ggplot(iris, aes(Sepal.Length, Petal.Length)) + geom_point() p4 <- ggplot(iris, aes(Sepal.Width, Petal.Width)) + geom_point() grid.arrange(p1, p2, p3, p4)

が、grid.arrange は引数を ドット ... で受け取ること、また呼び出すと直ちに描画を行うことから、以下のような場合は少し面倒だ。
- 描画が直ちに行われるため、サブプロットの描画設定をまとめて変更できない。単一のテーマを使う場合でも、個々のプロットそれぞれについてあらかじめ処理を行っておく必要がある。
- リストが渡せない。サブプロットの数を動的に変更したい場合は、リストに入れて
do.callが必要である。
というわけで、自作パッケージ {ggfortify} でこの辺の操作を少し便利にできるようにした。サブプロット周りの処理は ggmultiplot クラスで行う。
コンストラクタの引数にはリストが渡せるため、サブプロット数が変わる場合でも楽だと思う。また、インスタンスに対して + 演算子を用いることで ggplot と同じ処理を各プロットに対して適用できる。
# install.packages('ggfortify') library(ggfortify) p <- new('ggmultiplot', plots = list(p1, p2, p3, p4)) p # 略。上記 grid.arrange と同じ出力 # theme_bw を全サブプロットに適用 p + theme_bw()

サブプロットの要素には リストと同じようにアクセスできる。[ でのアクセスは ggmultiplot インスタンスに、[[ での アクセスは ggplot インスタンスになる。代入もできるため、一部のプロットにだけ処理を適用したい場合は以下のように書ける。
p[2:3] <- p[2:3] + stat_smooth(method = 'lm') p
+ 演算子の右項に ggplot インスタンスを指定するとサブプロットが追加できる。
p + (ggplot(iris, aes(Sepal.Width, fill = Species)) + geom_density())
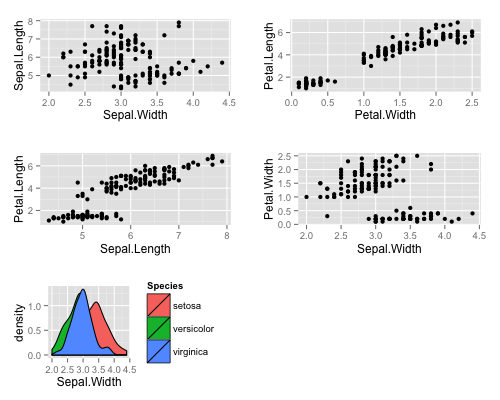
ggmultiplot 作成時に ncol もしくは nrow キーワードを指定することで、サブプロットの列数 / 行数が指定できる。
p <- new('ggmultiplot', plots = list(p1, p2, p3, p4), ncol = 3) p

autoplot との組み合わせ
{ggfortify} を使うと主要なクラスを ggplot2::autoplot 関数で描画できるようになる。autoplot にリストを渡した場合は、リストの各要素が autoplot 可能であればそれらをサブプロットとして描画する。
サンプルとして、{broom} パッケージの vignettes にある例をやってみたい。ここでは、適当なデータに対して クラスタ数を変えながら kmeans クラスタリングを行った結果をプロットしている。
これは autoplot を使って以下のように書ける。
kclusts <- data.frame(k=1:9) %>% dplyr::group_by(k) %>% dplyr::do(kclust = kmeans(iris[-5], .$k)) autoplot(kclusts$kclust, data = iris[-5], ncol = 3) + theme(legend.position = "none")
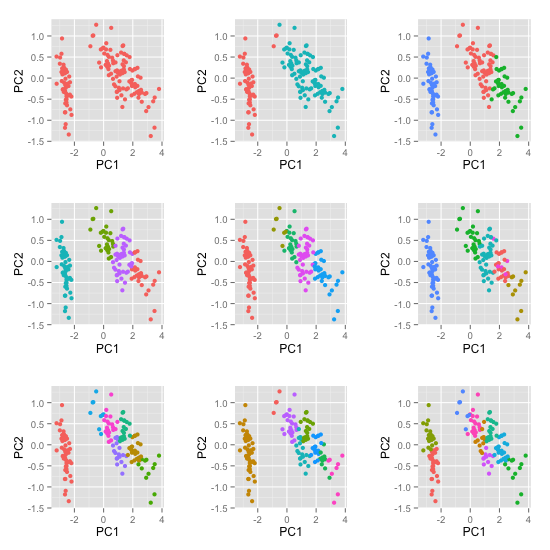
一行目は今なら {purrr} を使ったほうが簡潔でわかりやすくなると思う。
library(purrr) kclusts <- purrr::map(1:9, ~ kmeans(iris[-5], .)) autoplot(kclusts, data = iris[-5], ncol = 3) + theme(legend.position = "none") # 略
まとめ
{ggfortify} では ggmultiplot クラス、ならびに autoplot を利用してサブプロットを簡単に扱うことができる。

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
{purrr} でリストデータを操作する <2>
前の記事に続けて、{purrr} で {rlist} 相当の処理を行う。今回はレコードの選択とソート。
サンプルデータは前回と同じものを利用する。リストの表示も同じく Hmisc::list.tree を使う。
library(rlist) library(pipeR) library(purrr) packages <- list( list(name = 'dplyr', star = 979L, maintainer = 'hadley' , authors = c('hadley', 'romain')), list(name = 'ggplot2', star = 1546L, maintainer = 'hadley' , authors = c('hadley')), list(name = 'knitr', star = 1047L, maintainer = 'yihui' , authors = c('yihui', 'hadley', '...and all')) ) packages ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[3]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
レコードの選択
rlist Tutorial: Filtering 後半の内容。
ある条件にあてはまるレコードのうち最初の N 件だけを取得したいとき、{rlist} には専用の関数 list.find がある。{purrr} には対応する関数はないが、keep %>% head で同じ結果が得られる。
packages %>>% rlist::list.find(star >= 1000, 1) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::keep(~ .$star >= 1000) %>% head(n = 1) # 略
また、{rlist} には list.findi という インデックスを返す関数がある。purrr::keep では、引数に logical 型のベクトルを渡せば TRUE に対応する要素のみを残すことができる。
packages %>>% rlist::list.findi(star >= 1000, 2) ## [1] 2 3 purrr::keep(seq_along(packages), flatmap(packages, ~ .$star >= 1000)) %>% head(n = 2) # 略
条件にあてはまる最初のレコードだけを取得したいときは rlist::first もしくは purrr::detect。
rlist::list.first(packages, star >= 1000) ## x = list 4 (792 bytes) ## . name = character 1= ggplot2 ## . star = integer 1= 1546 ## . maintainer = character 1= hadley ## . authors = character 1= hadley purrr::detect(packages, ~ .$star >= 1000) # 略
レコード数がそこまで多くない場合は keep %>% head(n = 1) でよいと思うが、結果に 1 レベル目が入れ子として残ってしまう。上と同じ結果を得るには purrr::flatten ( unlist(recursive = FALSE) と同じ ) を通す。
purrr::keep(packages, ~ .$star >= 1000) %>% head(n = 1) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley purrr::keep(packages, ~ .$star >= 1000) %>% head(n = 1) %>% purrr::flatten() ## x = list 4 (792 bytes) ## . name = character 1= ggplot2 ## . star = integer 1= 1546 ## . maintainer = character 1= hadley ## . authors = character 1= hadley
同じく、最後のレコードを取得したいときは rlist::last もしくは purrr::detect(.right = TRUE)。これも自分としては keep %>% tail(n = 1) のほうが覚えやすい。
list.last(packages, star >= 1000) ## x = list 4 (920 bytes) ## . name = character 1= knitr ## . star = integer 1= 1047 ## . maintainer = character 1= yihui ## . authors = character 3= yihui hadley ... purrr::detect(packages, ~ .$star >= 1000, .right = TRUE) # 略 purrr::keep(packages, ~ .$star >= 1000) %>% tail(n = 1) %>% purrr::flatten() # 略
類似の処理として、{rlist} には list.take という最初の N レコードを取得する関数と list.skip という最初の N レコードを除外する関数がある。{purrr} には対応する関数はないが、組み込みの head と tail でよいのでは...。
packages %>>% rlist::list.take(2) ## x = list 2 (1696 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% head(n = 2) # 略 packages %>% rlist::list.skip(1) ## x = list 2 (1768 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... packages %>% tail(length(.) - 1) # 略
ある条件が初めて偽となるまでレコードを取得する場合は rlist::list.takeWhile もしくは purrr::head_while。
packages %>>% rlist::list.takeWhile(star <= 1500) ## x = list 1 (896 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain packages %>% purrr::head_while(~ .$star <= 1500) # 略
ある条件が初めて偽となったレコード以降を取得したい場合は rlist::list.skipWhile。{purrr} には対応する処理はないが、purrr::detect_index を使って以下のように書ける。
packages %>>% rlist::list.skipWhile(star <= 1500) ## x = list 2 (1768 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... purrr::detect_index(packages, ~ .$star > 1500) ## [1] 2 packages[purrr::detect_index(packages, ~ .$star > 1500):length(packages)] # 略
一方、{purrr} には tail_while という末尾からみて条件にあてはまるレコードだけを取得する関数がある。
packages %>% purrr::tail_while(~ .$star <= 1500) ## x = list 1 (968 bytes) ## . [[1]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
ある条件を満たす要素がいくつあるかを調べるには rlist::list.count。{purrr} なら keep %>% length でよい。
list.count(packages, star > 1000) ## [1] 2 purrr::keep(packages, ~ .$star > 1000) %>% length() # 略
{rlist} には リストの要素の名前を正規表現で抽出する list.match がある。{purrr} なら keep でできる。stringr::str_match はマッチした結果を matrix で返すため、complete.cases で logical に変換する。
data <- list(p1 = 1, p2 = 2, a1 = 3, a2 = 4) list.match(data, "p[12]") ## x = list 2 (416 bytes) ## . p1 = double 1= 1 ## . p2 = double 1= 2 complete.cases(stringr::str_match(names(data), 'p[12]')) ## [1] TRUE TRUE FALSE FALSE keep(data, complete.cases(stringr::str_match(names(data), 'p[12]'))) # 略
logical の処理
条件に当てはまるかどうかを logical ベクトルとして返すには rlist::list.is。これは rlist::list.mapv と同じなのでは...? {purrr} なら flatmap もしくは map_lgl。
packages %>>% rlist::list.is(star < 1500) ## [1] TRUE FALSE TRUE packages %>>% rlist::list.mapv(star < 1500) # 略 packages %>% purrr::flatmap(~ .$star < 1500) # 略 packages %>% purrr::map_lgl(~ .$star < 1500) # 略
また、logical のリスト全体の真偽値を以下のように取得できる
- 要素全てが
TRUEかどうか:rlist::list.allもしくはpurrr::every。 - 要素いずれかが
TRUEかどうか:rlist::list.anyもしくはpurrr::some。
自分は purrr::flatmap %>% all もしくは purrr::flatmap %>% any のほうが覚えやすい。
ただし、{purrr} v0.1.0 の every, some にはバグがあり正しく動作しない。下のスクリプトを実行するためには GitHub から最新の master をインストールする必要がある。
rlist::list.all(packages, star >= 500) ## [1] TRUE # install_github('hadley/purrr') # が必要 purrr::every(packages, ~ .$star >= 500) # 略 packages %>% purrr::flatmap(~ .$star >= 500) %>% all() # 略 rlist::list.any(packages, star >= 1500) ## [1] TRUE # install_github('hadley/purrr') # が必要 purrr::some(packages, ~ .$star >= 1500) packages %>% purrr::flatmap(~ .$star >= 1500) %>% any() # 略
要素の削除
{rlist} では list.remove で指定した名前をリストから除外できる。{purrr} には discard という条件にマッチする要素をリストから除外する関数がある ( purrr::keep とは逆 )。条件の否定をとれば keep でも書ける。
rlist::list.remove(data, c("p1", "p2")) ## x = list 2 (416 bytes) ## . a1 = double 1= 3 ## . a2 = double 1= 4 purrr::discard(data, names(data) %in% c("p1", "p2")) # 略 purrr::keep(data, !(names(data) %in% c("p1", "p2"))) # 略
{rlist} で、名前ではなく 条件式を使ってリストから除外したい場合は list.exclude。{purrr} の場合は 上と同じく discard もしくは keep。
packages %>>% rlist::list.exclude("yihui" %in% authors) ## x = list 2 (1696 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::discard(~ "yihui" %in% .$authors) # 略
また、{rlist} には 欠損などの異常値を リストから除去する list.clean という関数がある。下の例では リストの 1 レベル目にある欠損 "b" を除去している。{purrr} では同じく discard。
x <- list(a = 1, b = NULL, c = list(x = 1, y = NULL, z = logical(0L), w = c(NA, 1))) x ## x = list 3 (1040 bytes) ## . a = double 1= 1 ## . b = NULL 0 ## . c = list 4 ## . . x = double 1= 1 ## . . y = NULL 0 ## . . z = logical 0 ## . . w = double 2= NA 1 rlist::list.clean(x) ## x = list 2 (960 bytes) ## . a = double 1= 1 ## . c = list 4 ## . . x = double 1= 1 ## . . y = NULL 0 ## . . z = logical 0 ## . . w = double 2= NA 1 purrr::discard(x, ~ is.null(.)) # 略
rlist::list.clean は recursive = TRUE を指定することで 処理を再帰的に適用できる。{purrr} ではこういった再帰的な処理は (自分で関数を書かない限り) できないと思う。
rlist::list.clean(x, recursive = TRUE) ## x = list 2 (912 bytes) ## . a = double 1= 1 ## . c = list 3 ## . . x = double 1= 1 ## . . z = logical 0 ## . . w = double 2= NA 1
要素の更新
rlist Tutorial: Updating に相当する内容。
rlist::list.map や purrr::map を用いたリストの選択では、結果に含める属性を明示的に指定する必要があった。
これは対象のリストに選択したい属性が多い場合はすこし面倒だ。
rlist::list.update を使うと、もともとのリストの属性を残した上で 指定した要素を追加 / 更新できる。{purrr} で同じことをするには map + update_list。
packages %>>% rlist::list.update(lang = 'R', star = star + 1) ## x = list 3 (3160 bytes) ## . [[1]] = list 5 ## . . name = character 1= dplyr ## . . star = double 1= 980 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . . lang = character 1= R ## . [[2]] = list 5 ## . . name = character 1= ggplot2 ## . . star = double 1= 1547 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . . lang = character 1= R ## . [[3]] = list 5 ## . . name = character 1= knitr ## . . star = double 1= 1048 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . . lang = character 1= R packages %>% purrr::map(~ purrr::update_list(., lang = 'R', star = ~ .$star + 1)) # 略
一部の要素を取り除きたい場合は NULL を指定する。これは {purrr} も同じ。
packages %>>% rlist::list.update(maintainer = NULL, authors = NULL) ## x = list 3 (1464 bytes) ## . [[1]] = list 2 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . [[2]] = list 2 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . [[3]] = list 2 ## . . name = character 1= knitr ## . . star = integer 1= 1047 packages %>% purrr::map(~ purrr::update_list(., maintainer = NULL, authors = NULL)) # 略
ソート
rlist Tutorial: Sorting に相当する内容。
{rlist}, {purrr} とも 標準の sort ( 値のソート ) と order ( インデックスのソート ) それぞれに対応した関数を持つ。
x = c('a', 'c', 'd', 'b') sort(x) ## [1] "a" "b" "c" "d" order(x) ## [1] 1 4 2 3
sort のようにソートした値を得るためには rlist::list.sort もしくは purrr::sort_by。
packages %>>% rlist::list.sort(star) ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . [[3]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::sort_by('star') # 略
{rlist} では 列名を括弧でくくると逆順となる。{purrr} には対応する記法がないが、結果を rev で逆順にすればよい。
packages %>>% rlist::list.sort((star)) ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . [[3]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain packages %>% purrr::sort_by('star') %>% rev() # 略
order のように結果をインデックスで得るためには rlist::list.order もしくは purrr::order_by。
rlist::list.order(packages, star) ## [1] 1 3 2 purrr::order_by(packages, 'star') ## [1] 1 3 2
まとめ
前回と同じく、{purrr} では map (flatmap)、keep (discard) でだいたいのことはできる。処理を少し変えたい場合は 組み込み関数を適当に組み合わせればよいため、追加での学習コストは低いと思う。
一方、{rlist} はパッケージの中だけで処理が完結するので、そちらにメリットを感じる方は {rlist} を使ったほうがよさそう。
さらに続きます。

- 作者: Hadley Wickham,石田基広,市川太祐,高柳慎一,福島真太朗
- 出版社/メーカー: 共立出版
- 発売日: 2016/01/23
- メディア: 単行本
- この商品を含むブログ (25件) を見る
{purrr} でリストデータを操作する <1>
R で関数型プログラミングを行うためのパッケージである {purrr}、すこし使い方がわかってきたので整理をしたい。RStudio のブログの記載をみると、とくにデータ処理フローを関数型のように記述することが目的のようだ。
The core of purrr is a set of functions for manipulating vectors (atomic vectors, lists, and data frames). The goal is similar to dplyr: help you tackle the most common 90% of data manipulation challenges.
ここでいう"関数型プログラミング言語"とは Haskell のような静的型の言語を想定しており、型チェック、ガード、リフトなど断片的に影響を受けたと思われる関数群をもつ。ただし、同時に Haskell を目指すものではない、とも明言されている。
そもそも R は Scheme の影響をうけているためか、関数型らしい言語仕様やビルトイン関数群 Map, Reduce, Filter などをすでに持っている。ただ、これらの関数群は引数の順序からパイプ演算子と組み合わせて使いにくい。{purrr} でこのあたりが使いやすくなっているとうれしい。
R: Common Higher-Order Functions in Functional Programming...
※ 上記の Map など、S のリファレンスに見当たらなかったため R 独自だと思っていますが、間違っていたら教えてください。
{purrr} によるリストの操作
{purrr} を使うとリストやベクトルの処理が書けるのかをまとめたい。リスト操作のためのパッケージである {rlist} とできること、記法をくらべてみる。
{purrr} を利用する目的は 処理を簡単に、見やすく書くことであるため、{rlist} と比べて可読性が悪いもの、複雑になるものは "できない" 処理として扱う。サンプルデータとして、R の著名パッケージに関連した情報のリストを用意した。
packages <- list( list(name = 'dplyr', star = 979L, maintainer = 'hadley' , authors = c('hadley', 'romain')), list(name = 'ggplot2', star = 1546L, maintainer = 'hadley' , authors = c('hadley')), list(name = 'knitr', star = 1047L, maintainer = 'yihui' , authors = c('yihui', 'hadley', '...and all')) ) packages ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[3]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
1レベル目がレコード、以降のレベルが各レコードの要素となっている。この記事では主に以下二つの操作に関連した機能について記載する。
- 要素に対する操作: 特定の要素を選択する。特定の要素を元に新しい要素をつくる (マッピングする)。
- レコードに対する操作: 特定のレコードを選択する。
準備: リストの表示
既定の print ではリストの階層構造がわかりにくく、出力も長い。わかりやすくするため Hmisc::list.tree を利用した出力を記載する。また、出力が同一のものは省略する。
library(Hmisc) l1 <- list(a = 1, b = c(3L, 4L), c = list(x = c(5, 6), y = 'AAA')) # 既定での表示 l1 ## $a ## [1] 1 ## ## $b ## [1] 3 4 ## ## $c ## $c$x ## [1] 5 6 ## ## $c$y ## [1] "AAA" # list.tree での表示 (名前 = 型 要素数 = 値 と読む) Hmisc::list.tree(l1) ## l1 = list 3 (968 bytes) ## . a = double 1= 1 ## . b = integer 2= 3 4 ## . c = list 2 ## . . x = double 2= 5 6 ## . . y = character 1= AAA
パッケージのロード
library(rlist) library(pipeR) library(purrr) library(dplyr)
要素の選択とマッピング
rlist Tutorial: Mapping に相当する内容。
要素の選択
R 標準の lapply 他 apply 系の関数に対応するのは rlist::list.map と purrr::map。purrr::map では第二引数に文字列を渡すとその要素の抽出、ラムダ式 ( ~ .$name )を渡すとその式の適用になる。
lapply(packages, function(x) { x$name }) ## x = list 3 (360 bytes) ## . [[1]] = character 1= dplyr ## . [[2]] = character 1= ggplot2 ## . [[3]] = character 1= knitr rlist::list.map(packages, name) # 略 purrr::map(packages, 'name') # 略 purrr::map(packages, ~ .$name) # 略
複数の要素を一度に選択したい場合、{rlist} には rlist::list.select という選択専用の関数がある。{purrr} では渡せる式が一つのため、ラムダ式を利用する。
rlist::list.select(packages, maintainer, star) ## x = list 3 (1488 bytes) ## . [[1]] = list 2 ## . . maintainer = character 1= hadley ## . . star = integer 1= 979 ## . [[2]] = list 2 ## . . maintainer = character 1= hadley ## . . star = integer 1= 1546 ## . [[3]] = list 2 ## . . maintainer = character 1= yihui ## . . star = integer 1= 1047 purrr::map(packages, ~ .[c('maintainer', 'star')]) # 略
補足 第二引数にベクトルを渡した場合の処理は、以下のように階層的な選択になる ( l[[c('a', 'b')]] と一緒)。
l2 <- list(list(a=list(b=1)), list(a=list(b=2))) l2 ## x = list 2 (1176 bytes) ## . [[1]] = list 1 ## . . a = list 1 ## . . . b = double 1= 1 ## . [[2]] = list 1 ## . . a = list 1 ## . . . b = double 1= 2 purrr::map(l2, c('a', 'b')) ## x = list 2 (152 bytes) ## . [[1]] = double 1= 1 ## . [[2]] = double 1= 2
{rlist}, {purrr} でのラムダ式
f <- function(.) {. + 1} に対応する無名関数を {rlist}, {purrr} それぞれの記法で書く。形式はチルダの有無以外は同じ。ドットが引数に対応する。
nums <- c(a = 3, b = 2, c = 1) rlist::list.map(nums, . + 1) ## x = list 3 (544 bytes) ## . a = double 1= 4 ## . b = double 1= 3 ## . c = double 1= 2 purrr::map(nums, ~ . + 1) # 略
{rilst} では ラムダ式中の .i で元のリストの位置 (インデックス) を、.name で名前 (names) をそれぞれ参照できる。purrr のラムダ式には対応する記法がないため、近いことをやるためには直接 map の引数として渡す必要がある。
rlist::list.map(nums, .i) ## x = list 3 (544 bytes) ## . a = integer 1= 1 ## . b = integer 1= 2 ## . c = integer 1= 3 purrr::map(seq_along(nums), ~ .) # namesは変わってしまう ## x = list 3 (216 bytes) ## . [[1]] = integer 1= 1 ## . [[2]] = integer 1= 2 ## . [[3]] = integer 1= 3
rlist::list.map(nums, paste0("Name: ", .name)) ## x = list 3 (688 bytes) ## . a = character 1= Name: a ## . b = character 1= Name: b ## . c = character 1= Name: c purrr::map(names(nums), ~ paste0("Name: ", .)) # namesは変わってしまう ## x = list 3 (360 bytes) ## . [[1]] = character 1= Name: a ## . [[2]] = character 1= Name: b ## . [[3]] = character 1= Name: c
また、内部的にはラムダ式を eval で評価をしているため、変数として扱われない位置にあるドットは評価されない。
purrr::map(nums, ~ list(. = 1)) ## x = list 3 (1312 bytes) ## . a = list 1 ## . . . = double 1= 1 ## . b = list 1 ## . . . = double 1= 1 ## . c = list 1 ## . . . = double 1= 1
要素の追加、変更
元となるリストの値から新しいリストを作りたい場合はラムダ式でリストを返す。
rlist::list.map(packages, list(star = star, had = 'hadley' %in% authors)) ## x = list 3 (1320 bytes) ## . [[1]] = list 2 ## . . star = integer 1= 979 ## . . had = logical 1= TRUE ## . [[2]] = list 2 ## . . star = integer 1= 1546 ## . . had = logical 1= TRUE ## . [[3]] = list 2 ## . . star = integer 1= 1047 ## . . had = logical 1= TRUE purrr::map(packages, ~ list(star = .$star, had = 'hadley' %in% .$authors)) # 略
結果の型の変更
結果をベクトルで取得したい場合、{rilst} では list.mapv。{purrr} では flatmap もしくは map_int ( map_xxx のように結果の型を指定できる関数群がある )。
rlist::list.mapv(packages, star) ## [1] 979 1546 1047 purrr::flatmap(packages, 'star') # 略 purrr::map_int(packages, 'star') # 略
data.frame としたい場合は rlist::list.stack。{purrr} では dplyr::bind_rows に渡す。
packages %>>% rlist::list.select(name, star) %>>% rlist::list.stack() ## name star ## 1 dplyr 979 ## 2 ggplot2 1546 ## 3 knitr 1047 packages %>% purrr::map(~ .[c('name', 'star')]) %>% dplyr::bind_rows()) # 略
関数の適用
返り値を変更せずに関数を適用するには rlist::list.iter と purrr::walk。パイプ処理の間に出力やプロットなど意図する返り値を持たない処理を挟み込む場合に利用する。
r <- rlist::list.iter(packages, cat(name, ":", star, "\n")) ## dplyr : 979 ## ggplot2 : 1546 ## knitr : 1047 r <- purrr::walk(packages, ~ cat(.$name, ":", .$star, "\n")) # 略
レコードの選択
rlist Tutorial: Filtering 前半の単純な条件での選択。
{rilst} では list.filter。{purrr} では keep。ラムダ式が使えるのは map などと同じ。
packages %>>% list.filter(star >= 1500) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::keep(~ .$star >= 1500) # 略
packages %>>% list.filter("yihui" %in% authors) ## x = list 1 (968 bytes) ## . [[1]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... packages %>% purrr::keep(~ "yihui" %in% .$authors) # 略
まとめ
{purrr} によるリストデータの属性、レコードに対する操作を記載した。purrr::map と purrr::keep だけでもパイプ演算子 + ラムダ式と組み合わせて幅広い処理ができそうだ。
11/28追記 続きです。

- 作者: Hadley Wickham,石田基広,市川太祐,高柳慎一,福島真太朗
- 出版社/メーカー: 共立出版
- 発売日: 2016/01/23
- メディア: 単行本
- この商品を含むブログ (25件) を見る
R パッケージを CRAN で公開する
少し前に 自作パッケージを CRAN で公開したのだが ブログに書くのを忘れていた。CRAN 公開時の注意点に関して、日本語の説明があまりない / 情報が古いので簡単にまとめたい。
パッケージの作成
この資料を読みましょう。
www.slideshare.net
継続的インテグレーション (CI)
Travis CI は R をサポート (community supportだが) しているため、.travis.yml に2行記載するだけで利用できる。CI 上でパッケージのチェック (R CMD check) も走るので利用したほうが楽。
複数の環境でテストを実行したい場合、Travis CI では Build Matrix という仕組みがある。R では実行したいテストの数だけ env: タグ中に環境変数を記載するのがよいと思う。自分のパッケージでは {ggplot2} の公式版 / GitHub 上の開発版で 2 つのテストを実行している。
そのほかの CI ツール
ほか、R から CI を利用するツールとして、以下のようなものもある。
- r-appveyor: Windows 用の CI 環境である AppVeyor が利用できる。
- r-builder: Travis CI を R から利用する。R-devel が利用できる。
vignettes の作成
11/30追記 {knitr} で作るのがカンタン。vignettes 化したい {knitr}ドキュメント中に以下のようなタグを含めればよい。VignetteIndexEntry は vignettes へのリンクテキストとして利用される。ASCII文字以外を含める場合、DESCRIPTION ファイル中で Encoding: UTF-8 を指定する。
<!--
%\VignetteEngine{knitr::knitr}
%\VignetteIndexEntry{Introduction to ggfortify package}
-->
参考 Package vignettes: How to build package vignettes with knitr | knitr
パッケージのチェック
開発版 (R-devel) の日次ビルド を利用して、R CMD Check --as-cran package_name.tar.gz で行う。開発版のほうが警告が厳しく出るようだ。
自分は 開発版環境を AWS 上に構築している。 Amazon Linux であれば以下のスクリプトでインストールできると思う。
R CMD check では ERROR, WARNING はもちろん、NOTE も "checking CRAN incoming feasibility" 1 つを除いて全て表示されないようにするのが望ましい。
特に、{dplyr} 等で NSE を使っている場合は xxx: no visible binding for global variable 'xxx' といった NOTE が表示されるが、これらについても CRAN 管理者から指摘される場合がある。 SE を使うか、関数中に (ダミーとして) 同名の変数を作るなどで回避しておくのがよい。
パッケージのサブミット
FTP で直接あげてはダメ。Submit package to CRAN に必要事項を記載して Upload package をする。その後、確認のメールが来たら Confirmation する。
公開待ちのパッケージは 以下の FTP サーバにて確認できる。
パッケージによっては 拡張子の後に以下のようなステータス情報が付けられる場合がある。これは CRAN 公式の情報ではなく、いくつかのフォーラム文書をみて自分が推測したものなので間違っているかもしれない。
.save:UploadされたがConfirmationされていないもの。.pending: CRAN 管理者にて何らかの理由でペンディングされているもの (放っておくしかない)。.noemail:Confirmationはされているが確認時のメールが CRAN に飛んでいないもの。
参考 What are the steps in submitting an R-package to CRAN and how long does each step take? - Stack Overflow (ほぼ情報ない)
その後の対応
CRAN 管理者から指摘がきたら適切に対応してください。
パッケージの公開
11/30追記 CRAN 上でのテストとレビューに問題がなければ CRAN 公開後にメールがくる。その後 1日程度すると Windows 用のバイナリのビルドが自動的に行われ、改めて通知がくる。
岩波データサイエンス Vol.1
ご恵贈いただきました。 ありがとうございます! あわせてタスクもいただきました (下部)。
書籍のコンテンツ
各章ごとの内容は id:sfchaos さんが詳しく紹介されています。
まだ すべて読めていないのですが、以下 3 点がよいポイントだと思います。
- 理論 と サンプルプログラム 両方の記載がある
BUGS,Stan,PyMC3と主要なパッケージが網羅されている- サンプルは単純な回帰だけでなく 時系列 / 空間ベイズを含む
補足 書籍には コラム "Pythonとは" という データ分析視点での Python 紹介があるのですが、中身は結構な pandas 推しでした。著者の方、いったい何者なんだ...。
Stan 入門
依頼により、著者の松浦さんが作成した RStan サンプルの PyStan 版を作成させていただきました。
以下リポジトリの "Example..." フォルダ中に含まれる Jupyter Notebook ( .ipynb ) を開くと、 GitHub 上でプログラムと出力を確認することができます。
RStan と PyStan の API 差異については公式ドキュメントに記載がありますがアップデートがされていません。下表にサンプル中で利用したものを簡単にまとめます。
RStan |
PyStan |
概要 |
|---|---|---|
stan(...) |
pystan.stan(...) |
モデルのコンパイルとサンプリングの実行 |
stan_model(...) |
pystan.StanModel(...) |
モデルのコンパイル |
sampling(...) |
StanModel.sampling(...) |
サンプリングの実行 |
extract(...) |
StanFit4model.extract() |
サンプリング結果を取得 |
traceplot(...) |
StanFit4model.plot(...) |
サンプリングの経過をプロット |
書籍や Rstan のサンプルと合わせてご参照ください。

- 作者: 岩波データサイエンス刊行委員会
- 出版社/メーカー: 岩波書店
- 発売日: 2015/10/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (4件) を見る
{rbokeh} で Bokeh を R から使いたい
はじめに
Bokeh は Python 以外にも R, Scala, Julia 用のパッケージを提供している。パッケージといっても Python の Bokeh と連携するものではなく、Bokeh がブラウザでのレンダリングに使っている Bokeh.js を各言語で扱えるようにするもののようだ。そのため、各パッケージはそれぞれ単体で利用できる。
うち、R 用のパッケージである {rbokeh} を使ってみたい。R には {htmlwidgets} を使った JavaScript 利用の可視化パッケージが多数提供されているが、数が多くて使い方が覚えられない。できれば {rbokeh} だけを使いたい。また、Pythonと見た目が揃えられると嬉しい。
インストール
現時点で CRAN には公開されていないため、GitHub からインストールする。現時点のバージョンは 0.2.3.2。
install.packages('htmlwidgets') install.packages('devtools') library(devtools) devtools::install_github('bokeh/rbokeh')
データの準備
サンプルとして {ggplot2} に含まれる mpg データを利用する。単に {rbokeh} を利用するだけなら {ggplot2} は不要。
library(ggplot2) library(rbokeh) dim(mpg) # [1] 234 11 head(mpg) # manufacturer model displ year cyl trans drv cty hwy fl class # 1 audi a4 1.8 1999 4 auto(l5) f 18 29 p compact # 2 audi a4 1.8 1999 4 manual(m5) f 21 29 p compact # 3 audi a4 2.0 2008 4 manual(m6) f 20 31 p compact # 4 audi a4 2.0 2008 4 auto(av) f 21 30 p compact # 5 audi a4 2.8 1999 6 auto(l5) f 16 26 p compact # 6 audi a4 2.8 1999 6 manual(m5) f 18 26 p compact
基本的な操作
Python の Bokeh は matplotlib を連想させる API を持っているが、 {rbokeh} は別の API ( {ggplot2} や {ggvis} に近い ) を持つ。そのため、API は言語ごとに覚える必要がある。
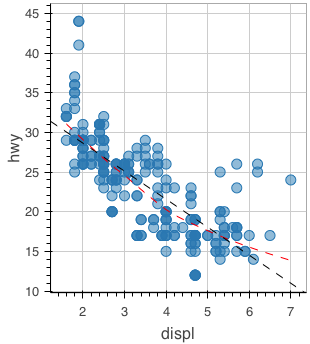
mpg データの displ 列と hwy 列を利用して散布図を描画する。
figure() %>% ly_points(x = displ, y = hwy, data = mpg)

加えて、 class 列で色分けする。サイズの変更など、利用できるオプションは help(ly_points) で確認できる。
figure() %>% ly_points(x = displ, y = hwy, color = class, data = mpg)

色名は文字列で指定することもできる。
figure() %>% ly_points(x = displ, y = hwy, color = 'red', data = mpg)
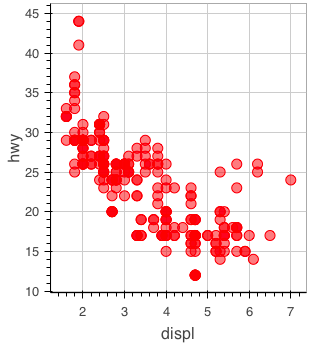
NSE と SE
{rbokeh} では、{ggplot2} のように NSE ( Non-standard evaluation )と SE ( Standard evaluation ) 用の関数を明示的に分けていない。そのため、一部のオプションは以下のようにSEでも動作する。
# scatter figure() %>% ly_points(x = 'displ', y = 'hwy', data = mpg) # 出力省略
が、 SE では動作しないものもあるため、現時点では NSE を使ったほうがよさそうだ。
# scatter figure() %>% ly_points(x = 'displ', y = 'hwy', color = 'class', data = mpg) # 以下にエラー nchar(opts[[fld]]) : 'nchar()' は文字ベクトルを要求します
平滑化 / 回帰直線の描画
R のモデルを関数に渡すことで描画できる。lowess に対しては ly_lines を、lm に対しては ly_abline を用いる。この使い分けはちょっと覚えにくい。
figure() %>% ly_points(x = displ, y = hwy, data = mpg) %>% ly_lines(lowess(mpg$displ, mpg$hwy), color = "red", type = 2) %>% ly_abline(lm(hwy ~ displ, data = mpg), type = 2)
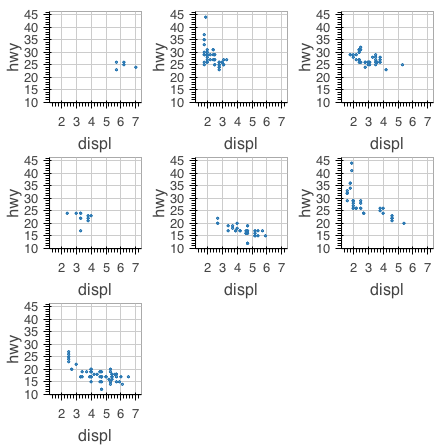
サブプロット ( facet ) の描画
{htmlwidgets} ブログ中の {rbokeh} 紹介記事 では、サブプロットを lapply + {pipeR} を使って描画する例が記載されている。自分は {dplyr} 好きなので {dplyr} でやりたい。
library(dplyr) mpg %>% dplyr::group_by(class) %>% dplyr::do(dummy = ly_points(figure(width = 200, height = 200), x = displ, y = hwy, data = ., size = 2)) %>% { as.list(.[['dummy']]) } %>% grid_plot(nrow = 3, ncol = 3, same_axes = TRUE )
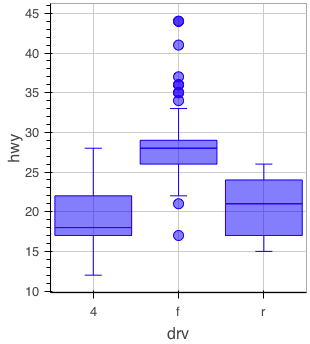
様々なプロット
以降、{rbokeh} で描画できるプロットを例示する。
箱ひげ図
figure() %>% ly_boxplot(x = drv, y = hwy, data = mpg)
ヒストグラム
figure() %>% ly_hist(x = hwy, data = mpg)
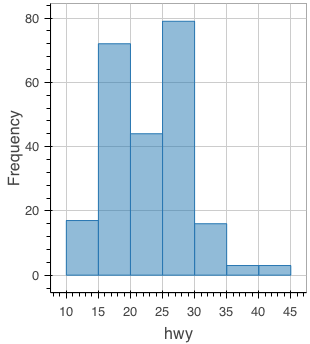
ヒストグラムをグループ別に描画するには少し手間が必要。グループ別に塗り分けるためのカラーパレットを用意し、Reduce で各グループ別のヒストグラムを追加する。
library(scales) colors <- scales::hue_pal()(length(levels(mpg$class))) colors <- setNames(colors, levels(mpg$class)) colors # 2seater compact midsize minivan pickup subcompact suv # "#F8766D" "#C49A00" "#53B400" "#00C094" "#00B6EB" "#A58AFF" "#FB61D7" ghist <- function(fig, group) { ly_hist(fig, x = hwy, data = dplyr::filter(mpg, class == group), breaks = seq(10, 45, 5), color = colors[[group]]) } Reduce(ghist, levels(mpg$class), figure())
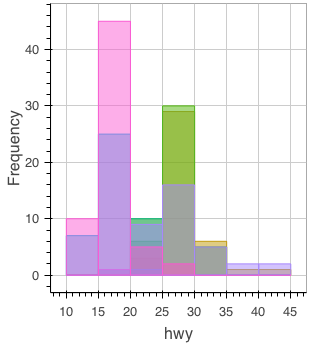
カーネル密度推定のプロット
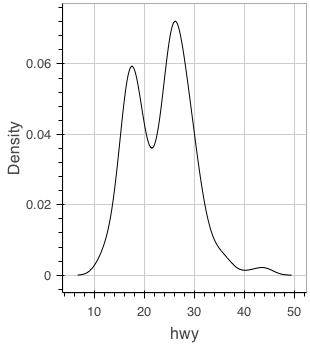
グループ分けしたい場合はヒストグラム同様の処理が必要。
figure() %>% ly_density(x = hwy, data = mpg)
折れ線グラフ
df <- data.frame(x = c(1, 2, 3), y = c(4, 2, 5)) figure() %>% ly_lines(x = x, y = y, data = df)

地図
地図は {maps} パッケージのデータを利用してプロットができる。
library(maps) figure() %>% ly_map("state", color = "gray")

また、 Google Maps を利用することもできる。
gmap(lat = 35.684, lng = 139.753, zoom = 15, map_type = 'terrain')
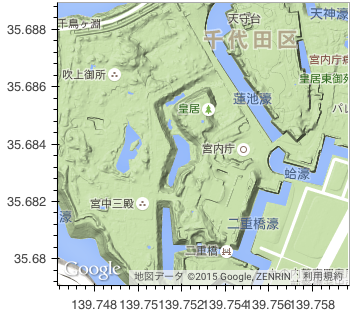
現在 (簡単には) できないもの
R でよく使う種類のプロットを優先しているせいか、以下のような基本的なプロットにはまだ対応していないようだ。
| 種類 | GitHub Issue | 備考 |
|---|---|---|
| 棒グラフ | #8 | 頑張れば ly_hist で描ける。 |
| エリアプロット | #10 | |
| ヒートマップ | #86 | ly_hexbin はある。ly_image で近いことはできるが、軸のラベル付けが手間。 |
| 円/ドーナツ |
まとめ
{rbokeh}、上に挙げた基本的なプロットが使えるようになれば実用できそうな感じだ。Bokeh.js 側には主要な可視化が揃っているため、他のパッケージと比べると利用範囲が広いこと、見た目を複数言語で揃えられるのがメリットかなと思う。
R で 状態空間モデル: {dlm} の最尤推定を可視化する
{dlm} において、状態空間モデルが最尤推定される過程がみたい。以下内容の補足的なエントリ。
「状態空間時系列分析入門」では 引き続き 第8章 に相当。

- 作者: J.J.F.コマンダー,S.J.クープマン,Jacques J.F. Commandeur,Sime Jan Koopman,和合肇
- 出版社/メーカー: シーエーピー出版
- 発売日: 2008/09
- メディア: 単行本
- 購入: 2人 クリック: 4回
- この商品を含むブログを見る
データの準備
データは著者のサポートサイト、An Introduction to State Space Time Series Analysis から入手できる。
library(dlm) # install_github('sinhrks/ggfortify') library(ggfortify) ukdrivers <- read.table('UKdriversKSI.txt', skip = 1) ukdrivers <- ts(ukdrivers[[1]], start = c(1969, 1), frequency = 12) ukdrivers <- log(ukdrivers) y <- ukdrivers
モデルの作成と状態推定値のプロット
サンプルとして、第3章3節 確率的レベルと確定的傾きをもつローカル線形トレンドモデル を作成する。
parm <- log(c(var(y), 0.001)) buildFun <- function(parm){ dlmModPoly(order = 2, dV = exp(parm[1]), dW = c(exp(parm[2]), 0)) } dlmFit <- dlmMLE(y, parm = parm, build = buildFun) # カルマンフィルタ dlmFiltered <- dlmFilter(y, buildFun(dlmFit$par)) # 平滑化 dlmSmoothed <- dlmSmooth(y, buildFun(dlmFit$par))
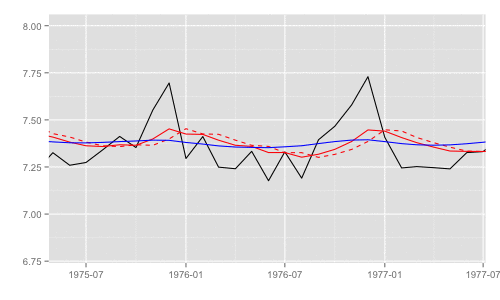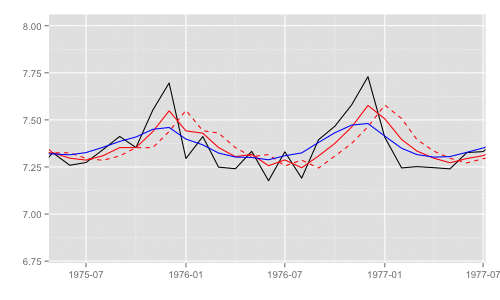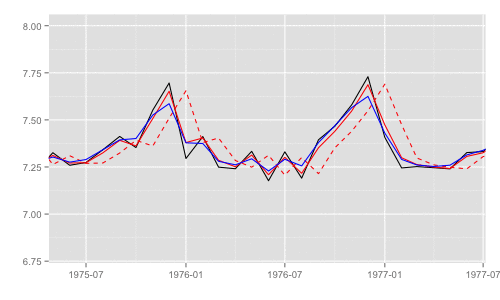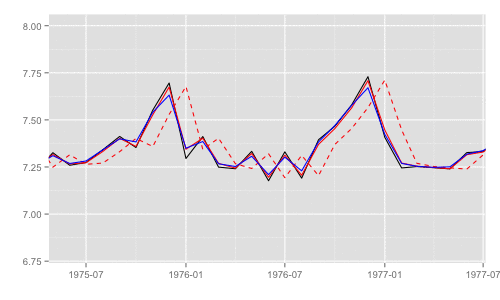
第8章ではモデルの推定値として 3 つの値が出てくるため、その差異がわかるようにプロットする。用語は状態空間時系列分析入門のものを利用。
p <- autoplot(y) p <- autoplot(dlmFiltered$a[, 1], colour = 'red', linetype = 'dashed', p = p) p <- autoplot(dlmFiltered$m[, 1], colour = 'red', p = p) p <- autoplot(dlmSmoothed, colour = 'blue', p = p) p + ylim(c(6.8, 8))
- 黒実線 (
y): 観測時系列 - 赤破線 (
dlmFiltered$a): カルマンフィルタの1期先予測 (予測ステップでの予測値) - 赤実線 (
dlmFiltered$m): カルマンフィルタのフィルタ化状態 (更新ステップでの予測値) - 青実線 (
dlmSmoothed): 平滑化状態
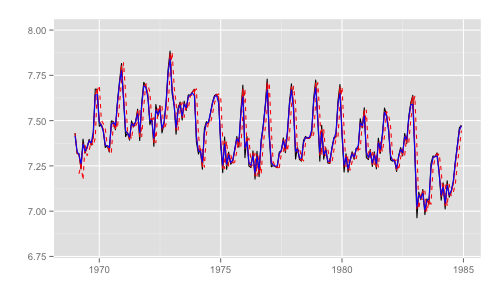
一部期間を拡大するとそれぞれの差異がわかる。これらのうち、最尤推定時の尤度計算式中で直接 利用されるのは "1期先予測" 時の予測誤差 (赤破線) とその標準偏差。
p + xlim(as.Date('1975', '%Y'), as.Date('1977', '%Y')) + ylim(c(6.8, 8))
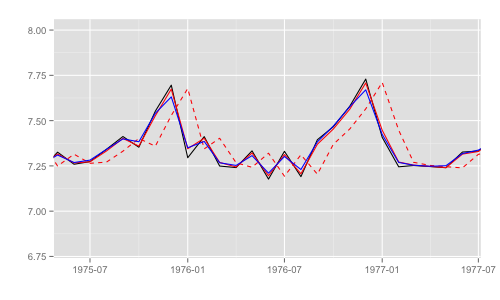
補足 上のモデルでは FF = [1, 0] のため、観測値の推定値 == 状態推定値。
dlmFiltered$mod$FF # [,1] [,2] # [1,] 1 0 all((dlmFiltered$a %*% t(dlmFiltered$mod$FF)) == dlmFiltered$a[, 1]) # [1] TRUE
補足 カルマンフィルタの動きについては以前こんなエントリを書いた。2つ目のエントリにカルマンフィルタの予測 / フィルタ化状態がどういった感じで動いているか記載がある (平滑化は記載ない)。
予測誤差と予測誤差分散
最尤推定の尤度計算式は先のエントリに記載。計算に必要な (1期先) 予測誤差と予測誤差標準偏差は dlm:::residuals.dlmFiltered で求めることができる。
# 既定では標準化されるため、 type = 'raw' を指定して実値をとる resid <- residuals(dlmFiltered, type = 'raw') # 予測誤差 resid$res[1:12] # [1] 7.430707083 -3.827523556 0.111504168 -0.021616238 0.225665269 -0.044983932 # [7] 0.045295381 0.061233329 -0.021088564 0.049603994 0.270853650 0.007138991 # 予測誤差標準偏差 resid$sd[1:12] # [1] 4472.1359566 2236.0679824 0.1922632 0.1596384 0.1484746 0.1429560 # [7] 0.1396797 0.1375109 0.1359691 0.1348166 0.1339224 0.1332084
ふつうに計算して求めるなら、dlm::dlmFiltered インスタンスの各プロパティを利用して、
# 予測誤差 (y - (matrix(as.numeric(dlmFiltered$a), 192, 2) %*% t(dlmFiltered$mod$FF)))[1:12] # [1] 7.430707083 -3.827523556 0.111504168 -0.021616238 0.225665269 -0.044983932 # [7] 0.045295381 0.061233329 -0.021088564 0.049603994 0.270853650 0.007138991 # もしくは (y - dlmFiltered$f)[1:12] # [1] 7.430707083 -3.827523556 0.111504168 -0.021616238 0.225665269 -0.044983932 # [7] 0.045295381 0.061233329 -0.021088564 0.049603994 0.270853650 0.007138991 # 予測誤差標準偏差 f <- function(i) { FF <- dlmFiltered$mod$FF V <- dlmFiltered$mod$V diag(crossprod(dlmFiltered$D.R[i, ] * t(FF %*% dlmFiltered$U.R[[i]])) + V) } drop(t(sqrt(sapply(seq(along = dlmFiltered$U.R), f))))[1:12] # [1] 4472.1359566 2236.0679824 0.1922632 0.1596384 0.1484746 0.1429560 # [7] 0.1396797 0.1375109 0.1359691 0.1348166 0.1339224 0.1332084
最尤推定の可視化
最尤推定時のパラメータの変化を記録するため、dlm::dlmMLE を書き換えた関数を定義する。LL_ckbookcompat はテキスト記載の対数尤度 (に近いもの) を計算するための関数 ( 詳細はこちら )。
# optim 1 ステップごとに作成されたモデルを保存 models <<- list() dlmMLE_wrapper <- function (y, parm, build, method = "L-BFGS-B", ..., debug = FALSE) { logLik <- function(parm, ...) { mod <- build(parm, ...) # グローバル変数に追加 models[[length(models) + 1]] <<- mod return(dlmLL(y = y, mod = mod, debug = debug)) } return(optim(parm, logLik, method = method, ...)) } # 推定するパラメータ数が 2 のため、総和をとる範囲を変更 LL_ckbookcompat <- function(y, mod) { n <- length(y) filtered <- dlmFilter(y, mod) resid <- residuals_dlmcompat(filtered) F <- (as.numeric(resid$sd)^2)[3:n] v <- as.numeric(resid$res)[3:n] return(-0.5 * (n * log(2 * pi) + sum(log(F) + (v^2/F)))) }
続けて、アニメーション用の処理を定義して実行。
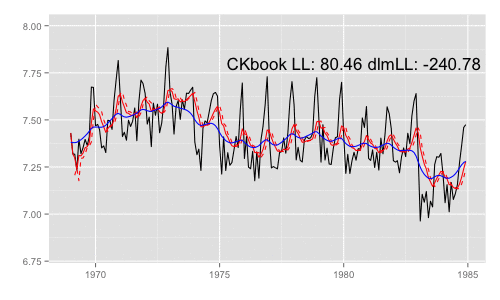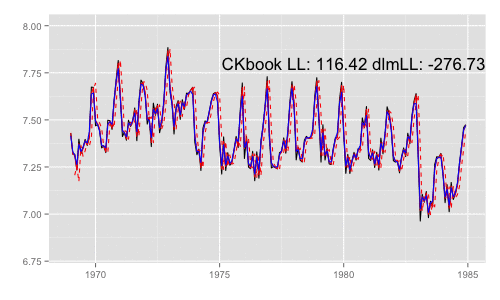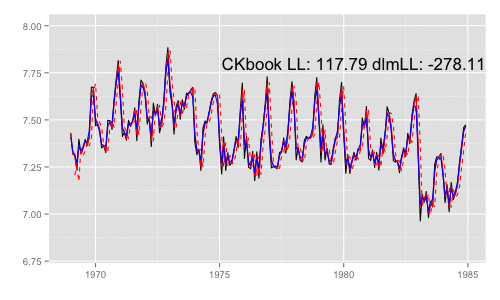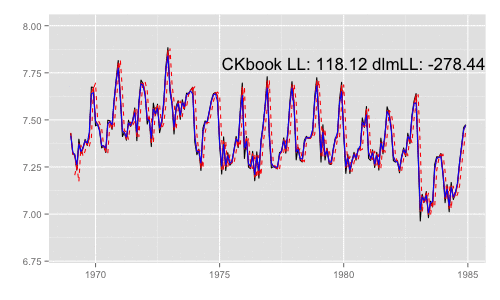
library(animation) models <<- list() dlmMLE_wrapper(y, parm = parm, build = buildFun) saveGIF({ for (mod in models[1:65]) { filtered <- dlmFilter(y, mod) p <- autoplot(filtered, fitted.colour = 'red', fitted.linetype = 'dashed') p <- autoplot(dlmSmooth(y, mod), colour = 'blue', p = p) label <- paste('CKbook LL:', round(LL_ckbookcompat(y, mod), 2), 'dlmLL:', round(dlmLL(y, mod), 2)) p <- p + annotate('text', label = label, x = as.Date('1980', '%Y'), y = 7.8, size = 6) print(p) Sys.sleep(0.01) } }, interval = 0.3, movie.name = "dlm.gif", ani.width = 500, ani.height = 300)
最尤推定時に各状態推定値がどのように変化していくかをみる。左側 "CKbookLL" はテキスト記載の対数尤度計算式 LL_ckbookcompat の値。右側 "dlmLL" は dlm::dlmLL の値 ( optim で最適化される値 )。
テキストに記載された数値より、収束時には左側の値が 0.6247935 x 観測時系列の長さ (192) = 119.9604 程度になるはずだ。
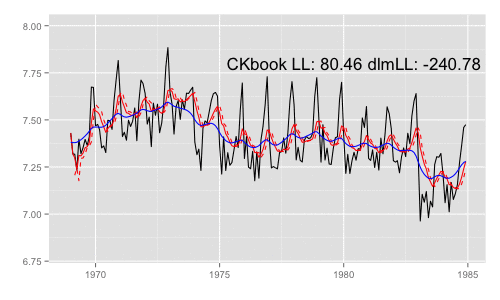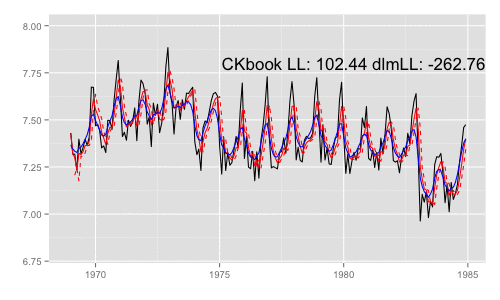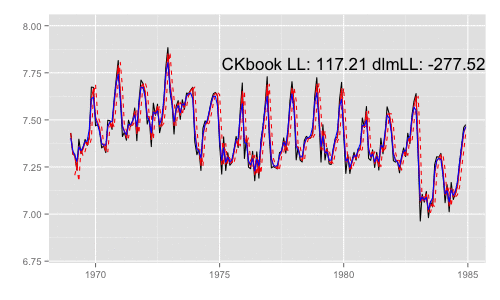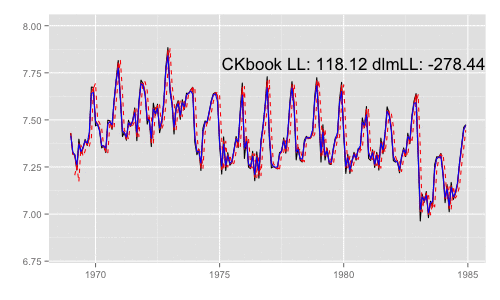
補足 {dlm} は既定では optim の際に method = 'L-BFGS-B' を利用する。可視化目的であれば Nelder-Mead 法なんかを使ったほうが 素人目には "最適化らしい" 動きになる気がする。
models <<- list() dlmMLE_wrapper(y, parm = parm, build = buildFun, method = 'Nelder-Mead') # 以降略

R で 状態空間モデル: 状態空間時系列分析入門を {rstan} で再現したい
前の記事でもリンクさせていただいているが、サイト 「状態空間時系列分析入門」をRで再現する では以下のテキストを {dlm}, {KFAS} で再現されており非常にありがたい。これらのパッケージの使い方については リンク先を読めば困らない感じだ。
自分も勉強のために似たことやりたい、、でも同じことやるのもなあ、、と考えた結果 同テキストの内容 {rstan} を使ってやってみた。
補足 Stan には状態空間表現用の関数 gaussian_dlm_obs ( 利用例 ) があるのだが、自分は使ったことがない。7章までのモデルは全て漸化式で表現されているため、それらを Stan のモデルとして記述した。

- 作者: J.J.F.コマンダー,S.J.クープマン,Jacques J.F. Commandeur,Sime Jan Koopman,和合肇
- 出版社/メーカー: シーエーピー出版
- 発売日: 2008/09
- メディア: 単行本
- 購入: 2人 クリック: 4回
- この商品を含むブログを見る
github リポジトリ
各スクリプト / モデルは GitHub にあげた。現在 7 章までのモデルと kick する R スクリプト, markdown ファイルを置いている。
モデルの一覧
各章のモデルについて、その実行結果を RPubs にあげている。各モデルの概要とリンクを以下に記載する。自分のモデリング能力不足のため いくつか課題があると考えており、特に怪しいものには * をつけた。
- 第1章 はじめに
- 第2章 ローカル・レベル・モデル
- 第3章 ローカル線形トレンド・モデル
- 第4章 季節要素のあるローカル・レベル・モデル
- 第5章 説明変数のあるローカル・レベル・モデル
- 第6章 干渉変数のあるローカル・レベル・モデル
- 第7章 英国シートベルト法とインフレーション・モデル
かんたんな説明
必要パッケージ
まず、実行には以下のCRAN非公開パッケージが必要である。後者はどうでもよいが {pforeach} はいろいろ捗るのでインストールしていない方はただちに入れるべきだ。
hoxo-m/pforeach:Stan並列化に利用。sinhrks/ggfortify: 結果のプロットに利用。
データの読み込み
データは著者のサポートサイト、An Introduction to State Space Time Series Analysis から入手できる。各データは適宜 ts インスタンス化しておく。
Stan の実行
Stan の呼び出しは全て以下のように並列化して行う。
model_file <- 'モデルを記述したファイルのパス' # モデルのコンパイルを実行 stan_fit <- stan(file = model_file, chains = 0) # Stan を並列実行する。実行結果に sflist2stanfit を適用し、stanfit インスタンスを得る fit <- pforeach(i = 1:4, .final = sflist2stanfit)({ stan(fit = stan_fit, data = standata, chains = 1, seed = i) })
また、モデルが収束したかどうかは 全ての Rhat が 1.1 未満となっているかどうかで確認する。そのままでは収束しない / しにくいモデルには、初期値 / 事前分布 / 制約を入れて収束させた。
is.converged <- function(stanfit) { summarized <- summary(stanfit) all(summarized$summary[, 'Rhat'] < 1.1) } stopifnot(is.converged(fit))
また、テキストに最尤推定の結果が記載されている場合は、以下のようにして近い値になっているかを確認した。値がずれる場合は stopifnot を外し、差異を出力する。
# is.almost.fitted の定義は各リンク先を参照 stopifnot(is.almost.fitted(mu[[1]], 7.4157))
結果のプロット
テキストのグラフを再現する形で行う。使っているパッケージの説明はこちら。
- RPubs - Plotting Time Series with ggplot2 and ggfortify
- RPubs - Plotting State Space Time Series with ggplot2 and ggfortify
課題
以下の2つについてはもう少し Stan に習熟できたら直したい。
複数の確率的状態をもつモデルをうまく収束させたい
このようなモデルは 収束しにくく、また 最尤推定した状態推定値 ( テキスト、{dlm}、{KFAS}の結果 ) とずれやすい。特に 現状 制約を入れて収束させているモデルについて、事前分布 / 初期値 / 尤度関数の範囲でなんとかしたい。
モデルの一覧で * をつけたものがこれにあたる。
テキストの尤度関数を再現したい
テキストに記載の式 (概要は以下記事参照) に揃えようとすると、自分の記述では収束しにくくなってしまう。
R で 状態空間モデル: {dlm} で単変量モデルの状態空間表現を利用する
こちらの続きで、状態空間時系列分析入門の第8章の内容。

- 作者: J.J.F.コマンダー,S.J.クープマン,Jacques J.F. Commandeur,Sime Jan Koopman,和合肇
- 出版社/メーカー: シーエーピー出版
- 発売日: 2008/09
- メディア: 単行本
- 購入: 2人 クリック: 4回
- この商品を含むブログを見る
状態空間モデルでは、トレンド、季節効果、説明変数効果などさまざまな要因を考慮することができる。また これらの要因を単一の状態空間表現で統一的に扱える。時刻 における観測値
と状態
は以下の式で書ける (前回と同じ)。
この式であらわされたモデルを {dlm} で利用する際にどうすればよいのかをまとめたい。
vignette にあるとおり、{dlm} ではモデルを dlm::dlmModPoly などのモデル記述関数の組み合わせで定義できる。これらの関数は便利だが、上の式との関係は外側からはよくわからない。
上式から {dlm} のモデルを作成する際には dlm::dlm を直接使うのがよさそうだ。dlm::dlm の引数は上式にほぼ対応しているため、式表現されたモデルをそのまま実装できる。また、モデル記述関数も内部的には dlm::dlm を使っているため、これら関数の理解にも役立つ。dlm::dlm の引数と上式の対応は前回書いたとおり。
FF:に対応。
V:に対応。
GG:に対応。
W:に対応。
m0:の事前分布の平均。
C0:の事前分布の分散。
J...: 各パラメータが時間変化するとき利用。
参考 サイト 「状態空間時系列分析入門」をRで再現する はこのテキストを {dlm}, {KFAS} で再現している。こちらは dlm::dlm ではなく dlm::dlmModPoly などの関数を使ってモデル作成している。
準備
まずデータを読み込む。データは著者のサポートサイト、An Introduction to State Space Time Series Analysis から入手できる。
library(dlm) # install_github('sinhrks/ggfortify') library(ggfortify) ukdrivers <- read.table('UKdriversKSI.txt', skip = 1) ukdrivers <- ts(ukdrivers[[1]], start = c(1969, 1), frequency = 12) ukdrivers <- log(ukdrivers)
以降、テキストに記載のモデル順に dlm::dlm から利用する場合のプログラムを列挙する。値 / 推定するパラメータを変更したい場合は buildObj 内の対応する要素を適当に修正すればよい。
1. ローカルレベルモデル (確定的レベル)
それぞれのモデルについて 状態 として何が考慮されているかを書いておく。
:
は確定的レベル (経時変化しない)。
補足 それぞれのモデルの式表現を書こうとしたのだが、はてなマークダウン記法だと TeX 記法中 の "&" がうまく処理できないようなので諦めた。詳細はテキストと見比べてください。
y <- ukdrivers parm<- log(var(y)) buildObj <- function(parm) { dlm(FF = matrix(1, 1, 1), V = matrix(exp(parm), 1, 1), GG = matrix(1, 1, 1), W = matrix(0, 1, 1), m0 = 0, C0 = matrix(1e+07, 1, 1)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] # [1,] 1 # # $V # [,1] # [1,] 0.02935256 # # $GG # [,1] # [1,] 1 # # $W # [,1] # [1,] 0 # # $m0 # [1] 0 # # $C0 # [,1] # [1,] 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

赤破線がカルマンフィルタ、青破線が平滑化した値。以降もプロットするが、確率的状態を使うと 実データにほぼフィットするため、グラフからでは差がよくわからない。
2. ローカル線形トレンドモデル (確率的レベルと確率的傾き)
:
は確率的レベル (経時変化する)、
は確率的傾き(経時変化する)。
parm <- log(c(var(y), 0.001, 0.001)) buildObj <- function(parm) { dlm(FF = matrix(c(1, 0), 1, 2), V = matrix(exp(parm[1]), 1, 1), GG = matrix(c(1, 1, 0, 1), 2, 2, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, exp(parm[3])), 2, 2, byrow = TRUE), m0 = c(0, 0), C0 = matrix(c(1e+07, 0, 0, 1e+07), 2, 2, byrow = TRUE)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] # [1,] 1 0 # # $V # [,1] # [1,] 0.002118549 # # $GG # [,1] [,2] # [1,] 1 1 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.01212741 0.00000e+00 # [2,] 0.00000000 1.92431e-10 # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

3. 季節要素のあるローカルレベルモデル (確率的レベルと確率的季節)
:
は確率的レベル、
は 四半期単位の確率的季節のダミー項。
ukinflation <- read.table('UKinflation.txt', skip = 1) ukinflation <- ts(ukinflation[[1]], start = c(1950, 1), frequency = 4) y <- ukinflation parm <- log(c(var(y), 0.00001, 0.00001)) buildObj <- function(parm) { dlm(FF = matrix(c(1, 1, 0, 0), 1, 4), V = matrix(c(exp(parm[1])), 1, 1), GG = matrix(c(1, 0, 0, 0, 0, -1, -1, -1, 0, 1, 0, 0, 0, 0, 1, 0), 4, 4, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0, 0, exp(parm[3]), 0, 0, 0, 0, 0, 0, 0, 0, 0, 0), 4, 4, byrow = TRUE), m0 = c(0, 0, 0, 0), C0 = matrix(c(1e+07, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 1e+07), 4, 4, byrow = TRUE)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] [,3] [,4] # [1,] 1 1 0 0 # # $V # [,1] # [1,] 3.37127e-05 # # $GG # [,1] [,2] [,3] [,4] # [1,] 1 0 0 0 # [2,] 0 -1 -1 -1 # [3,] 0 1 0 0 # [4,] 0 0 1 0 # # $W # [,1] [,2] [,3] [,4] # [1,] 2.124158e-05 0.000000e+00 0 0 # [2,] 0.000000e+00 4.345176e-07 0 0 # [3,] 0.000000e+00 0.000000e+00 0 0 # [4,] 0.000000e+00 0.000000e+00 0 0 # # $m0 # [1] 0 0 0 0 # # $C0 # [,1] [,2] [,3] [,4] # [1,] 1e+07 0e+00 0e+00 0e+00 # [2,] 0e+00 1e+07 0e+00 0e+00 # [3,] 0e+00 0e+00 1e+07 0e+00 # [4,] 0e+00 0e+00 0e+00 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

4. 説明変数のあるローカルレベルモデル (確率的レベル)
:
は確率的レベル、
は説明変数の係数。
説明変数は dlm::dlm へ引数 X として渡す。
ukpetrol <- read.table('logUKpetrolprice.txt', skip = 1) ukpetrol <- ts(ukpetrol, start = start(ukdrivers), frequency = frequency(ukdrivers)) y <- ukdrivers x <- ukpetrol parm <- c(var(y), 0.001) buildObj <- function(parm) { dlm(FF = matrix(c(1, 0), 1, 2), V = matrix(exp(parm[1]), 1, 1), GG = matrix(c(1, 0, 0, 1), 2, 2, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0), 2, 2, byrow = TRUE), m0 = c(0, 0), C0 = matrix(c(1e+07, 0, 0, 1e+07), 2, 2, byrow = TRUE), JFF = matrix(c(0, 1), 1, 2), X = as.matrix(x)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] # [1,] 1 1 # # $V # [,1] # [1,] 0.002347965 # # $GG # [,1] [,2] # [1,] 1 0 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.01166743 0 # [2,] 0.00000000 0 # # $JFF # [,1] [,2] # [1,] 0 1 # # $X # V1 # [1,] -2.273 # [2,] -2.279 # [3,] ... # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07 filtered <- dlmFilter(y, dlmModObj)$m smoothed <- dlmSmooth(y, dlmModObj)$s p <- autoplot(y) p <- autoplot(filtered[, 1] + filtered[, 2] * x, colour = 'red', linetype = 'dashed', p = p) autoplot(smoothed[, 1] + smoothed[, 2] * x, colour = 'blue', p = p)

5. 干渉変数のあるローカルレベルモデル (確率的レベル)
:
は確率的レベル、
は干渉変数の係数。
変数名は異なるが、式は説明変数のあるローカルレベルモデルと同一。
ukseats <- c(rep(0, (1982 - 1968) * 12 + 1), rep(1, (1984 - 1982) * 12 - 1)) ukseats <- ts(ukseats, start = start(ukdrivers), frequency = frequency(ukdrivers)) x <- ukseats parm <- c(var(y), 0.001) buildObj <- function(parm) { dlm(FF = matrix(c(1, 0), 1, 2), V = matrix(exp(parm[1]), 1, 1), GG = matrix(c(1, 0, 0, 1), 2, 2, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0), 2, 2, byrow = TRUE), m0 = c(0, 0), C0 = matrix(c(1e+07, 0, 0, 1e+07), 2, 2, byrow = TRUE), JFF = matrix(c(0, 1), 1, 2), X = as.matrix(x)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] # [1,] 1 0 # # $V # [,1] # [1,] 0.002692686 # # $GG # [,1] [,2] # [1,] 1 0 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.01041175 0 # [2,] 0.00000000 0 # # $JFF # [,1] [,2] # [1,] 0 1 # # $X # [,1] # [1,] 0 # [2,] 0 # [3,] ... # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07 filtered <- dlmFilter(y, dlmModObj)$m smoothed <- dlmSmooth(y, dlmModObj)$s p <- autoplot(y) p <- autoplot(filtered[, 1] + filtered[, 2] * x, colour = 'red', linetype = 'dashed', p = p) autoplot(smoothed[, 1] + smoothed[, 2] * x, colour = 'blue', p = p)

6. 季節要素と干渉変数のあるローカルレベルモデル (確率的レベルと確率的季節)
:
は確率的レベル、
は確率的季節、
は干渉変数の係数。
補足 テキスト記載の数式は3項の季節要素、説明変数、干渉変数を含む 6 状態が記載されている。季節要素が3項のため英国インフレーションモデルについて記載していると思われるが、このモデルには説明変数がないため状態数があわない。そのため、テキスト中の式を英国インフレーションモデルに合うよう書き換えている。
y <- ukinflation ukpulse <- rep(0, length.out = length(ukinflation)) ukpulse[4*(1975-1950)+2] <- 1 ukpulse[4*(1979-1950)+3] <- 1 ukpulse <- ts(ukpulse, start = start(ukinflation), frequency = frequency(ukinflation)) x <- ukpulse parm <- log(c(var(y), 0.00001, 0.00001)) buildObj <- function(parm) { dlm(FF = matrix(c(1, 1, 0, 0, 1), 1, 5), V = matrix(c(exp(parm[1])), 1, 1), GG = matrix(c(1, 0, 0, 0, 0, 0, -1, -1, -1, 0, 0, 1, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1), 5, 5, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0, 0, 0, exp(parm[3]), 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0), 5, 5, byrow = TRUE), m0 = c(0, 0, 0, 0, 0), C0 = matrix(c(1e+07, 0, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 0, 1e+07), 5, 5, byrow = TRUE), JFF = matrix(c(0, 0, 0, 0, 1), 1, 5), X = as.matrix(x)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] [,3] [,4] [,5] # [1,] 1 1 0 0 1 # # $V # [,1] # [1,] 2.27569e-05 # # $GG # [,1] [,2] [,3] [,4] [,5] # [1,] 1 0 0 0 0 # [2,] 0 -1 -1 -1 0 # [3,] 0 1 0 0 0 # [4,] 0 0 1 0 0 # [5,] 0 0 0 0 1 # # $W # [,1] [,2] [,3] [,4] [,5] # [1,] 1.783797e-05 0.000000e+00 0 0 0 # [2,] 0.000000e+00 4.310206e-07 0 0 0 # [3,] 0.000000e+00 0.000000e+00 0 0 0 # [4,] 0.000000e+00 0.000000e+00 0 0 0 # [5,] 0.000000e+00 0.000000e+00 0 0 0 # # $JFF # [,1] [,2] [,3] [,4] [,5] # [1,] 0 0 0 0 1 # # $X # [,1] # [1,] 0 # [2,] 0 # [3,] ... # # $m0 # [1] 0 0 0 0 0 # # $C0 # [,1] [,2] [,3] [,4] [,5] # [1,] 1e+07 0e+00 0e+00 0e+00 0e+00 # [2,] 0e+00 1e+07 0e+00 0e+00 0e+00 # [3,] 0e+00 0e+00 1e+07 0e+00 0e+00 # [4,] 0e+00 0e+00 0e+00 1e+07 0e+00 # [5,] 0e+00 0e+00 0e+00 0e+00 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

モデルの作り方
とはいえ、最初から上式のような形でモデルが定義できていることはないと思う。まずは簡単なモデルを dlm::dlmModPoly などで作って、それをベースに書き換えていくのが良さそう。作ったモデルに適当なパラメータを与えて初期化すれば、モデルの次元 / 中身を確認できる。
y <- ukdrivers parm<- log(c(var(y), 0.001)) buildFun <- function(parm){ dlmModPoly(order = 2, dV = exp(parm[1]), dW = c(exp(parm[2]), 0)) } buildFun(parm) # $FF # [,1] [,2] # [1,] 1 0 # # $V # [,1] # [1,] 0.02935256 # # $GG # [,1] [,2] # [1,] 1 1 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.001 0 # [2,] 0.000 0 # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07
R で 状態空間モデル: {dlm} の対数尤度計算について
状態空間モデルを扱う R パッケージである {dlm} では、モデルのパラメータ推定を最尤法で行なう。その際に使われる対数尤度の計算式はパッケージによって違いがあるようで、推定結果が自分の参照しているテキストとずれる。その違いを確かめたい。
当該のテキストはこちら。今回はこれを持っている方向けの内容。

- 作者: J.J.F.コマンダー,S.J.クープマン,Jacques J.F. Commandeur,Sime Jan Koopman,和合肇
- 出版社/メーカー: シーエーピー出版
- 発売日: 2008/09
- メディア: 単行本
- 購入: 2人 クリック: 4回
- この商品を含むブログを見る
参考 サイト 「状態空間時系列分析入門」をRで再現する はこのテキストを {dlm}, {KFAS} で再現しており、対数尤度の差異についても記載がある。
テキストでは単変量状態空間モデルの 時刻 における観測値
と状態
を以下の式であらわしている。
1期先の状態を更新するカルマンフィルタは、
: カルマンゲイン
: 状態推定誤差分散
: カルマンフィルタでの1期先予測誤差分散
これらを用いて、対数尤度関数は式 (8.7) で以下のように定義されている。
: 観測時系列の長さ
: 推定する状態数
: カルマンフィルタの1期先予測誤差
この式で計算される対数尤度は {dlm} のものと異なる。差異がある理由として記載する 2 点がわかったが、これらを解消してもまだ結果はあわない。
1. テキストの記載による差異
まず、テキストの各モデルに対数尤度として記載されている数値は 上式の計算結果そのものでない。P162 に添付されているプログラムに 対数尤度を系列の長さ で割る処理がしれっと入っており、処理後の値が対数尤度として記載されている。そのため、対数尤度に戻すためには各モデルに記載されている数値に 系列の長さ
をかける必要がある。
例えば 2.1 ローカルレベルモデル (確定的レベルモデル) に対数尤度として記載されている数値 0.3297597 に、系列の長さ 192 をかけた 63.31386 が 式 (8.7) の計算結果にあたる。
2. {dlm}の対数尤度計算式との差異
テキストで利用しているデータ利用して、{dlm} との差異を確かめたい。まずデータを読み込む。データは著者のサポートサイト、An Introduction to State Space Time Series Analysis から入手できる。
ukdrivers <- read.table('UKdriversKSI.txt', skip = 1) ukdrivers <- ts(ukdrivers[[1]], start = c(1969, 1), frequency = 12) ukdrivers <- log(ukdrivers) y <- ukdrivers
パラメータを受け取ってローカルレベルモデルを返す関数 buildFun を定義し、dlm::dlmMLE で最尤推定する。結果は リストになり、推定されたパラメータ par や 対数尤度 (を代替するもの/後述) value がプロパティとして含まれている。
library(dlm) parm <- c(var(y)) buildFun <- function(x) { dlmModPoly(order = 1, dV = parm, dW = 0) } dlmFit <- dlmMLE(y, parm = parm, build = buildFun) class(dlmFit) # [1] "list" names(dlmFit) # [1] "par" "value" "counts" "convergence" "message" dlmFit$par # [1] 0.02935256
続けて、最尤推定したパラメータ dlmFit$par を使ってモデルを作成する。プロパティの意味は vignette の記載がわかりやすい。テキストと式/添字をあわせると
FF:に対応。
V:に対応。
GG:に対応。
W:に対応。
m0:の事前分布の平均。
C0:の事前分布の分散。
J...: 各パラメータが時間変化するとき利用。
dlmMod <- buildFun(dlmFit$par) class(dlmMod) # [1] "dlm" names(dlmMod) # [1] "m0" "C0" "FF" "V" "GG" "W" "JFF" "JV" "JGG" "JW" V(dlmMod) # [,1] # [1,] 0.02935256
ここで、{dlm} の内部で最尤推定がどのように行なわれているのか確かめたい。
dlm::dlmMLE の定義をみると、最尤推定は dlm::dlmLL を optim で最適化することで行っている。
dlmMLE # function (y, parm, build, method = "L-BFGS-B", ..., debug = FALSE) # { # logLik <- function(parm, ...) { # mod <- build(parm, ...) # return(dlmLL(y = y, mod = mod, debug = debug)) # } # out <- optim(parm, logLik, method = method, ...) # return(out) # } # <environment: namespace:dlm>
dlm::dlmLL はその名前から対数尤度のようにも見えるが、対数尤度とは符号が逆 ( optim で最適化するため、尤もらしいほど値が小さくなる )。
dlmLL(y = y, mod = buildFun(dlmFit$par)) # [1] -230.7721
補足 パラメータ推定時の dlm::dlmLL の値は dlm::dlmMLE の結果にも含まれている。
dlmFit$value # [1] -230.7721
dlm::dlmLL の処理は少し条件分岐が多いため省略するが、上のモデルについて必要な処理は以下のようなものであることがわかる。カルマンフィルタ dlm::dlmFilter を適用した結果から予測誤差、予測誤差標準偏差を求めて 対数尤度の代替となる値を算出している。
# 予測誤差、予測誤差標準偏差を計算 residuals_dlmcompat <- function (object) { mod <- object$mod # yは観測値 (入力), a は状態の予測値 res <- drop(object$y - object$a %*% t(mod$FF)) f <- function(i) { # 予測誤差標準偏差 diag(crossprod(object$D.R[i, ] * t(mod$FF %*% object$U.R[[i]])) + mod$V) } SD <- drop(t(sqrt(sapply(seq(along = object$U.R), f)))) return(list(res = res, sd = SD)) } # dlmLLと同じ値を計算 LL_dlmcompat <- function(y, mod) { filtered <- dlmFilter(y, mod) resid <- residuals_dlmcompat(filtered) # 予測誤差分散 F <- as.numeric(resid$sd)^2 v <- as.numeric(resid$res) return(0.5 * sum(log(F) + v^2/F)) } LL_dlmcompat(y = y, mod = buildFun(dlmFit$par)) # [1] -230.7721
これを上式 (8.7) と比べると以下の差異がある。
- 符号が逆 (
optimでの処理のため ) - 第1項目がない ( パラメータ非依存のため
optimには影響ない ) - 第2項目の総和の範囲が異なる
これをテキストに準じて書き換えると、
LL_ckbookcompat <- function(y, mod) { n <- length(y) filtered <- dlmFilter(y, mod) resid <- residuals_dlmcompat(filtered) F <- (as.numeric(resid$sd)^2)[2:n] v <- as.numeric(resid$res)[2:n] return(-0.5 * (n * log(2 * pi) + sum(log(F) + v^2/F))) } LL_ckbookcompat(y = y, mod = buildFun(dlmFit$par)) # [1] 62.39492
となり、テキストの対数尤度 63.31386 に結構 近づいた (が、あわない、、)。対数尤度の計算結果はパッケージによって異なる、ということは覚えておいたほうがよさそうだ。
補足: このモデルにおいては、 {KFAS} の結果も上記修正後の結果と一致している。
library(KFAS) kfasMod <- SSModel(y ~ SSMtrend(degree=1, Q=list(matrix(0))), H = matrix(NA)) kfasFit <- fitSSM(kfasMod, inits = parm, method = "BFGS") kfasFit$optim.out$value # [1] -62.39492 KFS(kfasFit$model)$logLik # [1] 62.39492