R {ggplot2} で独自の geom を手軽に作りたい
重要 このエントリは {ggplot2} 1.1.0 以前の情報です。v2.0.0 以降の方法は vignettes "Extending ggplot2" を読んでください。
はじめに
{ggplot2} を使っていると、新しい描画図形 (geom) を作りたいなという場合がたまにある。その方法は {ggplot2} の Wiki に書いてあり、手順は、
- 描画図形の実体を
grid::Grobとして定義する (リンク先の例ではfieldGrob) - 定義した
Grobを 呼び出す描画用クラスをggplot2:::Geomを継承して作る (リンク先の例ではGeomField) - 描画用関数
geom_xxxを作る (リンク先の例ではgeom_field)
手順のうち 新しい Grob を作るのは少し面倒な感じだ。が、作りたい geom が 既存 {ggplot2} 関数へのちょっとした追加処理や 組み合わせで表現できる場合には新しい Grob を作る必要もなく、わりと手軽にできる。ここではその方法を書く。
やりたいこと
ggplot2::geom_ribbon を階段状に描画したい。背景はこちら。
@berobero11 そのステキ関数の存在を知りませんでした。信頼区間の塗りつぶしが一手間必要そうなので、少し時間ください。
— sinhrks (@sinhrks) 2015, 3月 241. 既存の描画用クラス (Geom) を継承して作る
ribbon を階段状に描画するには、geom_ribbon の描画直前に データを階段状に変換する必要がある。他は geom_ribbon 同様に動けばよいので、描画用のクラスは ggplot2:::Geom ではなく ggplot2:::GeomRibbon を継承してつくればよさそうだ。
補足 データを階段状に変更する処理は ggplot2 の geom-path-step.R のものを多少変更して作成。
library(ggplot2) library(proto) # 描画用クラスを定義 GeomConfint <- proto(ggplot2:::GeomRibbon, { objname <- "confint1" required_aes <- c("x", "ymin", "ymax") draw <- function(., data, scales, coordinates, na.rm = FALSE, ...) { if (na.rm) data <- data[complete.cases(data[required_aes]), ] data <- data[order(data$group, data$x), ] # ここでデータを階段状に変換 data <- .$stairstep_confint(data) ggplot2:::GeomRibbon$draw(data, scales, coordinates, na.rm = FALSE, ...) } stairstep_confint <- function (., data) { # データを階段状に変換するメソッド data <- as.data.frame(data)[order(data$x), ] n <- nrow(data) ys <- rep(1:n, each = 2)[-2 * n] xs <- c(1, rep(2:n, each = 2)) data.frame(x = data$x[xs], ymin = data$ymin[ys], ymax = data$ymax[ys], data[xs, setdiff(names(data), c("x", "ymin", "ymax"))]) } }) # 描画用クラスを呼び出す描画関数を定義 geom_confint1 <- function (mapping = NULL, data = NULL, stat = "identity", position = "identity", na.rm = FALSE, ...) { GeomConfint$new(mapping = mapping, data = data, stat = stat, position = position, na.rm = na.rm, ...) }
このとき、GeomConfint$draw の data としては、以下のように描画用に変換された data.frame が渡される。そのため、描画用データに対して処理を行う場合はこの方法が便利。
# x ymin ymax PANEL group colour fill size linetype alpha # 1 1 -1 1 1 1 NA grey20 0.5 1 0.5 # 2 2 -2 2 1 1 NA grey20 0.5 1 0.5 # 3 3 -3 3 1 1 NA grey20 0.5 1 0.5 # 4 4 -4 4 1 1 NA grey20 0.5 1 0.5
描画してみる。上で定義した描画関数 geom_confint1 がそのまま geom として使える。
df <- data.frame(x = c(1, 2, 3, 4), upper = c(1, 2, 3, 4), lower = c(-1, -2, -3, -4)) ggplot(data = df) + geom_confint1(aes(x = x, ymin = lower, ymax = upper), alpha = 0.5)

2. 描画用関数 geom_xxx だけを定義して作る
また、今回の場合 塗りつぶしが必要なければ 複数の geom_step の組み合わせでも描ける。このときは描画関数を以下のように定義すればよい。
geom_confint2 <- function (mapping = NULL, data = NULL, stat = "identity", position = "identity", na.rm = FALSE, ...) { # 上側 / 下側の階段関数 geom_step に渡す mapping を作成 mapping1 <- mapping mapping1['y'] <- mapping['ymax'] mapping2 <- mapping mapping2['y'] <- mapping['ymin'] g1 <- geom_step(mapping = mapping1, data = data, stat = stat, position = position, na.rm = na.rm, ...) g2 <- geom_step(mapping = mapping2, data = data, stat = stat, position = position, na.rm = na.rm, ...) # 複数の geom はリストで返す list(g1, g2) }
描画する。
ggplot(data = df) + geom_confint2(aes(x = x, ymin = lower, ymax = upper))

補足 ただし、上の例では mapping の変換を伴うため、以下の書式では動かない。
ggplot(data = df, mapping = aes(x = x, ymin = lower, ymax = upper)) + geom_confint2() # Error: 引数に異なる列数のデータフレームが含まれています: 7, 0
まとめ
独自の geom は正当な方法以外でもわりと手軽に作れる。
- 既存の描画用クラス (
Geom) を継承して作る - 描画用関数
geom_xxxだけを定義して作る
R {R6} で 別クラス同士の演算子を定義したい
先日の Tokyo.R で紹介されていた {R6} パッケージが使いやすそうだったため、自分の パッケージ でも 使ってみるべく試した。
作りたいのは 複数の ggplot インスタンスをまとめて描画するコンテナクラス。{ggplot2} では 種類の異なる 複数のプロットをサブプロットにできないため、自分のパッケージでは以下のようなコンテナを作っている。普通にサブプロット描画したいだけならこんなクラスは必要ないが、{ggplot2} と同じように 加算演算子 + を使って テーマの変更とかしたい。これは以下のような感じで動く。
p1 <- qplot(Petal.Width, Petal.Length, colour = Species, data = iris) p2 <- qplot(Sepal.Width, Petal.Length, colour = Species, data = iris) mp <- new('ggmultiplot', plots = list(p1, p2)) mp + theme_bw()

S4 class で書く
現在は 以下のような S4 クラスでの定義を使っている。パッケージ中では行数/列数指定時のレイアウト調整などもやっているが、今回は関係ないので省略。
library(ggplot2) library(gridExtra) # S4 クラス定義 setClass('ggmultiplot', representation(plots = 'list')) # ggmultiplot に対する加算演算子を定義 setMethod('+', c('ggmultiplot', 'ANY'), function(e1, e2) { # 第二引数を plots の各プロットに順番に適用 plots <- lapply(e1@plots, function(x) { x + e2 }) new('ggmultiplot', plots = plots) }) # print メソッドを定義 # gridExtra::grid.arrange を使って複数のプロットを描画 setMethod('print', 'ggmultiplot', function(x) { nplots = length(x@plots) if (nplots==1) { print(x@plots[[1]]) } else { args <- c(x@plots, list(ncol = 2)) do.call(gridExtra::grid.arrange, args) } }) # show メソッドを定義 setMethod('show', 'ggmultiplot', function(object) { print(object) })
{R6} class で書く
これを {R6} クラスに書き換えるとこんな感じ。クラス定義自体はシンプルになってうれしい。S4 用の setMethod は {R6} に対して使えないため、加算演算子は S3 の総称関数として定義する必要がある。
library(R6) # クラス定義 ggmultiplotR6 <- R6::R6Class('ggmultiplotR6', public = list( plots = list(), initialize = function(plots) { self$plots <- plots }, print = function() { nplots = length(self$plots) if (nplots==1) { print(self$plots[[1]]) } else { args <- c(self$plots, list(ncol = 2)) do.call(gridExtra::grid.arrange, args) } } ) ) `+.ggmultiplotR6` <- function(e1, e2) { if ('ggmultiplotR6' %in% class(e1)) { plots <- lapply(e1$plots, function(x) { x + e2 }) return(ggmultiplotR6$new(plots = plots)) } else { e2name <- deparse(substitute(e2)) if (ggplot2::is.theme(e1)) return(ggplot2:::add_theme(e1, e2, e2name)) else if (ggplot2::is.ggplot(e1)) return(ggplot2:::add_ggplot(e1, e2, e2name)) } }
が、これをそのまま実行すると 以下の通りエラーになる。
p1 <- qplot(Petal.Width, Petal.Length, colour = Species, data = iris) p2 <- qplot(Sepal.Width, Petal.Length, colour = Species, data = iris) mp <- ggmultiplotR6$new(plots = list(p1, p2)) mp + theme_bw() # 以下にエラー mp + theme_bw() : 二項演算子の引数が数値ではありません # 追加情報: 警告メッセージ: # メソッド ("+.ggmultiplotR6", "+.gg") は "+" に対しては矛盾しています
エラーの理由は、 S3 総称関数が 複数の引数を受け取った場合、すべて引数について対応する関数を探すため。上の例では、ggmultiplotR6 と gg インスタンス同士の加算になり、定義した +.ggmultiplotR6 と {ggplot2} 内で定義されている +.gg が衝突してエラーになる。詳細は {R6} 作者の方の以下の説明がわかりやすい。
リンク先に書いてある通り、演算子に対応する関数の定義を同一にすれば回避できる。
`+.gg` <- `+.ggmultiplotR6` mp <- ggmultiplotR6$new(plots = list(p1, p2)) mp + theme_bw() # OK (出力省略)
補足 演算子がパッケージ中で定義される場合は、上の方法ではなく.onload 中で registerS3method する必要がある (詳細はリンク先)。
まとめ
{R6} を使う場合は クラス同士の演算は 単一の S3 総称関数として定義する必要がある。そのため、クラスが増えてくると定義が結構複雑になりそう。
また、上の例のように 異なるパッケージ中のクラスとの演算を定義したい場合、相手側の 演算子定義と衝突しないような配慮が必要。相手側を上書きするのはあまりうれしくないので、自分のパッケージは当面 S4 を使うことにした。
ということで {R6} で 演算子を使いたい場合は少し気をつけたほうがよさげ。
2/28 追記
下のやり方でいけるかも。後で試します。
2/29 追記
試してみた。setOldClass を以下のように使うことで、新規で定義した関数は期待通り setMethod できる。
class(mp) # [1] "ggmultiplotR6" "R6" setOldClass(c('ggmultiplotR6', 'R6')) isClass('ggmultiplotR6') # [1] TRUE setGeneric("foo", signature = "x", def = function(x) standardGeneric("foo") ) # [1] "foo" setMethod("foo", c(x = "ggmultiplotR6"), definition = function(x) { "I'm the method for `R6`" }) # [1] "foo" foo(mp) # [1] "I'm the method for `R6`"
しかしながら、既存の演算子への setMethod には効果がない。出力を見た感じ、S3 総称関数の定義が使われているようだ。
setMethod('+', c(e1 = 'ggmultiplotR6', e2 = 'ANY'), function(e1, e2) { plots <- lapply(e1$plots, function(x) { x + e2 }) ggmultiplotR6$new(plots = plots) }) # [1] "+" mp + theme_bw() # NULL
何かいいやり方がないかは探したい。
RStan / PyStan 開発版を GitHub からインストールする
最近ちょっとした事情で Stan を使いたく、状態空間モデルの勉強とあわせて こんな感じ でやっている。その環境構築ネタ。
補足 Stan って何?という方は StanTutorial がわかりやすい。
Stan の公式バインディングとしては R 用の RStan、Python 用の Pystan、コマンドライン用の CmdStanと 3 つある。うち、自分が使うのは RStan と PyStan。
- RStan Getting Started · stan-dev/rstan Wiki · GitHub
- Getting started — PyStan 2.5.0.2dev documentation
これらの標準版のインストールについては、上のドキュメントに OS 別のハマりどころも含めて整理されていてよい。標準版しか使わないよ、という方は上だけ読んでおけば OK。
自分はある issue の修正確認のために GitHub にある開発版をインストールしようとしたのだが、手順も見当たらずちょっとハマったのでメモ。
補足 自分の OS は Mac OSX 10.10.1 だが、Linux 系は同じ手順でいけると思う。
RStan
必要パッケージのインストール
一度 普通に RStan をインストールして、依存パッケージは入った状態になっている前提で。通常インストールされる依存パッケージに加え、make するには {RInside} が必要。
install.packages('RInside')
git リポジトリの clone
以降はシェルから。RStan のリポジトリは R のラッパー部分のみを管理しており、 Stan のコア部分は同梱されていない。そのため、Stan, RStan 両方を clone する必要がある。clone 後、Stan のコア部分を RStan の指定のフォルダにコピーする。
git clone git://github.com/stan-dev/stan.git git clone git://github.com/stan-dev/rstan.git cp -r stan/* rstan/stan cd rstan/rstan
makefile の変更
自分は TeX 設定してないので、vignette, manual をビルドしないように設定 ( makefile 108 行目のコメントアウトを外す)。
ifeq ($(NOTBUILD),FALSE) - $(R) CMD build rstan --md5 $(BUILD_OPTS) # --no-build-vignettes --no-manual + $(R) CMD build rstan --md5 $(BUILD_OPTS) --no-build-vignettes --no-manual endif
インストール
make build make install
インストール結果の確認
リポジトリ上のバージョンはリリースまで書き変わらない (直前のリリースのバージョンになっている) ようなので、ライブラリインストールパスのタイムスタンプが書き換わっているかどうかを確認した。
02/01追記 rstan のロード時に、インストール時刻と git のリビジョン番号が出ていた。これで確認すればよい。
library(rstan) # rstan (Version 2.5.0, packaged: 2015-01-24 06:59:27 UTC, GitRev: 7d6bf44c5b45)
PyStan
必要パッケージのインストール
標準版で必要なもの ( Numpy, Cython ) はインストールされている前提。
git リポジトリの clone
シェルから。コアとバインディング両方を clone するのは RStan と同様。必要なリポジトリを clone してコピーする。
git clone git://github.com/stan-dev/stan.git git clone git://github.com/stan-dev/pystan.git cp -r stan/* pystan/pystan/stan cd pystan
(2015/01/31時点の情報) pystan/_chain.pyx の修正
PyStan と Stan は完全に同期してメンテナンスされているわけではないらしく、2015/01/31 時点でそのままではビルドできない。原因は pystan/_chain.pyx が Stan のリポジトリから削除されたファイル var_stack_def.hpp を読み込もうとしているため。とりあえず該当箇所をコメントアウトする。
参考 https://groups.google.com/forum/#!topic/stan-dev/5WMyEDliS0I
- cdef extern from "stan/agrad/rev/var_stack_def.hpp": - pass + # cdef extern from "stan/agrad/rev/var_stack_def.hpp": + # pass
インストール
自分は リポジトリからインストールするときは setup.py develop を指定している。これは、ソースファイルを site-packages 以下にコピーせず、現在のパスにあるものを利用するオプション。リポジトリに対して行った修正が再インストールなしで反映されるので便利。
参考 setuptools - Python setup.py develop vs install - Stack Overflow
ARCHFLAGS は PyStan Wiki を元に指定。
ARCHFLAGS=-Wno-error=unused-command-line-argument-hard-error-in-future sudo python setup.py develop
インストール結果の確認
setup.py develop オプションでインストールしたとき、ライブラリのパスは git リポジトリのパスになる。
import pystan pystan # <module 'pystan' from '/Users/xxx/Documents/Git/pystan/pystan/__init__.py'>
終わり。
R {ggplot2} の散布図に凸包 / 確率楕円を描きたい
小ネタ。{ggplot2} でグループ別の散布図を描くときに、ちょっと飾り付けをしてグループをわかりやすくしたい。
凸包 (Convex)
最初にベースとなる散布図を描く。
library(dplyr) library(ggplot2) df <- iris p <- ggplot(df, aes(x = Petal.Width, y = Petal.Length)) + geom_point() p

まずは 散布図全体について凸包をとる。ある点の集合の凸包は、 grDevices::chull で計算できる。chull は凸な点の index を返すので、この返り値に含まれるデータのみをフィルタして geom_polygon に渡せばよい。
chull(df[c('Petal.Width', 'Petal.Length')]) # [1] 44 17 23 14 33 25 135 123 119 110 145 115 hulls <- df[chull(df[c('Petal.Width', 'Petal.Length')]), ] hulls # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 44 5.0 3.5 1.6 0.6 setosa # 17 5.4 3.9 1.3 0.4 setosa # 23 4.6 3.6 1.0 0.2 setosa # 14 4.3 3.0 1.1 0.1 setosa # 33 5.2 4.1 1.5 0.1 setosa # 25 4.8 3.4 1.9 0.2 setosa # 135 6.1 2.6 5.6 1.4 virginica # 123 7.7 2.8 6.7 2.0 virginica # 119 7.7 2.6 6.9 2.3 virginica # 110 7.2 3.6 6.1 2.5 virginica # 145 6.7 3.3 5.7 2.5 virginica # 115 5.8 2.8 5.1 2.4 virginica p + geom_polygon(data = hulls, alpha = 0.2)

凸包をグループ別に描画したい場合は、{dplyr} で各グループへ chull を適用する。
p <- ggplot(df, aes(x = Petal.Width, y = Petal.Length, colour = Species)) + geom_point() hulls <- df %>% dplyr::group_by(Species) %>% dplyr::do(.[chull(.[c('Petal.Width', 'Petal.Length')]), ]) hulls # Source: local data frame [22 x 5] # Groups: Species # # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 1 5.4 3.9 1.3 0.4 setosa # 2 4.6 3.6 1.0 0.2 setosa # 3 4.3 3.0 1.1 0.1 setosa # 4 5.2 4.1 1.5 0.1 setosa # 5 4.8 3.4 1.9 0.2 setosa # 6 5.1 3.8 1.9 0.4 setosa # 7 5.0 3.5 1.6 0.6 setosa # 8 5.1 2.5 3.0 1.1 versicolor # 9 4.9 2.4 3.3 1.0 versicolor # 10 5.8 2.7 4.1 1.0 versicolor # 11 6.1 2.8 4.7 1.2 versicolor # 12 6.0 2.7 5.1 1.6 versicolor # 13 6.7 3.0 5.0 1.7 versicolor # 14 5.9 3.2 4.8 1.8 versicolor # 15 5.8 2.8 5.1 2.4 virginica # 16 4.9 2.5 4.5 1.7 virginica # 17 6.0 2.2 5.0 1.5 virginica # 18 6.1 2.6 5.6 1.4 virginica # 19 7.7 2.8 6.7 2.0 virginica # 20 7.7 2.6 6.9 2.3 virginica # 21 7.2 3.6 6.1 2.5 virginica # 22 6.7 3.3 5.7 2.5 virginica p + geom_polygon(data = hulls, alpha = 0.2)
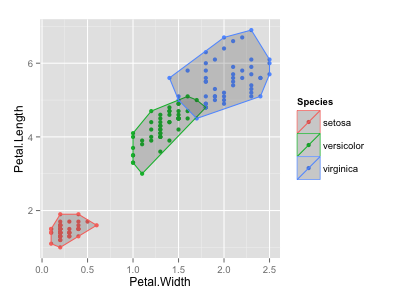
参考 以下リンクにある plyr::ddply を使うバージョンを参考にした。
確率楕円 (Probability Ellipse)
こっちは ggplot2 1.0.0 以降なら stat_ellipse 一発なので簡単。
p + stat_ellipse()

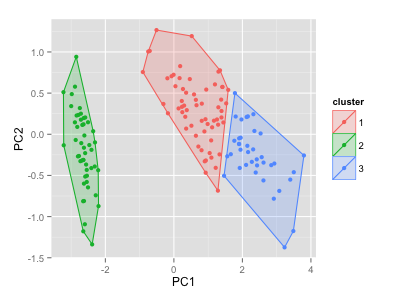
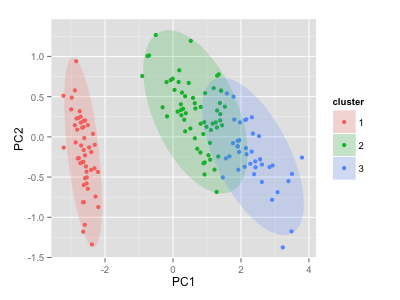
クラスタリング結果への凸包 / 確率楕円の描画
もともとやりたかったのは {ggplot2} + {ggfortify} でクラスタリング結果を autoplot するときに凸包 / 確率楕円を描画すること。これを autoplot 時の frame オプションで指定できるようにした。
ついでに {cluster} パッケージの非階層クラスタリング法 cluster::clara, cluster::fanny, cluster::pam も autoplot できるようにした。
# library(devtools) # install_github('sinhrks/ggfortify') library(ggplot2) library(ggfortify) df <- iris[-5] autoplot(kmeans(df, 3), original = iris, frame = TRUE)
library(cluster) autoplot(clara(df, 3), frame = TRUE, frame.type = 'norm')
ほか、細かい使い方はこちら。
クラスタリング以外の autoplot についてはこちら。

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
R で Google Speech API を使ってこっそりがんばりたい
この記事は R Advent Calendar 2014 (ATND) の26日目の記事です。
こういう話がある。
すばらしいパッケージだ。特に yeah::zoi はよい。これを使えば今日も一日頑張れそうな気がする。
library(yeah) yeah::zoi()
しかし、この関数は 周りに人がいる場合は 利用に若干のさしさわりがある。テキストで表示したら?とも思うが、ただの代替テキスト表示ではなんか味気ないし、パッケージに新しい関数が追加された場合に即座に楽しめない。なんとかする方法はないだろうか、、、?
少し考えた結果、Google の音声認識 API である Google Speech API を使うことにした。音声ファイルを認識 -> テキスト化して表示すれば、周りに人がいる場合もこっそり何度でも楽しめるし、新しい関数が追加されてもすぐに追従することが可能だ。
Google Speech API 利用手順
Google Speech API を利用するには、以下2つの手続きが必要。
Chromium Dev group への参加
Chromium Dev group へアクセスしてグループに join する。これをやっておかないと、Google Developer Console の API 一覧に Speech API が表示されない。
参考: Google Speech API V2 - Stack Overflow
Google Developer Console での API 有効化 & KEY 取得
手順は以下に記載されている。
.flac ファイルの準備
Google Speech API へは .flac ファイルを送る必要があるようだ。ffmpeg でも何でも 好きなものを使って変換する。
ffmpeg -i zoi.wav -vn -ac 1 -ar 16000 -acodec flac zoi.flac
補足 R のライブラリパスは .Library で確認できる。この中から yeah/sounds/ ディレクトリを探す。
.Library
# [1] "/Library/Frameworks/R.framework/Resources/library"
プログラム
上記リンクに記載されている Python のサンプルプログラムを参考に。 まず Google Speech API からレスポンスを取得するところまで。
library(jsonlite) library(httr) fname <- 'zoi.flac' apikey = '<your API key>' url <- paste0('https://www.google.com/speech-api/v2/', 'recognize?xjerr=1&client=chromium&lang=ja-JP', '&maxresults=10&pfilter=0&xjerr=1&key=', apikey) r <- POST(url, content_type('audio/x-flac; rate=16000'), body = upload_file(fname))
返り値は json 形式になる。が、レスポンス中の json データには root が 2つあるため、{httr} が内部で利用している {jsonlite} ではパースに失敗してエラーになってしまう。
content(r, encoding = 'utf-8') # Error in parseJSON(txt) : parse error: trailing garbage # {"result":[]} {"result":[{"alternative":[{"tr # (right here) ------^
中身を表示したければ、明示的に type = 'text' を指定して文字列としてパース & 表示する。音声が聞けないのは やはりちょっと寂しい、、、。が、そのぶん認識結果の候補が 複数 出てくるので たくさんがんばれる気がする。
txt <- content(r, type = 'text', encoding = 'utf-8') # ※出力は適当に整形 txt # [1] "{\"result\":[]}\n # {\"result\":[{\"alternative\": # [{\"transcript\":\"今日も1日頑張るぞい\",\"confidence\":0.84639674}, # {\"transcript\":\"今日も1日頑張るぞ\"}, # {\"transcript\":\"今日も1日頑張るぞぃ\"}, # {\"transcript\":\"今日も一日頑張るぞ\"}, # {\"transcript\":\"今日も一日がんばるぞぃ\"}, # {\"transcript\":\"今日も1日がんばるぞぃ\"}, # {\"transcript\":\"今日も1日頑張るぞオイ\"}, # {\"transcript\":\"今日も一日がんばるゾ\"}, # {\"transcript\":\"今日も一日がんばるぞオイ\"}, # {\"transcript\":\"今日も1日がんばるゾ\"}], # \"final\":true}],\"result_index\":0}\n"
結果を data.frame として取得したければ、文字列置換で valid な json フォーマットにしてから jsonlite::fromJSON。
library(stringr) jobj <- fromJSON(paste0('[', stringr::str_replace(txt, c('\n'), c(",")), ']')) jobj['result'][[1]][[2]]['alternative'][[1]][[1]] # transcript confidence # 1 今日も1日頑張るぞい 0.8463967 # 2 今日も1日頑張るぞ NA # 3 今日も1日頑張るぞぃ NA # 4 今日も一日頑張るぞ NA # 5 今日も一日がんばるぞぃ NA # 6 今日も1日がんばるぞぃ NA # 7 今日も1日頑張るぞオイ NA # 8 今日も一日がんばるゾ NA # 9 今日も一日がんばるぞオイ NA # 10 今日も1日がんばるゾ NA
まとめ
Google Speech API を使うことによって、周りを気にせず がんばるぞい 可能な環境を構築することができた。
R {arules} によるアソシエーション分析をちょっと詳しく <2>
こちらの続き。
データの作り方 (承前)
単体の list や data.frame から arules::transactions インスタンスを作る方法は前回まとめた。
加えて、一般のデータでありえそうな 正規化された形を考える。サンプルは コンビニのPOSデータをイメージして、
の 2 テーブルからなるデータとする。必要な部分だけ抜き出すと、例えばこんな形。
library(arules) tran.df = data.frame(日時 = paste0('2014-12-22 ', seq(9, 20, 1), ':00'), レジ番号 = rep(1, 12), レシート番号 = seq(1, 12), 年齢層 = rep(c('30代', '20代', '10代'), 4)) goods.df = data.frame(レシート番号 = c(1, 2, 2, 3, 4, 4, 5, 6, 6, 6, 7, 7, 8, 8, 9, 9, 10, 10, 11, 12, 12), 購入商品 = c('おにぎり', 'コーラ', 'サンドイッチ', 'お茶', 'おにぎり', 'お茶', 'お茶', 'コーラ', 'サンドイッチ', 'お茶', 'おにぎり', 'サンドイッチ', 'おにぎり', 'お茶', 'サンドイッチ', 'コーラ', 'おにぎり', 'サンドイッチ', 'おにぎり', 'コーラ', 'サンドイッチ')) tran.df # 日時 レジ番号 レシート番号 年齢層 # 1 2014-12-22 9:00 1 1 30代 # 2 2014-12-22 10:00 1 2 20代 # 3 2014-12-22 11:00 1 3 10代 # 4 2014-12-22 12:00 1 4 30代 # 5 2014-12-22 13:00 1 5 20代 # 6 2014-12-22 14:00 1 6 10代 # 7 2014-12-22 15:00 1 7 30代 # 8 2014-12-22 16:00 1 8 20代 # 9 2014-12-22 17:00 1 9 10代 # 10 2014-12-22 18:00 1 10 30代 # 11 2014-12-22 19:00 1 11 20代 # 12 2014-12-22 20:00 1 12 10代 # レシート番号 購入商品 # 1 1 おにぎり # 2 2 コーラ # 3 2 サンドイッチ # 4 3 お茶 # 5 4 おにぎり # 6 4 お茶 # 7 5 お茶 # 8 6 コーラ # 9 6 サンドイッチ # 10 6 お茶 # 11 7 おにぎり # 12 7 サンドイッチ # 13 8 おにぎり # 14 8 お茶 # 15 9 サンドイッチ # 16 9 コーラ # 17 10 おにぎり # 18 10 サンドイッチ # 19 11 おにぎり # 20 12 コーラ # 21 12 サンドイッチ
これを 1 トランザクション = レシート番号ごとに集計して arules::transactions インスタンスにしたい。
まず goods.df を トランザクション形式にするためには、各アイテムをレシート番号単位でまとめたリストに変換 -> transactions 化すればよいので、以下のような関数 decanonicalize を作って、
library(magrittr) library(dplyr) decanonicalize <- function(row, rdf){ details <- rdf %>% dplyr::filter(レシート番号 == row$レシート番号) %>% magrittr::extract2('購入商品') %>% as.vector() } tran.list <- split(tran.df, rownames(tran.df)) tran <- lapply(tran.list, decanonicalize, goods.df) tran # $`1` # [1] "おにぎり" # ..... # $`12` # [1] "コーラ" "サンドイッチ" tran <- as(tran, 'transactions') LIST(tran) # $`1` # [1] "おにぎり" # ..... # $`12` # [1] "コーラ" "サンドイッチ"
さらに マスタの顧客属性 (ここでは年齢層のみ) を紐付けたい、なんて場合はそれぞれを arules::transactions にしてから merge。
tran.base <- as(select(tran.df, 年齢層), 'transactions') tran <- merge(tran.base, tran) LIST(tran) # [[1]] # [1] "年齢層=30代" "おにぎり" # ..... # [[12]] # [1] "年齢層=10代" "コーラ" "サンドイッチ"
補足 原則、何か前処理したい場合は arules::transactions 化する前に行って merge したほうが楽。例外は arules::addComplement (後述)。
補足 縦方向にトランザクションを追加したい場合は c(tran1, tran2)
ルール抽出時の前処理
arules::transactions インスタンスができたので、前回と同じく arules::apriori で普通のルール抽出ができるようになった。
# 経過出力を抑制 control <- list(verbose = FALSE) rules <- apriori(tran, parameter = list(support = 0.2), control = control) inspect(rules) # lhs rhs support confidence lift # 1 {コーラ} => {サンドイッチ} 0.3333333 1 2
ここで、あるアイテムを "含まない" 場合のルールを抽出したいことがある。そんなときは arules::addComplement を使って、トランザクションにダミーアイテムを追加してルール抽出すればよい。トランザクションに "コーラを含まない" 場合のダミーアイテム "!コーラ" を追加してルール抽出すると、
tran <- addComplement(tran, 'コーラ', '!コーラ') rules <- apriori(tran, parameter=list(support=0.2), control = control) inspect(rules) # lhs rhs support confidence lift # 1 {コーラ} => {サンドイッチ} 0.3333333 1.0 2.0 # 2 {お茶} => {!コーラ} 0.3333333 0.8 1.2 # ..... # 5 {年齢層=30代, # !コーラ} => {おにぎり} 0.2500000 1.0 2.0
2番目のルールのように "お茶を買った人はコーラを同時に買いにくい" という画期的な発見がもたらされることがある。
頻出アイテムセットの取得
また 条件 -> 結論の形にこだわらず頻出アイテムセットを取り出したい場合は arules::eclat
isets <- eclat(tran, parameter=list(support=0.3)) class(isets) inspect(isets) # items support # 1 {コーラ, # サンドイッチ} 0.3333333 # ..... # 11 {コーラ} 0.3333333
arules::apriori で抽出したルールに含まれる アイテムセットを取得するには arules::generatingItemsets。ルールとして絞られた結果からアイテムセットを取り出すため、上の結果とは一致しない。重複した アイテムセットを削除したい場合は unique。
inspect(unique(generatingItemsets(rules))) # items support # 1 {コーラ, # サンドイッチ} 0.3333333 # ..... # 4 {年齢層=30代, # おにぎり, # !コーラ} 0.2500000
また、アイテムセットに対してはいくつかの集合演算が定義されている。各アイテムセットが それぞれ 最大の頻出アイテムセットかどうか (自分自身を含むほかの頻出アイテムセットがないかどうか) を調べるには arules::is.maximal
# ラベルを一覧表示するため data.frame に変換 as(isets, 'data.frame')$items # [1] {コーラ,サンドイッチ} {お茶,!コーラ} {おにぎり,!コーラ} {!コーラ} # [5] {おにぎり} {サンドイッチ} {お茶} {年齢層=30代} # [9] {年齢層=20代} {年齢層=10代} {コーラ} # 11 Levels: {!コーラ} {おにぎり,!コーラ} {おにぎり} {お茶,!コーラ} {お茶} {コーラ,サンドイッチ} ... {年齢層=30代} is.maximal(isets) # [1] TRUE TRUE TRUE FALSE FALSE FALSE FALSE TRUE TRUE TRUE FALSE
また、抽出した頻出アイテムセットについて元データ (トランザクション) を確認したい場合は arules::supportingTransactions でトランザクション IDを拾ってスライス。
LIST(supportingTransactions(isets[1], tran)) # $`{コーラ,サンドイッチ}` # [1] 4 5 9 12 LIST(tran[LIST(supportingTransactions(isets[1], tran))[[1]]]) # [[1]] # [1] "年齢層=30代" "コーラ" "サンドイッチ" # ..... # [[4]] # [1] "年齢層=10代" "コーラ" "サンドイッチ"
ファイルへの書き込み / 読み込み
arules::transactions をファイルに保存する形式として basket 形式と single 形式の二通りがある。それぞれ、
basket形式: 1 トランザクションを 1 行に保存する形式single形式: 1 アイテム 1 行に保存する形式 (アイテムごとに正規化した形式)
arules 内での処理を考えた場合、 basket 形式 のほうが使い勝手はよい。
basket 形式
まず書き込みは arules::write でファイル名 + フォーマットを指定すればよい。
write(tran, file = 'basket.tsv', format = 'basket')
書き込まれたファイルの中身はこんな感じになる。
年齢層=30代 おにぎり !コーラ 年齢層=20代 おにぎり サンドイッチ !コーラ ..... 年齢層=10代 コーラ サンドイッチ
読み込みは arules::read.transactions で、同じくファイル名 + フォーマットを指定。
LIST(read.transactions('basket.tsv', format='basket')) # [[1]] # [1] "!コーラ" "おにぎり" "年齢層=30代" # ..... # [[12]] # [1] "コーラ" "サンドイッチ" "年齢層=10代"
single 形式
single で保存する場合は、各トランザクションに含まれる アイテムの個数が一致していない場合はエラーになるようだ。
write(tran, file = 'single.tsv', format = 'single') # Error in data.frame(transactionID = rep(names(l), lapply(l, length)), : # arguments imply differing number of rows: 0, 41
別のサンプルデータを使って挙動をみる。
df <- data.frame(x = c(TRUE, FALSE, TRUE), y = c(TRUE, TRUE, FALSE), z = c(TRUE, TRUE, FALSE)) tran.single <- as(df, 'transactions') write(tran.single, file='single.tsv', format='single')
書き込まれたファイルの中身にはヘッダがついている。
transactionID item 1 1 x=TRUE 2 1 y=TRUE 3 1 z=TRUE 4 2 x=FALSE ..... 9 3 z=FALSE
single 形式での読み取りの際には、 cols オプションで "トランザクションIDを含む列", "アイテム名を含む列" を vector として指定する必要がある。
また、read.transactions には、read.tableのようにヘッダをスキップするオプションがない、、、。そのままだとヘッダ部分もトランザクションとして読まれてしまい、あまりうれしくない。
LIST(read.transactions('single.tsv', format='single', cols=c(2, 3))) # $`1` # [1] "y=TRUE" "z=TRUE" # ..... # $`3` # [1] "x=TRUE" "y=FALSE" "z=FALSE" # # $item # [1] "1"
遺された謎、、、itemsetInfo
arules::transactions は @itemsetInfo というプロパティを持っており、これは arules::itemsetInfo で参照できる。が、{arules} のソースをみても このプロパティを ルール抽出 / 頻出アイテムセット抽出に使っている様子はない。何に使うんだこれは、、、?
itemsetInfo(tran) # data frame with 0 columns and 0 rows
とりあえず以下のようにすれば データに @itemsetInfo プロパティを持たせられることはわかった。単純に アイテムをカテゴリ分けするためのものなのだろうか。
m = list(飲料=c('コーラ', 'お茶'), 食品=c('おにぎり', 'サンドイッチ')) m # $飲料 # [1] "コーラ" "お茶" # # $食品 # [1] "おにぎり" "サンドイッチ" im <- as(as.data.frame(m), 'itemMatrix') itemsetInfo(im) # itemsetID # 1 飲料 # 2 食品 im['飲料'] # itemMatrix in sparse format with # 1 rows (elements/transactions) and # 4 columns (items) LIST(im['飲料']) # $飲料 # [1] "お茶" "コーラ"
まとめ
{arules}でのトランザクションデータ作成 / 前処理などをざっとまとめた。- 次回以降は 系列パターンマイニングを行う
{arulesSequences}の予定。
R {arules} によるアソシエーション分析をちょっと詳しく <1>
今週は系列パターンマイニング用 R パッケージ {arulesSequences} と格闘していた。使い方にところどころよくわからないポイントがあり、思ったよりも時間がかかってしまった。
関連パッケージである {arules} ともども、ネットには簡単な分析についての情報はあるが、 データの作り方/操作についてはまとまったものがないようだ。とりあえず自分が調べたことをまとめておきたい。2 パッケージで結構なボリュームになるため、全 4 記事分くらいの予定。
概要
まずはパターンマイニングの手法を簡単に整理する。いずれもトランザクションと呼ばれるデータの系列を対象にする。トランザクションとは 1レコード中に複数の要素 (アイテム) を含むもの。例えば、
- POSデータ: 1トランザクション = POSレジの売上 1回。アイテムはそのときに売れた個々の商品。
- アンケート調査: 1トランザクション = アンケートの1回答。アイテムは個々の設問への解答内容。
複数のトランザクションからなるデータから、よく起きるパターンを発見・列挙する手法が頻出パターンマイニング。{arules} と {arulesSequences} でできることは、
| アソシエーション分析 (Association analysis) | 系列パターンマイニング (Sequential pattern mining) | |
|---|---|---|
| 抽出されるパターンのイメージ | XのときYが発生しやすい | Xの後にYが発生しやすい |
| パッケージ | {arules} |
{arulesSequences} |
| 備考 | トランザクション中のアイテムの関係をみる。トランザクションの順序は関係ない | 連続するトランザクションに含まれるアイテムの関係をみる。トランザクションの順序が重要 |
補足 以降、処理したい一群のトランザクションを "データ", データ中の1レコードを "トランザクション" と書く。
補足 IDA@SMU: Intelligent Data Analysis Lab - Southern Methodist University をみると、{arules} ファミリーには他にも {arulesClassify}, {arulesNBMiner} という関連パッケージがあるようだ。こちらは使ったことないのでそのうち。
アソシエーション分析とは
非常に簡単にいうと、データ全体を集計して ありそうなパターン (相関ルール) を抽出するもの。トランザクション中に アイテム が含まれるときにアイテム
も含まれる、なんてのが相関ルール。アソシエーション分析の用語では
は条件 (left hand side:lhs)、
は結論 (right hand side:rhs) と呼ぶ。相関ルールは
と書く。
アソシエーション分析では、以下のような尺度でルールを評価する。
支持度 (support)
両方のアイテム (の和集合) を含むトランザクションがデータ中に占める比率になる。支持度が高いルールはデータ全体でみてよく起きやすい。
データ全体のトランザクション数を , アイテム
を含むトランザクション数を
とあらわすとき、支持度は、
確信度 (confidence)
トランザクション中にアイテム が含まれるとき、アイテム
も同時に含まれる比率。確信度が高いルールでは、アイテム
を含むトランザクションにはアイテム
も含まれやすい。
リフト (lift)
日本語難しいが、"ルールによって起こる の頻度 / 全体での
の頻度"。 一般に、リフトが 1 より大きい場合にルールが有効 = ルールによってアイテム
が含まれやすくなる、とみなす。
で、何を重視すれば?
というのは目的によってことなる。
- 広く浅くいきたいなら 支持度が大きく 確信度/リフトがそこそこのルールを選ぶ。
- 狭く深くいきたいなら 支持度は小さめでも 確信度/リフトの高いルールを選ぶ。
という感じ。また、各数値はトランザクションの総数 / アイテムの偏りによっても変わるため、他のルールと比べてよしあしを判断する。
補足 これらを、トランザクションに あるアイテムが含まれる確率 の推定量としてとらえるなら、 支持度 =
, 確信度 =
, リフト =
。
パッケージのインストール
アソシエーション分析を行う {arules} パッケージをインストール / ロードする。
install.packages('arules') library(arules)
サンプルデータのロード
{arules} では データを作るところが一番わかりにくい (そして動きを理解していないとデータ作れない) なので、今回はとりあえず天下りで。arules に入っている Income データを使う。
このデータは、世帯収入と世帯の属性をアイテムとした一群のトランザクションからなる。このデータをアソシエーション分析することにより、" ならば 世帯収入が
(高い/低い) であることが多い" といったルールが出てくる。
data(Income) tran <- Income
読み込んだデータは arules::transactions インスタンスになっている。
class(tran) # [1] "transactions" # attr(,"package") # [1] "arules"
transactions の確認
データの中身を表示してみる。
tran # transactions in sparse format with # 6876 transactions (rows) and # 50 items (columns)
、、、これ、中身どう見るのかな?? というのが最初のつまずきポイントだと思う。
arules::transactions 中の各トランザクションはスライシングで取得できる。そこからトランザクションの詳細 (アイテム) をみる場合は arules::LIST を使うのがよい。
表示されるアイテム、例えば "income=$40,000+" は、 data.frame でいうと "income" カラムの値が "$40,000+" であるという意味。ほかのアイテムも世帯の属性を示すものであることがわかる。
# スライシングした結果も transactions インスタンス tran[1] # transactions in sparse format with # 1 transactions (rows) and # 50 items (columns) LIST(tran[1]) # $`2` # [1] "income=$40,000+" "sex=male" # [3] "marital status=married" "age=35+" # [5] "education=college graduate" "occupation=homemaker" # [7] "years in bay area=10+" "dual incomes=no" # [9] "number in household=2+" "number of children=1+" # [11] "householder status=own" "type of home=house" # [13] "ethnic classification=white" "language in home=english"
補足 as(tran, 'data.frame') で data.frameへ、 もしくは as(tran, 'matrix') で matrix へ変換しても中身を確認できるが、アイテム数が多い場合は表示がわかりにくい。このとき、as.data.frame(tran), as.matrix(tran) では coerce エラーになるので注意。
また、複数トランザクションを選択してスライシング & アイテム表示もできる。
tran[5:10] # transactions in sparse format with # 6 transactions (rows) and # 50 items (columns) LIST(tran[5:10]) # $`6` # [1] "income=$40,000+" "sex=male" # [3] "marital status=married" "age=35+" # [5] "education=no college graduate" "occupation=retired" # [7] "years in bay area=10+" "dual incomes=no" # [9] "number in household=1" "number of children=0" # [11] "householder status=own" "type of home=house" # [13] "ethnic classification=white" "language in home=english" # .....
transactions の要約表示
要約表示は summary。頻出するアイテムの頻度、分布などが表示される。
summary(tran) # transactions as itemMatrix in sparse format with # 6876 rows (elements/itemsets/transactions) and # 50 columns (items) and a density of 0.28 # # most frequent items: # language in home=english education=no college graduate # 6277 4849 # number in household=1 ethnic classification=white # 4757 4605 # years in bay area=10+ (Other) # 4446 71330 # # element (itemset/transaction) length distribution: # sizes # 14 # 6876 # # Min. 1st Qu. Median Mean 3rd Qu. Max. # 14 14 14 14 14 14 # # includes extended item information - examples: # labels variables levels # 1 income=$0-$40,000 income $0-$40,000 # 2 income=$40,000+ income $40,000+ # 3 sex=male sex male # # includes extended transaction information - examples: # transactionID # 1 2 # 2 3 # 3 4
トランザクションデータの詳細
arules::transactions は S4 クラスで、以下のようなプロパティを持つ。
@transactionInfo: 各トランザクションの番号を含むdata.frame@data: 各トランザクションについて、個々のアイテムが含まれるかどうかを示すMatrix::ngCMatrixクラス (真偽値をセルの値とする疎行列)@itemInfo: データに含まれるアイテムの一覧を含むdata.frame@itemsetInfo: アイテムのセットを含むdata.frame
arules にはこれらのプロパティにアクセスするための関数が用意されている。トランザクションの IDを確認するには arules::transactionInfo。一部のみ表示するために head をかます。
head(transactionInfo(tran)) # transactionID # 1 2 # 2 3 # 3 4 # 4 5 # 5 6 # 6 7
アイテムの一覧を表示するには arules::itemInfo。
head(itemInfo(tran)) # labels variables levels # 1 income=$0-$40,000 income $0-$40,000 # 2 income=$40,000+ income $40,000+ # 3 sex=male sex male # 4 sex=female sex female # 5 marital status=married marital status married # 6 marital status=cohabitation marital status cohabitation
また アイテムのセットをあらわす arules::itemsetInfo は、 Income データには定義されていないので空の data.frameになる。これの使い方は別途。
head(itemsetInfo(tran)) # data frame with 0 columns and 0 rows
アイテムの頻度の確認
transactions に含まれるアイテムの発生頻度を確認するには arules::itemFrequency。既定だと相対頻度になるため、発生回数(絶対頻度)をみたい場合は type = 'absolute' を指定。
head(itemFrequency(tran, type = 'absolute')) # income=$0-$40,000 income=$40,000+ sex=male # 4280 2596 3067 # sex=female marital status=married marital status=cohabitation # 3809 2652 536
また、arules::itemFrequencyPlot で頻度をプロットできる。 アイテム数が多い場合は、 topN キーワードでプロットするアイテム数を指定する。
itemFrequencyPlot(tran, type = 'absolute', topN = 5, cex = 0.8)

また、個人的によく使うのは arules::affinity。アイテム に対する
の affinity
は、
で計算される。各アイテム別々での発生頻度に対する同時発生頻度で、affinity が大きいほど アイテム と
は同時に含まれやすい (別々に含まれることは少ない)。
出力大量になるためスライシングして表示。同時発生しえない組み合わせの affinity は 0。
affinity(tran)[1:4, 1:4] # income=$0-$40,000 income=$40,000+ sex=male sex=female # income=$0-$40,000 0.0000000 0.0000000 0.3399599 0.425877 # income=$40,000+ 0.0000000 0.0000000 0.2697309 0.277933 # sex=male 0.3399599 0.2697309 0.0000000 0.000000 # sex=female 0.4258770 0.2779330 0.0000000 0.000000
ルール抽出
今回は arules::apriori 。
apriori アルゴリズムでの頻出パターンマイニング
arules::transactions インスタンスをそのまま arules::apriori に渡せばルールが出てくる。出力は 8664 個のルールが見つかったことを示す。
rule <- apriori(tran) # parameter specification: # confidence minval smax arem aval originalSupport support minlen maxlen target ext # 0.8 0.1 1 none FALSE TRUE 0.1 1 10 rules FALSE # # algorithmic control: # filter tree heap memopt load sort verbose # 0.1 TRUE TRUE FALSE TRUE 2 TRUE # # apriori - find association rules with the apriori algorithm # version 4.21 (2004.05.09) (c) 1996-2004 Christian Borgelt # set item appearances ...[0 item(s)] done [0.00s]. # set transactions ...[50 item(s), 6876 transaction(s)] done [0.00s]. # sorting and recoding items ... [30 item(s)] done [0.00s]. # creating transaction tree ... done [0.00s]. # checking subsets of size 1 2 3 4 5 6 7 8 done [0.06s]. # writing ... [8664 rule(s)] done [0.00s]. # creating S4 object ... done [0.00s].
経過出力を消したい場合は control オプションで verbose = FALSE を指定。
apriori(tran, control = list(verbose = FALSE)) # set of 8664 rules
作成されたルールのセットは、arules::rules インスタンスになっている。
class(rule) # [1] "rules" # attr(,"package") # [1] "arules"
rules の確認
スライシングによって個々のルールが取得できるのは arules::transactions と同様。
中身を表示する場合は arules::inspect。
例えば 5番目のルールは、"dual incomes=no のとき language in home=english である" というルールを示す。列名はそれぞれ、条件(lhs)、結論(rhs)、支持度 (support)、確信度 (confidence)、リフト (lift) に対応。
inspect(rules[5]) # lhs rhs support confidence lift # 1 {dual incomes=no} => {language in home=english} 0.1364165 0.9196078 1.007364
補足 arules::inspect はルールを表示するための関数である (ルールをdata.frameに変換する関数ではない)。そのため、ルールの一部を表示したいときには arules::inspect に渡す前にフィルタする必要がある。
# OK inspect(head(rules)) # 略 # NG! head(inspect(rules)) # 略 (全ルール表示される)
補足 arules::LIST はrules に対しては使えない。
ルールの並べ替えと条件抽出
抽出されたルールを、並べ替え / フィルタして確認したいということはよくある。並べ替えには arules::sort。
# 信頼度 で並べ替え sort(rules, by = 'confidence') # set of 8664 rules # 信頼度の上位5件を表示 inspect(head(sort(rules, by = 'confidence'), n = 5)) # lhs rhs support confidence lift # 1 {marital status=single} => {dual incomes=not married} 0.4091041 1 1.671366 # 2 {marital status=single, # occupation=student} => {dual incomes=not married} 0.1449971 1 1.671366 # 3 {marital status=single, # householder status=live with parents/family} => {dual incomes=not married} 0.1884817 1 1.671366 # 4 {marital status=single, # type of home=apartment} => {dual incomes=not married} 0.1339442 1 1.671366 # 5 {marital status=single, # number in household=2+} => {dual incomes=not married} 0.1545957 1 1.671366
ルールの左辺、もしくは右辺に対して 条件を付けてフィルタする場合は arules::subset。arules::subsetには条件抽出を行うための条件式が渡せる。条件式中では、lhs, rhs, support, confidence, lift が変数として、また以下の演算子も使える。上記ヘルプ中に各演算子の使用例が記載されている。
%in%: 指定したアイテムを含むルールを抽出%pin%: 文字列を指定し、部分一致したアイテムを含むルールを抽出%ain%: 複数のアイテムをvectorで指定し、それらを全て含むルールを抽出
例えば、"income=$0-$40,000" になりやすいルールを見つけたい場合は、rhs が "income=$0-$40,000" であるルールを抽出すればよいので、
subset(rules, subset = rhs %in% 'income=$0-$40,000') # set of 571 rules # 詳細表示 inspect(head(subset(rules, subset = rhs %in% 'income=$0-$40,000'))) # lhs rhs support confidence lift # 1 {occupation=student} => {income=$0-$40,000} 0.1381617 0.8421986 1.353027 # 2 {householder status=live with parents/family} => {income=$0-$40,000} 0.1669575 0.8141844 1.308021 # 3 {type of home=apartment} => {income=$0-$40,000} 0.2242583 0.8137203 1.307276 # 4 {marital status=single} => {income=$0-$40,000} 0.3302792 0.8073231 1.296999 # 5 {marital status=single, # occupation=student} => {income=$0-$40,000} 0.1230366 0.8485456 1.363224 # 6 {age=14-34, # occupation=student} => {income=$0-$40,000} 0.1348168 0.8465753 1.360059 # 上記 かつ 信頼度が 0.84 より大きいルールを表示 inspect(head(subset(rules, subset = rhs %in% 'income=$0-$40,000' & confidence > 0.84))) # lhs rhs support confidence lift # 1 {occupation=student} => {income=$0-$40,000} 0.1381617 0.8421986 1.353027 # 2 {marital status=single, # occupation=student} => {income=$0-$40,000} 0.1230366 0.8485456 1.363224 # 3 {age=14-34, # occupation=student} => {income=$0-$40,000} 0.1348168 0.8465753 1.360059 # 4 {occupation=student, # dual incomes=not married} => {income=$0-$40,000} 0.1301629 0.8475379 1.361605 # 5 {education=no college graduate, # occupation=student} => {income=$0-$40,000} 0.1282723 0.8440191 1.355952 # 6 {occupation=student, # language in home=english} => {income=$0-$40,000} 0.1195462 0.8456790 1.358619
rules の要約表示
arules::transactions の場合と同じく summary。ルールの長さごとの頻度や、支持度 / 確信度 / リフトの分布が表示される。
summary(rules) # set of 8664 rules # # rule length distribution (lhs + rhs):sizes # 1 2 3 4 5 6 7 8 # 1 56 615 2287 3387 1925 385 8 # # Min. 1st Qu. Median Mean 3rd Qu. Max. # 1.000 4.000 5.000 4.888 6.000 8.000 # # summary of quality measures: # support confidence lift # Min. :0.1001 Min. :0.8000 Min. :0.8971 # 1st Qu.:0.1101 1st Qu.:0.8436 1st Qu.:1.0813 # Median :0.1241 Median :0.9027 Median :1.3297 # Mean :0.1393 Mean :0.9021 Mean :1.4099 # 3rd Qu.:0.1510 3rd Qu.:0.9574 3rd Qu.:1.5309 # Max. :0.9129 Max. :1.0000 Max. :4.3554 # # mining info: # data ntransactions support confidence # tran 6876 0.1 0.8 # head(quality(rules))
apriori 実行時の条件指定
arules::apriori の実行時点でフィルタ条件がわかっている場合は、その条件をキーワードとして渡すことによって抽出されるルールを絞り込める。各キーワードに対して引数のリストを渡す形なのでちょっとわかりにくい。詳細は arules::apriori。ならびに、各引数クラスのヘルプを参照。
parameter: 引数のリストまたはAPparameterインスタンスappearance: 引数のリストまたはAPappearanceインスタンスcontrol: 引数のリストまたはAPcontrolインスタンス
# 経過出力を抑制 control <- list(verbose = FALSE) # 支持度が 0.6 以上のルールのみ抽出 r <- apriori(tran, parameter = list(supp = 0.6, target = "rules"), control = control) inspect(r) # lhs rhs support confidence lift # 1 {} => {language in home=english} 0.9128854 0.9128854 1.0000000 # 2 {years in bay area=10+} => {language in home=english} 0.6013671 0.9300495 1.0188020 # 3 {ethnic classification=white} => {language in home=english} 0.6595404 0.9847991 1.0787763 # 4 {number in household=1} => {language in home=english} 0.6495055 0.9388270 1.0284171 # 5 {education=no college graduate} => {language in home=english} 0.6343805 0.8995669 0.9854106 # 条件 (左辺) が "occupation=student" のルールのみ抽出 r <- apriori(tran, appearance = list(items = c("occupation=student"), default = 'rhs'), control = control) inspect(r) # lhs rhs support confidence lift # 1 {} => {language in home=english} 0.9128854 0.9128854 1.0000000 # 2 {occupation=student} => {marital status=single} 0.1449971 0.8838652 2.1604897 # 3 {occupation=student} => {age=14-34} 0.1592496 0.9707447 1.6583454 # 4 {occupation=student} => {dual incomes=not married} 0.1535777 0.9361702 1.5646831 # 5 {occupation=student} => {income=$0-$40,000} 0.1381617 0.8421986 1.3530274 # 6 {occupation=student} => {education=no college graduate} 0.1519779 0.9264184 1.3136839 # 7 {occupation=student} => {language in home=english} 0.1413613 0.8617021 0.9439324
APparameter, APappearance, APcontrolはそれぞれ空のリスト、もしくは NULL からインスタンス化すれば、どんなインスタンスが作られているのかわかる。
as(list(), 'APparameter') # confidence minval smax arem aval originalSupport support minlen maxlen target ext # 0.8 0.1 1 none FALSE TRUE 0.1 1 10 rules FALSE
データの作り方
最後、単純な transactions データの作り方。よく使うのは以下の 3パターンだと思う。
vector のリストから作る
最も単純な方法。リストの各要素 (vector) が 1 トランザクション、 vector 内の各要素が アイテムになる。
l <- list(c('x', 'z'), c('x', 'y'), c('x', 'y')) as(l, 'transactions') # transactions in sparse format with # 3 transactions (rows) and # 3 items (columns) LIST(as(l, 'transactions')) # [[1]] # [1] "x" "z" # # [[2]] # [1] "x" "y" # # [[3]] # [1] "x" "y"
値が真偽値の data.frame から作る
data.frame のカラム名について、各カラムが含まれる / 含まれない場合それぞれをアイテムとして ルールを作成したい場合はこの形式。
df <- data.frame(x = c(TRUE, FALSE, TRUE), y = c(TRUE, TRUE, FALSE), z = c(TRUE, TRUE, FALSE)) as(df, 'transactions') # transactions in sparse format with # 3 transactions (rows) and # 6 items (columns) LIST(as(df, 'transactions')) # $`1` # [1] "x=TRUE" "y=TRUE" "z=TRUE" # # $`2` # [1] "x=FALSE" "y=TRUE" "z=TRUE" # # $`3` # [1] "x=TRUE" "y=FALSE" "z=FALSE"
値が factor の data.frame から作る
data.frame のカラム名が複数の値をとりうるカテゴリで、各値をアイテムとして扱いたい場合はこの形式。transactions インスタンスにする対象は 全て factor である必要がある。
df <- data.frame(x = factor(c(1, 2, 1)), y = factor(c('a', 'a', 'b')), z = factor(c('A', 'A', 'B'))) as(df, 'transactions') # transactions in sparse format with # 3 transactions (rows) and # 6 items (columns) LIST(as(df, 'transactions')) # $`1` # [1] "x=1" "y=a" "z=A" # # $`2` # [1] "x=2" "y=a" "z=A" # # $`3` # [1] "x=1" "y=b" "z=B"
まとめ
transactions,rulesに対する比較的 単純な処理をまとめた。- 単純な
transactionsインスタンスを作成できるようになった。
が、現実のデータは最初は上記のような扱いやすい形をしていないことも多いし、transactions に対していろいろと前処理が必要なこともある。今度はこのあたりの方法をまとめるつもり。
12/23追記 続きはこちら。
{flexclust} + DTW で 時系列を k-means クラスタリングする
概要
下の記事のつづき。下の記事では DTW (Dynamic Time Warping) 距離を使って階層的クラスタリングを行った。続けて、 DTW 距離を使って 非階層的クラスタリング (k-means法) を試してみる。
stats::kmeans では任意の距離関数を利用することはできないため、任意の距離関数が利用できる {flexclust} というパッケージを使うことにした。
補足 DTW についてはこちら。
動的時間伸縮法 / DTW (Dynamic Time Warping) を可視化する - StatsFragments
インストール
install.packages('flexclust') library(flexclust)
{flexclust} の使い方
サンプルデータは前回記事と同一。 まずは既定 (ユークリッド距離) で k-means してみる。サンプルデータは行持ちでわたす必要があるので転置する。
クラスタリングした結果は TSclust::cluster.evaluation を使って正分類率を算出して評価する。そのため、 {TSclust} もロードする。
library(TSclust) # クラスタ数は 3 res = kcca(t(data), 3) cluster.evaluation(rep(c(1, 2, 3), rep(N, 3)), res@cluster) # [1] 0.7705387
任意の距離関数で k-means する場合は、kcca の family オプションで、 kccaFamily インスタンス化した距離関数を渡してやればよい。ここでは、既定の方法と同じ flexclust::distEuclidean を距離関数として渡してみる。
res = kcca(t(data), 3, family = kccaFamily(dist = distEuclidean)) cluster.evaluation(rep(c(1, 2, 3), rep(N, 3)), res@cluster) # [1] 0.7705387
補足 ここではたまたま 1回目 / 2回目の正分類率が同じだったが、乱数 / 中心点の初期値によっては正分類率が異なることもありうる。
{flexclust} に任意の距離関数を渡す
DTW を使って k-means するためには、上記の distEuclidean を参考にして距離関数を定義してやればよさそうだ。どのような定義であればよいのか調べるため、flexclust::distEuclidean の実装をみてみる。引数と返り値は、
x: 入力データ (次元は データ数 x データの長さ)centers: 現在のクラスタの中心点 (次元は クラスタ数 x データの長さ)z: 各入力データと各クラスタの距離 (次元は データ数 x クラスタ数)
distEucledean # function (x, centers) # { # if (ncol(x) != ncol(centers)) # stop(sQuote("x"), " and ", sQuote("centers"), " must have the same number of columns") # z <- matrix(0, nrow = nrow(x), ncol = nrow(centers)) # for (k in 1:nrow(centers)) { # z[, k] <- sqrt(colSums((t(x) - centers[k, ])^2)) # } # z # } # <environment: namespace:flexclust> # x として渡ってくるもの (データ数 x データの長さ の行列) # サンプルの場合、実際は 60行目 / 20列目までデータあり [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] U1 -0.6327521 2.8410958 3.4529034 3.76949256 4.306223 5.2242329 1.305026 3.493067 8.0427642 U2 -0.9945734 3.4368117 -0.9907990 2.60606620 1.453351 0.8575501 2.541472 1.869034 0.6731607 U3 0.7014436 0.9778474 0.5219079 3.13321606 3.984465 3.8435608 3.707534 4.780511 7.7889760 # center として渡ってくるもの (クラスタ数 x 系列の長さ の行列) # サンプルの場合、実際は 20列目までデータあり [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] D3 0.62595969 1.1235099 1.592674 -0.949247 -0.8132157 -2.0273653 -4.4877420 -5.137003 N17 -0.05983237 0.2078591 1.261035 1.826098 1.4504397 2.3426289 3.7686407 3.463603 N4 -1.52153133 -2.0982198 -1.930059 -4.379938 -2.9796999 -0.9948817 0.4933508 -3.084777 # z として返すべきもの (データ数 x クラスタ数 の行列) # サンプルの場合、実際は 60行目 までデータあり [,1] [,2] [,3] [1,] 69.75362 16.51187 44.71353 [2,] 71.45904 18.28978 45.20475 [3,] 57.58485 9.22195 32.93481 [4,] 74.50569 21.34163 48.72438 [5,] 74.53808 20.40498 49.45025 [6,] 66.04604 14.45167 40.60733
形式がわかったので、 TSclust::diss.DTWARP を利用して同様のロジックを作成する。
distDTWARP <- function (x, centers) { if (ncol(x) != ncol(centers)) stop(sQuote("x"), " and ", sQuote("centers"), " must have the same number of columns") z <- matrix(0, nrow = nrow(x), ncol = nrow(centers)) for (k in 1:nrow(centers)) { z[, k] <- apply(x, 1, function(x) diss.DTWARP(x, centers[k, ])) } z }
いざ実行。
res_dtw = kcca(t(data), 3, family = kccaFamily(dist = distDTWARP))
、、、。
、、、。
、、、反応ないな、、、。
動いていることを信じて処理時間を計ってみる。
res_dtw = kcca(t(data), 3, family = kccaFamily(dist = distDTWARP)) # ユーザ システム 経過 # 1142.323 44.924 1306.208 cluster.evaluation(rep(c(1, 2, 3), rep(N, 3)), res_dtw@cluster) # [1] 0.7653292
なんだこれは、、、。遅いし あんま結果もよくない。
{flexclust} に任意のクラスタ中心点更新関数を渡す
調べてみると、どうも kccaFamily に原因がありそうだ。 kccaFamily には 距離関数 dist のほかに、クラスタ中心点の更新に使う関数 cent が渡せる。ここで、dist には関数を指定 / cent には何も指定しない場合、kccaFamily では以下の関数 centOptim を使ってクラスタ中心点を更新する。DTW は 普通の距離関数に比べると計算コストが高いため、この optim での最適化に非常に時間がかかっていたようだ。
centOptim <- function (x, dist) { foo <- function(p) sum(dist(x, matrix(p, nrow = 1))) optim(colMeans(x), foo)$par }
クラスタ中心点の更新処理については ユークリッド距離の最小化 or クラスタの中央値で行うようにしてみる。感覚的には、これらの方法で更新されたクラスタの中心点を使った場合も、クラスタ内の各要素 / 中心点の DTW 距離は小さくなるはずだ。
# ユークリッド距離最小となるように中心点更新 cent <- function(x) centOptim(x, dist = distEuclidean) res_dtw_ceuc <- kcca(t(data), 3, family = kccaFamily(dist = distDTWARP, cent = cent)) # ユーザ システム 経過 # 3.374 0.118 3.942 cluster.evaluation(rep(c(1, 2, 3), rep(N, 3)), res_dtw_ceuc@cluster) # [1] 0.7638889 # クラスタの中央値で中心点更新 cent <- function(x) apply(x, 2, median) res_dtw_cmed <- kcca(t(data), 3, family = kccaFamily(dist = distDTWARP, cent = cent)) # ユーザ システム 経過 # 5.926 0.209 7.039 cluster.evaluation(rep(c(1, 2, 3), rep(N, 3)), res_dtw_cmed@cluster) # [1] 0.7638889
試してみると、処理時間はかなり短くなった。
比較
k-means 法でのクラスタリングは初期値の影響を受けるため、100回ほどループさせて比較してみた。 比較したのは 以下の3手法
Euclidean: ユークリッド距離で k-meansDTW (Euclidean): DTW で k-means、クラスタ中心点は ユークリッド距離最小となるよう更新DTW (median): DTW で k-means、クラスタ中心点は 中央値で更新
横軸に正分類率 / 縦軸に頻度として頻度分布を描いてみた。ベストな値は DTW (Euclidean) で出ていたが、顕著な差があるようには見えない。ループ回数を増やせば / 元データがちょっと違えば逆転しそうである。
ただ、DTW (Euclidean) の結果はばらつきが少ないようには見える。

まとめ
{flexclust} を使って DTW 距離で k-means してみた。その際、クラスタ中心点の更新を既定 ( DTW距離 +
optim) で行うのは処理速度の点で現実的でない。クラスタ中心点の更新を ユークリッド距離 or 中央値で行うことで処理速度は改善する。しかし、上記の方法/データではユークリッド距離での k-meansと比較して 劇的な差はみられなかった。確かめられていないのは、
補足
こちらの本に DTW での k-meansというトピック (時系列ではなく手書き文字についてだが) があるようだ。高い、、、。

Developing Concepts in Applied Intelligence (Studies in Computational Intelligence)
- 作者: Kishan G. Mehrotra,Chilukuri Mohan,Jae C. Oh,Pramod K. Varshney,Moonis Ali
- 出版社/メーカー: Springer
- 発売日: 2011/06/10
- メディア: ハードカバー
- この商品を含むブログを見る
2015/06/03 追記: 直近でブクマいただいていたので補足を。DTW は ユークリッド距離と比べると計算量が多いため、 k-means のように距離計算を複数回 繰り返すアルゴリズムとはあまり相性がよくない。今回、自分はトレンドによって時系列を分類したかったのだが、最終的には上昇/下降など 解釈可能なパターンをいくつか教師データとして用意して 最近傍法を使って分類した (距離計算の回数を減らし、かつ 解釈できるパターンに分類する必要があったため)。
{TSclust} ではじめる時系列クラスタリング
概要
こちらで書いた 動的時間伸縮法 / DTW (Dynamic Time Warping) を使って時系列をクラスタリングしてみる。ここからは パッケージ {TSclust} を使う
{TSclust} のインストール
install.packages('TSclust') library(TSclust)
サンプルデータの準備
{TSclust} では時系列間の距離を計算する方法をいくつか定義している。クラスタリングの際にどの定義 (距離) を使えばよいかは 時系列を何によって分類したいのかによる。{TSclust} に実装されているものをいくつかあげると、
diss.ACF: ACFdiss.CID: Complexity Correlations (よくわからん) で補正したユークリッド距離diss.COR: ピアソン相関 (ラグは考慮しない)diss.EUCL: ユークリッド距離diss.DTWARP: DTW 距離diss.DWT: Wavelet変換してユークリッド距離
自分は、"ある時点からのトレンドが どういったパターン (上昇, 下降, etc...) に属するかで分類したい" ので、今回はユークリッド距離か DTW 距離が適切かなと思う。
まず、自分が実際に処理したいものに近そうなサンプルデータを準備した。条件として考慮したのは、
- 背後にランダムだが一定のトレンドを持つ
- かつランダムな AR 構造を持つ
もの。上記のようなデータを 以下の3グループ, 各 20 系列作成した。
- 上昇トレンド : 変数名で
group1。ラベルはUで始まる。 - 停滞トレンド : 変数名で
group2。ラベルはNで始まる。 - 下降トレンド : 変数名で
group3。ラベルはDで始まる。
set.seed(1) # 各グループの系列数 N = 20 # 系列の長さ SPAN = 24 # トレンドが上昇/ 下降する時の平均値 TREND = 0.5 generate_ts <- function(m, label) { library(dplyr) # ランダムな AR 成分を追加 .add.ar <- function(x) { x + arima.sim(n = SPAN, list(ar = runif(2, -0.5, 0.5))) } # 平均が偏った 乱数を cumsum してトレンドとする d <- matrix(rnorm(SPAN * N, mean = m, sd = 1), ncol = N) %>% data.frame() %>% cumsum() d <- apply(d, 2, .add.ar) %>% data.frame() colnames(d) <- paste0(label, seq(1, N)) d } group1 = generate_ts(TREND, label = 'U') group2 = generate_ts(0, label = 'N') group3 = generate_ts(-TREND, label = 'D') data <- cbind(group1, group2, group3) data <- as.data.frame(data)
作成した系列 (グループで色分け)

階層的クラスタリングの実行と評価
まずは DTW を使って階層的クラスタリングを行い、デンドログラムを描く。手順は、
TSclust::dissで距離行列を求める。- あとは普通の
hclust同様。
# DTW 距離で距離行列を作成 d <- diss(data, "DTWARP") # hclust は既定で実行 = 最遠隣法 h <- hclust(d) par(cex=0.6) plot(h, hang = -1)
結果をみると、左のノードから順に、上昇グループ ("U"), 下降グループ ("D"), 停滞グループ ("N") で けっこう綺麗に分かれている気がする。

また、TSclust::cluster.evaluation を使ってクラスタの正分類率が算出できる。DTW を使った場合の 正分類率は 86.8 % になった。
補足 ここでの正分類率は サンプル作成の際に利用した ベースのトレンドを正とした。上のグラフで分かる通り AR 成分によって各グループが混ざってしまっているため、どんな方法を使っても正分類率 100 % になることはないと思うが、ひとつの基準ということで。
# クラスタ数は 3 とする clusters <- cutree(h, 3) # 正分類率の算出 cluster.evaluation(rep(c(1, 2, 3), rep(N, 3)), clusters) # 0.8675466
手法の比較
{TSclust} のいくつかの手法を比較してみる。{TSclust} の実行中に何かエラーが出た手法は除いた。
plot_group <- function(plot.data, cluster = NULL, method = '') { library(ggplot2) library(tidyr) plot.data$index <- 1:SPAN plot.data <- gather(plot.data, variable, value, -index) if (is.null(cluster)) { method = 'Original' plot.data$colour <- substr(plot.data$variable, 1, 1) plot.data$colour <- factor(plot.data$colour, levels = c('U', 'N', 'D')) } else { cluster <- as.factor(cluster) plot.data$colour <- rep(cluster, rep(SPAN, length(cluster))) } p <- ggplot(plot.data, aes(x = index, y = value, group = variable, colour = colour)) + geom_line() + xlab('') + ylab('') + ggtitle(method) if (!is.null(cluster)) { cluster.true <- rep(c(1, 2, 3), rep(N, 3)) rate <- cluster.evaluation(cluster.true, cluster) rate <- paste0(round(rate * 100, 1), '%') p <- p + annotate(geom = 'text', x = 5, y = 20, label = rate, size = 10) } print(p) } methods <- c("ACF", "AR.LPC.CEPS", "AR.PIC", "CID", "COR", "CORT", "DTWARP", "DWT", "EUCL", "INT.PER", "PACF", "PDC", "PER", "SPEC.LLR", "SPEC.GLK", "SPEC.ISD") for (method in methods) { print(method) d <- diss(data, method) h <- hclust(d) clusters <- cutree(h, 3) plot_group(data, cluster = clusters, method = method) }
そこそこうまくいったものを上位から順に並べる。結果として、(最遠隣法では) DTW が最も正分類率が高かった。
※ 各系列について、クラスタリング後のラベルで色分け / 左上の数値が正分類率。
DTW

CID

Wavelet変換

ユークリッド距離

まとめ
{TSclust} パッケージで 階層的クラスタリングをするとき、DTW を使うとユークリッド距離よりもうまくトレンドを拾って分類できている (ように見える)。
クラスタリングする時系列の数が少ない場合は 階層的クラスタリングは使えそう。ただし、ちゃんとやる場合は {TSclust} 側の手法だけでなく hclust の手法との組み合わせについても あてはまりをみるべき。
自分は最終的に より系列数が多い実データにあてたいので、DTW 距離を使って k-means 法でクラスタリングしたい。stats::kmeans では 自作の距離関数を利用できないが、ちょっと調べたところ {flexclust} というパッケージを使えばよさげ。
cluster analysis - How to specify distance metric while for kmeans in R? - Stack Overflow
つづく、、、。
動的時間伸縮法 / DTW (Dynamic Time Warping) を可視化する
いま手元に 20万件くらいの時系列があって、それらを適当にクラスタリングしたい。どうしたもんかなあ、と調べていたら {TSclust} というまさになパッケージがあることを知った。
このパッケージでは時系列の類似度を測るためのさまざまな手法 (=クラスタリングのための距離) を定義している。うちいくつかの手法を確認し、動的時間伸縮法 / DTW (Dynamic Time Warping) を試してみることにした。
DTWの概要
時系列相関 (CCF) の場合は 片方を 並行移動させているだけなので 2つの系列の周期が異なる場合は 相関はでにくい。
DTW では 2つの時系列の各点の距離を総当りで比較した上で、系列同士の距離が最短となるパスを見つける。これが DTW 距離 になる。そのため、2つの系列の周期性が違っても / 長さが違っても DTW 距離を定義することができる。
アルゴリズム
英語版 Wikipedia がわかりやすい。 DTW の計算には、{TSculst} も内部で使っている {dtw} というパッケージがあるが、アルゴリズムがシンプルなのでここでは 直接 実装してみた。
dtw_distance <- function(ts_a, ts_b, d = function(x, y) abs(x-y), window = max(length(ts_a), length(ts_b))) { ts_a_len <- length(ts_a) ts_b_len <- length(ts_b) # コスト行列 (ts_a と ts_b のある2点間の距離を保存) cost <- matrix(NA, nrow = ts_a_len, ncol = ts_b_len) # 距離行列 (ts_a と ts_b の最短距離を保存) dist <- matrix(NA, nrow = ts_a_len, ncol = ts_b_len) cost[1, 1] <- d(ts_a[1], ts_b[1]) dist[1, 1] <- cost[1, 1] for (i in 2:ts_a_len) { cost[i, 1] <- d(ts_a[i], ts_b[1]) dist[i, 1] <- dist[i-1, 1] + cost[i, 1] } for (j in 2:ts_b_len) { cost[1, j] <- d(ts_a[1], ts_b[j]) dist[1, j] <- dist[1, j-1] + cost[1, j] } for (i in 2:ts_a_len) { # 最短距離を探索する範囲 (ウィンドウサイズ = ラグ) window.start <- max(2, i - window) window.end <- min(ts_b_len, i + window) for (j in window.start:window.end) { # dtw::symmetric1 と同じパターン choices <- c(dist[i-1, j], dist[i, j-1], dist[i-1, j-1]) cost[i, j] <- d(ts_a[i], ts_b[j]) dist[i, j] <- min(choices) + cost[i, j] } } return(dist[nrow(dist), ncol(dist)]) } ts_a <- AirPassengers[31:45] ts_b <- AirPassengers[41:55] dtw_distance(ts_a, ts_b) # 286
{dtw} パッケージの計算結果と一致することを確認。
library(dtw) d <- dtw::dtw(ts_a, ts_b, step.pattern = symmetric1) d$distance # 286
可視化
{animation} を含むコードは gist においた (重いです)。
- 2つの時系列の破線で結ばれた点同士を順に比較する
- 比較した2点間の距離 (コスト) を計算する。セルの色が緑色なら、比較した2点間の距離は小さい。(左下)
- その際、左側、左下、下にある距離行列のセル + 上で計算した2点間の距離 (コスト) を足して、最も小さい値をその時点の DTW 距離にする (そこまでの2点間の距離 (コスト) の和が最小になるようなパスが自動的に見つかる)。セルの色が青色なら、そのセルまでの DTW 距離は小さい。(右下)
- 距離行列の右上のセルに到達したとき、そのセルの値が 系列同士の DTW 距離になっている

{dtw} の dtwPlotTwoWay 関数を使うと DTW で 2 つの系列の各点がどのようにマッピングされたのかプロットできる。
dtwPlotTwoWay(d, ts_a, ts_b)

{dtw} の使い方
dtw では以下のようなオプションが指定でき、より柔軟に DTW 距離が求められる。
dist.method: 2点間の距離(コスト)を求める関数step.pattern: DTW距離行列にコストを追加する際の関係式。window.type: window の種類open.begin,open.end: 最初 / 最後の要素のうちマッチしないものを捨てるかどうか
step.pattern なんかはいろいろあって奥が深そう。
# step.pattern のうち、上のサンプルと同じ動きのもの symmetric1 # Step pattern recursion: # g[i,j] = min( # g[i-1,j-1] + d[i ,j ] , # g[i ,j-1] + d[i ,j ] , # g[i-1,j ] + d[i ,j ] , # ) # # Normalization hint: NA # べつのパターン asymmetric # Step pattern recursion: # g[i,j] = min( # g[i-1,j ] + d[i ,j ] , # g[i-1,j-1] + d[i ,j ] , # g[i-1,j-2] + d[i ,j ] , # ) # # Normalization hint: N
11/16追記: 続きはこちら。