Python XGBoost の変数重要度プロット / 可視化の実装
Gradient Boosting Decision Tree の C++ 実装 & 各言語のバインディングである XGBoost、かなり強いらしいという話は伺っていたのだが自分で使ったことはなかった。こちらの記事で Python 版の使い方が記載されていたので試してみた。
その際、Python でのプロット / 可視化の実装がなかったためプルリクを出した。無事 マージ & リリースされたのでその使い方を書きたい。まずはデータを準備し学習を行う。
import numpy as np import xgboost as xgb from sklearn import datasets import matplotlib.pyplot as plt plt.style.use('ggplot') xgb.__version__ # '0.4' iris = datasets.load_iris() dm = xgb.DMatrix(iris.data, label=iris.target) np.random.seed(1) params={'objective': 'multi:softprob', 'eval_metric': 'mlogloss', 'eta': 0.3, 'num_class': 3} bst = xgb.train(params, dm, num_boost_round=18)
1. 変数重要度のプロット
Python 側には R のように importance matrix を返す関数がない。GitHub 上でも F score を見ろ、という回答がされていたので F score をそのままプロットするようにした。
xgb.plot_importance(bst)

棒グラフの色、タイトル/軸のラベルは以下のように変更できる。
xgb.plot_importance(bst, color='red', title='title', xlabel='x', ylabel='y')

color にリストを渡せば棒ごとに色が変わる。色の順序は matplotlib の barh と同じく下からになる。また、ラベルを消したい場合は None を渡す。
xgb.plot_importance(bst, color=['r', 'r', 'b', 'b'], title=None, xlabel=None, ylabel=None)

XGBoost は内部的に変数名を保持していない。変数名でプロットしたい場合は 一度 F score を含む辞書を取得して、キーを差し替えてからプロットする。
bst.get_fscore() # {'f0': 17, 'f1': 16, 'f2': 95, 'f3': 59} iris.feature_names # ['sepal length (cm)', # 'sepal width (cm)', # 'petal length (cm)', # 'petal width (cm)'] mapper = {'f{0}'.format(i): v for i, v in enumerate(iris.feature_names)} mapped = {mapper[k]: v for k, v in bst.get_fscore().items()} mapped # {'petal length (cm)': 95, # 'petal width (cm)': 59, # 'sepal length (cm)': 17, # 'sepal width (cm)': 16} xgb.plot_importance(mapped)

2. 決定木のプロット
以下二つの関数を追加した。graphviz が必要なためインストールしておくこと。
to_graphviz: 任意の決定木をgraphvizインスタンスに変換する。IPython上であればそのまま描画できる。plot_tree:to_graphvizで取得したgraphvizインスタンスをmatplotlibのAxes上に描画する。
IPython から実行する。num_trees で指定した番号に対応する木が描画される。
xgb.to_graphviz(bst, num_trees=1)

エッジの色分けが不要なら明示的に黒を指定する。
xgb.to_graphviz(bst, num_trees=2, yes_color='#000000', no_color='#000000')

IPython を使っていない場合や、サブプロットにしたい場合には plot_tree を利用する。
_, axes = plt.subplots(1, 2) xgb.plot_tree(bst, num_trees=2, ax=axes[0]) xgb.plot_tree(bst, num_trees=3, ax=axes[1])

何かおかしいことをやっていたら 本体の方で issue お願いします。
10/3追記 その後の修正を以下にしました。変数名の指定などが簡単になっています。
Python xray で 多次元データを pandas ライクに扱う
はじめに
pandas では 2 次元、表形式のデータ ( DataFrame ) を主な対象としているが、ときには 3 次元以上のデータを扱いたい場合がある。そういった場合 以下のような方法がある。
MultiIndexを使い、2 次元のデータにマッピングする。- 3 次元データ構造である
Panel、4 次元のPanel4D、もしくは任意の次元のデータ構造 (PanelND) をファクトリ関数 で定義して使う。 numpy.ndarrayのまま扱う。
自分は MultiIndex を使うことが多いが、データを 2 次元にマップしなければならないため 種類によっては直感的に扱いにくい。Panel や PanelND は DataFrame と比べると開発が活発でなく、特に Panel4D、PanelND は 現時点で Experimental 扱いである。また、今後の扱いをどうするかも議論がある。numpy.ndarray では データのラベル付けができない。
xray とは
ラベル付きの多次元データを多次元のまま 直感的に扱えるパッケージとして xray がある。作者は pandas 開発チーム仲間の shoyer だ。そのため、API は pandas にかなり近いものになっている。
xray は大きく以下ふたつのデータ構造を持つ。これらを使うと多次元データをより直感的に操作することができる。
xray.DataArray:numpyの多次元配列にラベルでのアクセスを追加したもの。データは任意の次元を持つことができる。xray.Dataset: 複数のxray.DataArrayをまとめるセット。
インストール
pip で。
$ pip install xray
データの準備
まず、必要なパッケージをインポートする。
import numpy as np import pandas as pd pd.__version__ # '0.16.2' import xray xray.__version__ # '0.5.2'
サンプルデータとして、気象庁から 2015年7月20日〜25日の東京、八王子、大島の最高気温のデータを使いたい。以下のサイトからダウンロードした。データは 日付、場所 2 次元の配列となる。
- 出典:気象庁ホームページ (URL: http://www.data.jma.go.jp/gmd/risk/obsdl/index.php)

data2 = np.array([[34.2, 30.2, 33.5], [36.0, 29.3, 34.9], [35.3, 29.7, 32.8], [30.1, 27.6, 30.4], [33.6, 30.1, 33.9], [34.1, 28.0, 33.1]])
この numpy.ndarray から xray.DataArray インスタンスを作成する。データへアクセスするためのラベル ( pandas でいう index のようなもの ) は coords キーワードで指定する。また、dims キーワードを使って各次元それぞれにも名前をつけることができる。ここでは 以下のような DataArray を作成している。
- データは 2 つの次元
date,locationを持つ。 date次元はdatesで指定される 6 つの日付のラベルを持つ。location次元はlocsで指定される 3 つの場所のラベルを持つ。
2 次元のため、pandas.DataFrame でいうと date が indexに、 location が columns に対応しているイメージ。
locs = [u'八王子', u'大島', u'東京'] dates = pd.date_range('2015-07-20', periods=6, freq='D') da2 = xray.DataArray(data2, coords=[dates, locs], dims=['date', 'location']) da2 # <xray.DataArray (date: 6, location: 3)> # array([[ 34.2, 30.2, 33.5], # [ 36. , 29.3, 34.9], # [ 35.3, 29.7, 32.8], # [ 30.1, 27.6, 30.4], # [ 33.6, 30.1, 33.9], # [ 34.1, 28. , 33.1]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 各次元の名前 da2.dims # ('date', 'location') # 各次元の詳細 da2.coords # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # date 次元のラベル da2.coords['date'] # <xray.DataArray 'date' (date: 6)> # array(['2015-07-20T09:00:00.000000000+0900', # '2015-07-21T09:00:00.000000000+0900', # '2015-07-22T09:00:00.000000000+0900', # '2015-07-23T09:00:00.000000000+0900', # '2015-07-24T09:00:00.000000000+0900', # '2015-07-25T09:00:00.000000000+0900'], dtype='datetime64[ns]') # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ...
データの選択
xray でのデータ選択は pandas と類似の方法で行える。pandas でのデータ選択についてはこちらを。
ある次元から、特定のラベルをもつデータを選択したい場合、pandas と同じく .loc が使える。
# 7/25 のデータを選択 da2.loc[pd.Timestamp('2015-07-25')] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
また、対象の次元が datetime64 型の場合は、pandas と同じく日時文字列でも指定が可能 (部分文字列によるスライシング、詳細以下)。
# 7/25 のデータを選択 da2.loc['2015-07-25'] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
また、ラベルではなく位置 ( n番目など ) によって選択する場合は __getitem__ を使う。pandas では .iloc に対応。
# 末尾 = 最新の日付のデータを取得 da2[-1] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
2 次元目以降をラベル / 位置によって指定する場合は、引数をカンマで区切り、選択に利用する次元の位置に対応する値を渡す。
# date は全選択、location が東京のデータを選択 da2.loc[:, u'東京'] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # date は全選択、location が 3 番目 = 東京のデータを選択 da2[:, 2] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac'
データ選択に利用する次元自体もラベルで指定したい場合は .sel を使う。.sel では次元の順序に関係なく値を指定できるため、高次元になった場合もシンプルだ。
# 東京のデータを選択 da2.sel(location=u'東京') # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # 7/22, 7/23 の 東京のデータを選択 da2.sel(location=u'東京', date=pd.DatetimeIndex(['2015-07-22', '2015-07-23'])) # <xray.DataArray (date: 2)> # array([ 32.8, 30.4]) # Coordinates: # * date (date) datetime64[ns] 2015-07-22 2015-07-23 # location <U3 u'\u6771\u4eac'
もしくは .loc, __getitem__ に以下のような辞書を渡してもよい。
# 東京のデータを選択 da2.loc[dict(location=u'東京')] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac'
公式ドキュメント では、xray.DataArray からのデータ選択を以下のような表で整理している。
| 次元の指定 | 値の指定 | DataArray のメソッド |
|---|---|---|
| 位置 | 位置 | da2[:, 0] |
| 位置 | ラベル | da2.loc[:, u'東京'] |
| ラベル | 位置 | da2.isel(location=0), da2[dict(space=0)] |
| ラベル | ラベル | da2.sel(location=u'東京'), da2.loc[dict(location=u'東京')] |
次元の追加
ここまでは 2 次元のデータを使っていたが、最低気温と平均気温のデータを追加して 3 次元のデータとする。
# 最低気温 data3 = np.array([[24.7, 25.1, 25.8], [23.4, 24.3, 25.4], [22.7, 23.8, 25.4], [24.5, 24.4, 24.7], [24.9, 24.6, 25.0], [23.7, 24.8, 24.8]]) # 平均気温 data4 = np.array([[27.8, 26.5, 28.8], [29.7, 26.3, 29.4], [29.5, 26.5, 28.9], [26.9, 25.3, 27.0], [27.7, 26.4, 28.1], [29.0, 26.1, 28.6]]) data = np.dstack([data2, data3, data4]) # 日時、場所、データの種類 の 3 次元 data.shape # (6, 3, 3) da3 = xray.DataArray(data, coords=[dates, locs, [u'最高', u'最低', u'平均']], dims=['date', 'location', 'type']) da3 # <xray.DataArray (date: 6, location: 3, type: 3)> # array([[[ 34.2, 24.7, 27.8], # [ 30.2, 25.1, 26.5], # [ 33.5, 25.8, 28.8]], # # ... # # [[ 34.1, 23.7, 29. ], # [ 28. , 24.8, 26.1], # [ 33.1, 24.8, 28.6]]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
3 次元以上の場合もデータ選択のルールは一緒なのでわかりやすい。
# 7/24 のデータを選択 da3.loc['2015-07-24'] # <xray.DataArray (location: 3, type: 3)> # array([[ 33.6, 24.9, 27.7], # [ 30.1, 24.6, 26.4], # [ 33.9, 25. , 28.1]]) # Coordinates: # date datetime64[ns] 2015-07-24 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # 7/24 の 最高気温のデータを選択 da3.loc['2015-07-24', :, u'最高'] # <xray.DataArray (location: 3)> # array([ 33.6, 30.1, 33.9]) # Coordinates: # date datetime64[ns] 2015-07-24 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # type <U2 u'\u6700\u9ad8' # 東京 の 最高気温のデータを選択 da3.sel(location=u'東京', type=u'最高') # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # type <U2 u'\u6700\u9ad8'
算術演算
pandas と同じく、xray.DataArray 同士での算術演算が可能。各日の最高気温と最低気温の差を求めると、
da3.sel(type=u'最高') - da3.sel(type=u'最低') # <xray.DataArray (date: 6, location: 3)> # array([[ 9.5, 5.1, 7.7], # [ 12.6, 5. , 9.5], # [ 12.6, 5.9, 7.4], # [ 5.6, 3.2, 5.7], # [ 8.7, 5.5, 8.9], # [ 10.4, 3.2, 8.3]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 結果から 八王子 のデータだけを選択 (da3.sel(type=u'最高') - da3.sel(type=u'最低')).sel(location=u'八王子') # <xray.DataArray (date: 6)> # array([ 9.5, 12.6, 12.6, 5.6, 8.7, 10.4]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u516b\u738b\u5b50'
データのグループ化 / 集約
グループ化 / 集約も pandas とほぼ同じ形式でできる。location によってグループ化し、期間中の最高気温を出してみる。
da3.groupby('location') # <xray.core.groupby.DataArrayGroupBy at 0x109a56f90> da3.groupby('location').max() # <xray.DataArray (location: 3)> # array([ 36. , 30.2, 34.9]) # Coordinates: # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
グループ化した結果は、イテレーションによって順番に処理することもできる。
for name, g in da3.groupby('location'): print(name) print(g) # 八王子 # <xray.DataArray (date: 6, type: 3)> # array([[ 34.2, 24.7, 27.8], # [ 36. , 23.4, 29.7], # [ 35.3, 22.7, 29.5], # [ 30.1, 24.5, 26.9], # [ 33.6, 24.9, 27.7], # [ 34.1, 23.7, 29. ]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # location <U4 u'\u516b\u738b\u5b50' # # 以降略
データの結合 / 連結
上で作成した DataArray に、別のデータを追加したい。適当なデータを探したところ、ある成人男性のお住まいの気温データ を見つけた。これを日別で集計して連結したい。
# GitHub からデータを取得 df = pd.read_csv('https://raw.githubusercontent.com/dichika/mydata/master/room.csv') df['time'] = pd.to_datetime(df['time']) # light は光量、temperatur は気温 df.head() # time light temperature # 0 2015-02-12 01:45:04 463.0 26.784 # 1 2015-02-12 02:00:03 473.0 26.630 # 2 2015-02-12 02:15:04 467.9 25.983 # 3 2015-02-12 02:30:04 0.0 25.453 # 4 2015-02-12 02:45:04 0.0 23.650 # 期間中にフィルタ df = df[df['time'] >= pd.Timestamp('2015-07-20')] # 日時でグループ化 / 集約 agg = df.groupby(pd.Grouper(key='time', freq='D'))['temperature'].agg(['max', 'min', 'mean']) agg # max min mean # time # 2015-07-20 28.374 26.450 27.484604 # 2015-07-21 33.790 26.800 30.132792 # 2015-07-22 34.180 27.070 30.134375 # 2015-07-23 28.779 27.412 28.290918
xray でデータを連結するためには、連結する次元 (ここでは location) 以外のデータの要素数を一致させる必要がある。上記のデータは数日遅れで公開されているため、直近を NaN でパディングして xray.DataArray を作成する。
agg.loc[pd.Timestamp('2015-07-24'), :] = np.nan agg.loc[pd.Timestamp('2015-07-25'), :] = np.nan agg # max min mean # time # 2015-07-20 28.374 26.450 27.484604 # 2015-07-21 33.790 26.800 30.132792 # 2015-07-22 34.180 27.070 30.134375 # 2015-07-23 28.779 27.412 28.290918 # 2015-07-24 NaN NaN NaN # 2015-07-25 NaN NaN NaN d = xray.DataArray(agg.values.reshape(6, 1, 3), coords=[agg.index, [u'誰かの家'], [u'最高', u'最低', u'平均']], dims=['date', 'location', 'type']) d # <xray.DataArray (date: 6, location: 1, type: 3)> # array([[[ 28.374 , 26.45 , 27.48460417]], # [[ 33.79 , 26.8 , 30.13279167]], # [[ 34.18 , 27.07 , 30.134375 ]], # [[ 28.779 , 27.412 , 28.29091765]], # [[ nan, nan, nan]], # [[ nan, nan, nan]]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U4 u'\u8ab0\u304b\u306e\u5bb6' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
データの連結は xray.concat で可能。新しい場所のデータを追加 (連結) したいので、対象の次元として location を指定する。
da3 = xray.concat([da3, d], dim='location') da3.sel(location=u'誰かの家') # <xray.DataArray (date: 6, type: 3)> # array([[ 28.374 , 26.45 , 27.48460417], # [ 33.79 , 26.8 , 30.13279167], # [ 34.18 , 27.07 , 30.134375 ], # [ 28.779 , 27.412 , 28.29091765], # [ nan, nan, nan], # [ nan, nan, nan]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # location <U4 u'\u8ab0\u304b\u306e\u5bb6'
ほか、merge で結合もできる。
Dataset の利用
xray では、DataArray クラスを複数まとめて Dataset クラスとして扱うことができる。元となる DataArray は同じ次元でなくてもよい。
Dataset を作成するため、降水量、湿度 それぞれ 2 次元の DataArray を用意する。
# 降水量 precip = np.array([[0, np.nan, 0], [0, np.nan, np.nan], [0, 0, np.nan], [6.5, 1, 4.5], [30.0, 0, 7.0], [0, np.nan, np.nan]]) precip = xray.DataArray(precip, coords=[dates, locs], dims=['date', 'location']) precip # <xray.DataArray (date: 6, location: 3)> # array([[ 0. , nan, 0. ], # [ 0. , nan, nan], # [ 0. , 0. , nan], # [ 6.5, 1. , 4.5], # [ 30. , 0. , 7. ], # [ 0. , nan, nan]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 湿度 humid = np.array([[np.nan, 88, 75], [np.nan, 85, 65], [np.nan, 87, 61], [np.nan, 91, 80], [np.nan, 86, 83], [np.nan, 88, 80]]) humid = xray.DataArray(humid, coords=[dates, locs], dims=['date', 'location']) humid # <xray.DataArray (date: 6, location: 3)> # array([[ nan, 88., 75.], # [ nan, 85., 65.], # [ nan, 87., 61.], # [ nan, 91., 80.], # [ nan, 86., 83.], # [ nan, 88., 80.]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
これらと気温データをあわせて xray.Dataset を作成する。Dataset が持つ次元は Dimenstions, Coordinates に表示される。
Dataset に含まれる DataArray は Data variables 中に表示され、それぞれどの次元を含んでいるかがわかる。元データと同じく、気温は 3 次元、ほかは 2 次元のデータとなっている。
ds = xray.Dataset({'temperature': da3,
'precipitation': precip,
'humidity': humid})
ds
# <xray.Dataset>
# Dimensions: (date: 6, location: 4, type: 3)
# Coordinates:
# * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ...
# * location (location) object u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
# * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
# Data variables:
# precipitation (date, location) float64 0.0 nan 0.0 nan 0.0 nan nan nan ...
# temperature (date, location, type) float64 34.2 24.7 27.8 30.2 25.1 ...
# humidity (date, location) float64 nan 88.0 75.0 nan nan 85.0 65.0 ...
ds.data_vars
# Data variables:
# precipitation (date, location) float64 0.0 nan 0.0 nan 0.0 nan nan nan ...
# temperature (date, location, type) float64 34.2 24.7 27.8 30.2 25.1 ...
# humidity (date, location) float64 nan 88.0 75.0 nan nan 85.0 65.0 ...
Dataset からのデータ選択は、Dataset に含まれるすべての DataArray に対して行われる。DataArray とは異なり、次元は必ずラベルで指定する必要がある。
| 次元の指定 | 値の指定 | Dataset のメソッド |
|---|---|---|
| 位置 | 位置 | なし |
| 位置 | ラベル | なし |
| ラベル | 位置 | ds.isel(location=2), ds[dict(location=2)] |
| ラベル | ラベル | ds.sel(location=u'東京'), ds.loc[dict(location=u'東京')] |
# 東京 のデータを選択 ds.sel(location=u'東京') # <xray.Dataset> # Dimensions: (date: 6, type: 3) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u6771\u4eac' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # Data variables: # precipitation (date) float64 0.0 nan nan 4.5 7.0 nan # temperature (date, type) float64 33.5 25.8 28.8 34.9 25.4 29.4 32.8 ... # humidity (date) float64 75.0 65.0 61.0 80.0 83.0 80.0 # 東京 の 平均気温 を選択 ds.sel(location=u'東京', type=u'平均')['temperature'] # <xray.DataArray 'temperature' (date: 6)> # array([ 28.8, 29.4, 28.9, 27. , 28.1, 28.6]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # type <U2 u'\u5e73\u5747' # location object u'\u6771\u4eac' # 東京 の 湿度 を選択 ds.sel(location=u'東京')['humidity'] # <xray.DataArray 'humidity' (date: 6)> # array([ 75., 65., 61., 80., 83., 80.]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u6771\u4eac' # 対応する DataArray がない場合は NaN となる ds.sel(location=u'誰かの家') # <xray.Dataset> # Dimensions: (date: 6, type: 3) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u8ab0\u304b\u306e\u5bb6' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # Data variables: # precipitation (date) float64 nan nan nan nan nan nan # temperature (date, type) float64 28.37 26.45 27.48 33.79 26.8 30.13 ... # humidity (date) float64 nan nan nan nan nan nan
そのほかの操作も DataArray と同じようにできる。
# location ごとに期間中の最大値を計算 ds.groupby('location').max() # <xray.Dataset> # Dimensions: (location: 3) # Coordinates: # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # Data variables: # precipitation (location) float64 30.0 1.0 7.0 # temperature (location) float64 36.0 30.2 34.9 # humidity (location) float64 nan 91.0 83.0
pandas のデータ形式への変換
DataArray, Dataset はそれぞれ pandas のデータに変換できる。元データが 3 次元以上の場合、変換後の pandas のデータは MultiIndex を持つことになる。
また、上の例では 次元が異なる DataArray から Dataset を作成した。このとき、存在しない次元 ( ここでは type ) のデータはすべて同じ値でパディングされる。
ds.to_dataframe()
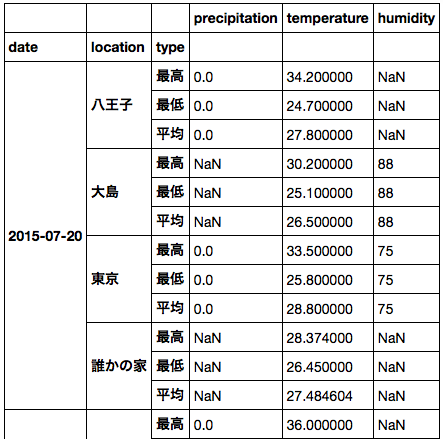
パディングしたくない場合は、個々の DataArray ごとに DataFrame に変換すればよい。
ds['humidity'].to_dataframe()
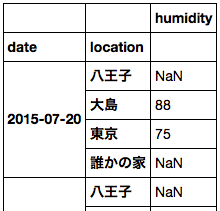
まとめ
xray を使えば 多次元のラベル付きデータを多次元のまま、pandas に近い方法で扱うことができる。
{rbokeh} で Bokeh を R から使いたい
はじめに
Bokeh は Python 以外にも R, Scala, Julia 用のパッケージを提供している。パッケージといっても Python の Bokeh と連携するものではなく、Bokeh がブラウザでのレンダリングに使っている Bokeh.js を各言語で扱えるようにするもののようだ。そのため、各パッケージはそれぞれ単体で利用できる。
うち、R 用のパッケージである {rbokeh} を使ってみたい。R には {htmlwidgets} を使った JavaScript 利用の可視化パッケージが多数提供されているが、数が多くて使い方が覚えられない。できれば {rbokeh} だけを使いたい。また、Pythonと見た目が揃えられると嬉しい。
インストール
現時点で CRAN には公開されていないため、GitHub からインストールする。現時点のバージョンは 0.2.3.2。
install.packages('htmlwidgets') install.packages('devtools') library(devtools) devtools::install_github('bokeh/rbokeh')
データの準備
サンプルとして {ggplot2} に含まれる mpg データを利用する。単に {rbokeh} を利用するだけなら {ggplot2} は不要。
library(ggplot2) library(rbokeh) dim(mpg) # [1] 234 11 head(mpg) # manufacturer model displ year cyl trans drv cty hwy fl class # 1 audi a4 1.8 1999 4 auto(l5) f 18 29 p compact # 2 audi a4 1.8 1999 4 manual(m5) f 21 29 p compact # 3 audi a4 2.0 2008 4 manual(m6) f 20 31 p compact # 4 audi a4 2.0 2008 4 auto(av) f 21 30 p compact # 5 audi a4 2.8 1999 6 auto(l5) f 16 26 p compact # 6 audi a4 2.8 1999 6 manual(m5) f 18 26 p compact
基本的な操作
Python の Bokeh は matplotlib を連想させる API を持っているが、 {rbokeh} は別の API ( {ggplot2} や {ggvis} に近い ) を持つ。そのため、API は言語ごとに覚える必要がある。
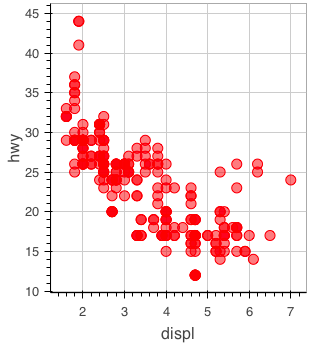
mpg データの displ 列と hwy 列を利用して散布図を描画する。
figure() %>% ly_points(x = displ, y = hwy, data = mpg)

加えて、 class 列で色分けする。サイズの変更など、利用できるオプションは help(ly_points) で確認できる。
figure() %>% ly_points(x = displ, y = hwy, color = class, data = mpg)

色名は文字列で指定することもできる。
figure() %>% ly_points(x = displ, y = hwy, color = 'red', data = mpg)
NSE と SE
{rbokeh} では、{ggplot2} のように NSE ( Non-standard evaluation )と SE ( Standard evaluation ) 用の関数を明示的に分けていない。そのため、一部のオプションは以下のようにSEでも動作する。
# scatter figure() %>% ly_points(x = 'displ', y = 'hwy', data = mpg) # 出力省略
が、 SE では動作しないものもあるため、現時点では NSE を使ったほうがよさそうだ。
# scatter figure() %>% ly_points(x = 'displ', y = 'hwy', color = 'class', data = mpg) # 以下にエラー nchar(opts[[fld]]) : 'nchar()' は文字ベクトルを要求します
平滑化 / 回帰直線の描画
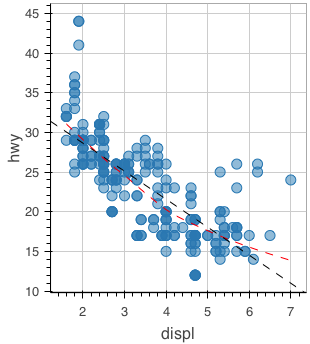
R のモデルを関数に渡すことで描画できる。lowess に対しては ly_lines を、lm に対しては ly_abline を用いる。この使い分けはちょっと覚えにくい。
figure() %>% ly_points(x = displ, y = hwy, data = mpg) %>% ly_lines(lowess(mpg$displ, mpg$hwy), color = "red", type = 2) %>% ly_abline(lm(hwy ~ displ, data = mpg), type = 2)
サブプロット ( facet ) の描画
{htmlwidgets} ブログ中の {rbokeh} 紹介記事 では、サブプロットを lapply + {pipeR} を使って描画する例が記載されている。自分は {dplyr} 好きなので {dplyr} でやりたい。
library(dplyr) mpg %>% dplyr::group_by(class) %>% dplyr::do(dummy = ly_points(figure(width = 200, height = 200), x = displ, y = hwy, data = ., size = 2)) %>% { as.list(.[['dummy']]) } %>% grid_plot(nrow = 3, ncol = 3, same_axes = TRUE )
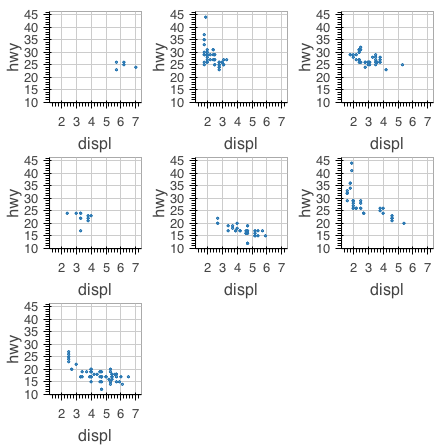
様々なプロット
以降、{rbokeh} で描画できるプロットを例示する。
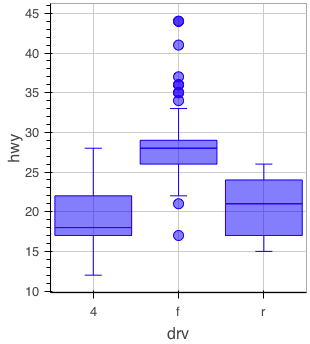
箱ひげ図
figure() %>% ly_boxplot(x = drv, y = hwy, data = mpg)
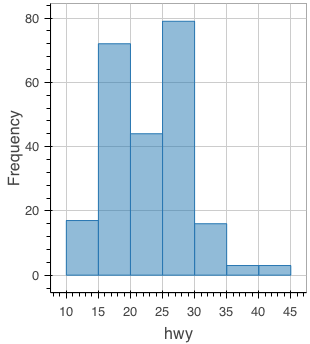
ヒストグラム
figure() %>% ly_hist(x = hwy, data = mpg)
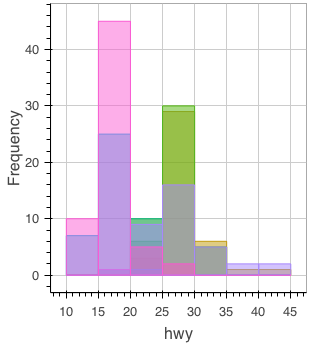
ヒストグラムをグループ別に描画するには少し手間が必要。グループ別に塗り分けるためのカラーパレットを用意し、Reduce で各グループ別のヒストグラムを追加する。
library(scales) colors <- scales::hue_pal()(length(levels(mpg$class))) colors <- setNames(colors, levels(mpg$class)) colors # 2seater compact midsize minivan pickup subcompact suv # "#F8766D" "#C49A00" "#53B400" "#00C094" "#00B6EB" "#A58AFF" "#FB61D7" ghist <- function(fig, group) { ly_hist(fig, x = hwy, data = dplyr::filter(mpg, class == group), breaks = seq(10, 45, 5), color = colors[[group]]) } Reduce(ghist, levels(mpg$class), figure())
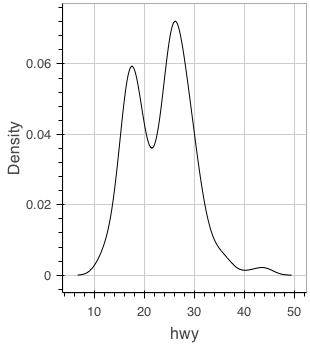
カーネル密度推定のプロット
グループ分けしたい場合はヒストグラム同様の処理が必要。
figure() %>% ly_density(x = hwy, data = mpg)
折れ線グラフ
df <- data.frame(x = c(1, 2, 3), y = c(4, 2, 5)) figure() %>% ly_lines(x = x, y = y, data = df)

地図
地図は {maps} パッケージのデータを利用してプロットができる。
library(maps) figure() %>% ly_map("state", color = "gray")

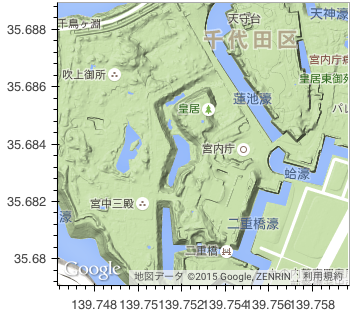
また、 Google Maps を利用することもできる。
gmap(lat = 35.684, lng = 139.753, zoom = 15, map_type = 'terrain')
現在 (簡単には) できないもの
R でよく使う種類のプロットを優先しているせいか、以下のような基本的なプロットにはまだ対応していないようだ。
| 種類 | GitHub Issue | 備考 |
|---|---|---|
| 棒グラフ | #8 | 頑張れば ly_hist で描ける。 |
| エリアプロット | #10 | |
| ヒートマップ | #86 | ly_hexbin はある。ly_image で近いことはできるが、軸のラベル付けが手間。 |
| 円/ドーナツ |
まとめ
{rbokeh}、上に挙げた基本的なプロットが使えるようになれば実用できそうな感じだ。Bokeh.js 側には主要な可視化が揃っているため、他のパッケージと比べると利用範囲が広いこと、見た目を複数言語で揃えられるのがメリットかなと思う。
Python geopandas + Bokeh で地理情報をプロットしたい
数日前、pandas を利用して地理情報をプロットするという非常によいエントリが翻訳されていた。
上のエントリ、前処理が手間に見えるが pd.read_html や .str アクセサを使えばもっと簡単に書けると思う、、、がそれは本題でない。
pandas で地理情報を扱う場合、geopandas という拡張パッケージを利用すると便利なため、その使い方を書きたい。また、処理を Python で完結させるため、QGIS ではなく Bokeh でプロットしたい。
geopandas のインストール
pip で。
$ pip install geopandas geopy
このエントリでは依存パッケージである shapely、geopy の機能も利用する。shapely は自動的にインストールされるはずだが、geopy については上のように別途インストールが必要。
地理情報の読み込み
以降の操作は IPython Notebook で行う。まずは必要なパッケージをインポートする。
%matplotlib inline import numpy as np import pandas as pd import geopandas as gpd
例として、東京都の地図を読み込んで利用したい。geopandas は、依存パッケージである fiona がサポートする地理情報ファイル形式を読み込むことができる。GitHub に 地球地図日本 のデータから作成した GeoJSON ファイルがあったため、こちらを利用させていただく。
ファイルの読み込みは、geopandas.read_file で行う。データは pandas.DataFrame を継承した geopandas.GeoDataFrame インスタンスとして読み込まれる。
df = gpd.read_file('tokyo.geojson') type(df) # geopandas.geodataframe.GeoDataFrame isinstance(df, pd.DataFrame) # True df.head() # area_en area_ja code geometry # 0 Tokubu 都区部 131211 POLYGON ((139.821051 35.815077, 139.821684 35.... # 1 Tokubu 都区部 131059 POLYGON ((139.760933 35.732206, 139.761002 35.... # 2 Tokubu 都区部 131016 POLYGON ((139.770135 35.705352, 139.770172 35.... # 3 Tokubu 都区部 131067 POLYGON ((139.809714 35.728135, 139.809705 35.... # 4 Tokubu 都区部 131091 (POLYGON ((139.719199 35.641847, 139.719346 35...
GeoDataFrame.plot を使うと、データを matplotlib を利用してプロットできる。
df.plot()

島嶼部があると細部がわからないため、都区部だけをフィルタしてプロットする。
df[df['area_en'] == 'Tokubu'].plot()

地理情報の処理
GeoDataFrame の各行はそれぞれひとつの領域 (ジオメトリオブジェクト) に対応している。各領域のジオメトリオブジェクトの実体は geometry キー中に shapely の Polygon もしくは MultiPolygon インスタンスとして保存されている。
Polygon には、領域各点の経度/緯度からなる tuple がリストとして保存されている。以下の処理で領域各点の経度/緯度をそれぞれ別のリストとして取り出すことができる。この処理は、続けて記載する Bokeh でのプロット時に必要になる。
# 1行目 (1領域目) を取得 s = df.iloc[0, :] s # area_en Tokubu # area_ja 都区部 # code 131211 # geometry POLYGON ((139.821051 35.815077, 139.821684 35.... # ward_en Adachi Ku # ward_ja 足立区 # Name: 0, dtype: object type(s) # <class 'pandas.core.series.Series'> # 領域のジオメトリオブジェクトの型を表示 type(s['geometry']) # <class 'shapely.geometry.polygon.Polygon'> # 領域の経度/緯度を取得 list(s['geometry'].exterior.coords[:5]) # [(139.821051, 35.815077), (139.821684, 35.814887), (139.822599, 35.814509), # (139.8229070000001, 35.81437), (139.822975, 35.814339)] # 経度、緯度別のリストに分割 lons, lats = zip(*list(s['geometry'].exterior.coords)) # 経度を表示 lons[:5] # (139.821051, 139.821684, 139.822599, 139.8229070000001, 139.822975, 139.822977)
地理情報のプロット
このデータを Bokeh でプロットしたい。Bokeh についてはこちらを。
Bokeh で地理情報をプロットするサンプルとして、ギャラリーにコロプレス図を描く例がある。Bokeh では 描画する各領域の x 座標 (経度)、y 座標 (緯度) をリストで指定する。各領域は複数座標からなるため、"各領域の x/y 座標のリスト" のリスト ( 例: [[境界1の点1のx座標, 境界1の点2のx座標, ...], [領域2の点1のx座標, ...], ...] ) を引数として用意する必要がある。
import shapely # "各領域の x 座標 (経度) のリスト" のリスト countries_xs = [] # "各領域の y 座標 (緯度) のリスト" のリスト countries_ys = [] for i, row in df.iterrows(): if row['area_en'] != 'Tokubu': # 都区部のみ描画 continue polygons = row['geometry'] if isinstance(polygons, shapely.geometry.multipolygon.MultiPolygon): # MultiPolygon に含まれる Polygon を取り出す polygons = [x for x in polygons] elif isinstance(polygons, shapely.geometry.polygon.Polygon): polygons = [polygons] else: raise ValueError for p in polygons: # 境界の各点を経度、緯度に分割 lons, lats = zip(*list(p.exterior.coords)) countries_xs.append(lons) countries_ys.append(lats)
作成したデータを Bokeh に渡す。出力先は IPython Notebook にした。
from bokeh import plotting # 出力先を指定 plotting.output_notebook() # figure を作成 p = plotting.figure(toolbar_location="left", plot_width=900, plot_height=700) # 各領域を描画 (塗り分けはしない) p.patches(countries_xs, countries_ys, fill_color='white', line_color="black", line_width=0.5) plotting.show(p)

Geocoding の利用
geopandas は geopy を利用して Geocoding (住所から緯度経度への変換) を行うための API geopandas.geocode.geocode を持っている。が、geopandas v0.1.1, geopy v1.10.0 では、これらのインターフェースに不整合があり そのままでは利用できない。詳細と回避策は以下 Stack Overflow を。
from geopandas.geocode import geocode # geopandas.geocode の API をそのまま使うとエラー geocode(['千代田区千代田1-1']) # AttributeError: 'module' object has no attribute 'MapQuest' # 回避策として geopy の以下プロパティを明示的に None にする import geopy geopy.geocoders.MapQuest = None geocode(['千代田区千代田1-1']) # address geometry # 0 1-1 Chiyoda, Chiyoda-ku, Tōkyō-to 100-0001, Japan POINT (139.7539454 35.6838012)
Geocoding を利用したデータの追加
上で作成した地図に、道路交通センサスにある各地点の交通量をプロットしたい。もっと細かいデータが見られるとよいのだが、公開情報ではこれくらいしかなさそうだ。以下のリンクから東京都の箇所別基本表 kasyo13.xls をダウンロードし、利用させていただく。
-
- 出典:「平成22年度 全国道路・街路交通情勢調査(道路交通センサス)一般交通量調査 集計表」(国土交通省)
pdf = pd.read_excel('kasyo13.xls', header=8, index_col=False) pdf.head() # (km) Unnamed: 1 Unnamed: 2 Unnamed: 3 Unnamed: 4 Unnamed: 5 # 0 13110100010 1 1010 東名高速道路 1 14110100090 # 1 13111010010 1 1101 中央自動車道富士吉田線 4 13200400170 # 2 13111010020 1 1101 中央自動車道富士吉田線 6 NaN # 3 13111010030 1 1101 中央自動車道富士吉田線 6 NaN # 4 13111010040 1 1101 中央自動車道富士吉田線 6 NaN # ヘッダがセル結合されており正しく読めないため、連番で上書き pdf.columns = range(len(pdf.columns)) # 計測地点の住所が入力されている列を表示 pdf.iloc[:, 28].head() # 0 東京~東名川崎 # 1 高井戸IC~世田谷区・東京都境間 # 2 高井戸IC~世田谷区・東京都境間 # 3 世田谷区・東京都境~調布IC間 # 4 高井戸IC~世田谷区・東京都境間 # Name: 28, dtype: object # NaN, 正しい住所でないデータをフィルタ pdf = pdf.dropna(subset=[28]) pdf = pdf[pdf[28].str.startswith('東京都')] # 計測地点住所と 24時間交通量の列のみにフィルタ pdf = pdf.iloc[:, [28, 36]] pdf.columns = ['計測地点', '交通量合計'] pdf.head() # 計測地点 交通量合計 # 22 東京都中央区八丁堀3丁目 107885 # 23 東京都中央区八丁堀1丁目 98482 # 24 東京都中央区新富1丁目 89089 # 25 東京都中央区銀座1丁目 94143 # 26 東京都中央区築地1丁目 93844
上のデータから、計測地点 カラムの住所をもとに、Geocoding で緯度経度を取得する。
codes = geocode(pdf['計測地点']) type(codes) # <class 'geopandas.geodataframe.GeoDataFrame'> codes.head() # address geometry # 22 3 Chome Hatchōbori, Chūō-ku, Tōkyō-to 104-0032... POINT (139.7756944 35.6755445) # 23 1 Chome Hatchōbori, Chūō-ku, Tōkyō-to 104-0032... POINT (139.7771192 35.6774165) # 24 1 Chome Shintomi, Chūō-ku, Tōkyō-to 104-0041, ... POINT (139.7725002 35.6729586) # 25 Ginza Itchome Station, 1 Chome-6 Ginza, Chūō-k... POINT (139.767045 35.67435) # 26 1 Chome Tsukiji, Chūō-ku, Tōkyō-to 104-0045, J... POINT (139.7716809 35.6702089)
取得したデータは上記のような GeoDataFrame となる。geometry カラムに 経度/緯度を示す Point インスタンスが含まれる。この座標と交通量を利用してバブルチャートが描きたい。Bokeh のギャラリーにバブルチャートをプロットする例があるため、それに倣うと、
pdf = pdf.join(codes) # 各点の x 座標 points_x = [] # 各点の y 座標 points_y = [] # 各点の円の半径 points_v = [] for i, row in pdf.iterrows(): points_x.append(row['geometry'].x) points_y.append(row['geometry'].y) points_v.append(row['交通量合計']) # 円の半径を適当に調整 points_v = np.sqrt(np.array(points_v)) / 50000 # Bokeh での描画 plotting.output_notebook() p = plotting.figure(toolbar_location="left", plot_width=900, plot_height=700) p.patches(countries_xs, countries_ys, fill_color='white', line_color="black", line_width=0.5) # バブルチャートを描画 p.scatter(points_x, points_y, radius=points_v, fill_alpha=0.1) plotting.show(p)

まとめ
geopandas を使って、地理情報に対する以下のような処理を行う方法を記載した。
- 各種地理情報フォーマットの読み込み/書き込み
- 各領域 (ジオメトリオブジェクト) からの座標 (緯度/経度) の取得
matplotlib,Bokehでのプロット- Geocoding
公式ドキュメント には、上以外に ジオメトリオブジェクトを操作 / 変換する例も記載されている。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (12件) を見る
Python pandas パフォーマンス維持のための 3 つの TIPS
pandas でそこそこ大きいデータを扱う場合、その処理速度が気になってくる。公式ドキュメントではパフォーマンス向上のために Cython や Numba を使う方法を記載している。
Enhancing Performance — pandas 0.16.2 documentation
が、軽く試したいだけなのに わざわざ Cythonや Numba を使うのは手間だし、かといってあまりに遅いのも嫌だ。そんなとき、pandas 本来のパフォーマンスをできるだけ維持するためのポイントを整理したい。
pandas に限らず、パフォーマンス改善の際にはボトルネックの箇所によってとるべき対策は異なる。pandas では速度向上/エッジケース処理のために データの型や条件によって内部で処理を細かく分けており、常にこうすれば速くなる! という方法を出すのは難しい。以下はこの前提のうえで、内部実装からみて まあほとんどの場合あてはまるだろう、、、という内容をまとめる。
環境構築
環境は EC2 の c4.xlarge インスタンス上に作成する。パフォーマンスを重視する場合、環境構築の時点で以下を行っておく。
numpyを各種 数値計算ライブラリBLAS,LAPACK,ATLASとリンクさせる。numexpr,bottleneckをインストールする。
EC2 の Amazon Linux であれば以下のコマンドでできる。
$ sudo yum -y install blas-devel lapack-devel atlas-devel $ sudo python -m pip install numpy $ sudo python -m pip install numexpr bottleneck
環境構築が正しくできたかを確認する。以降の処理は IPython Notebook 上で行う。
import numpy as np import numpy.distutils.system_info as sysinfo sysinfo.get_info('blas') # {'libraries': ['blas'], 'library_dirs': ['/usr/lib64'], 'language': 'f77'} sysinfo.get_info('lapack') # {'libraries': ['lapack'], 'library_dirs': ['/usr/lib64'], 'language': 'f77'} sysinfo.get_info('atlas') # {'define_macros': [('ATLAS_INFO', '"\\"3.8.4\\""')], # 'include_dirs': ['/usr/include'], # 'language': 'f77', # 'libraries': ['lapack', 'f77blas', 'cblas', 'atlas'], # 'library_dirs': ['/usr/lib64/atlas']} import pandas as pd pd.show_versions() # ... # pandas: 0.16.2 # ... # bottleneck: 1.0.0 # ... # numexpr: 2.4.3 # ...
サンプルデータの作成
サンプルデータ作成には pandas がテスト用に用意しているメソッドを使う。もっとも、似たようなデータが作れれば方法はなんでもいい。
pd.util.testing.rands_array: 指定した文字数 / 要素数のランダムな文字列のnp.ndarrayを作成。pd.util.testing.choice:np.ndarrayからsize個をサンプリング。
# 2 文字 / 3 要素の array を作成 pd.util.testing.rands_array(2, 3) # array(['Xn', 'ZC', 'zj'], dtype=object) # array から 5 回サンプリング pd.util.testing.choice(np.array(['a', 'b']), size=5) # array(['a', 'b', 'b', 'b', 'a'], dtype='|S1')
サンプルデータとして 100 万レコード / 3 列のデータを用意した。
np.random.seed(0) # レコード数 N = 1000000 chars1 = pd.util.testing.rands_array(5, 100) chars2 = pd.util.testing.rands_array(5, 10000) df = pd.DataFrame({'x': np.random.randn(N), 'y': pd.util.testing.choice(chars1, size=N), 'z': pd.util.testing.choice(chars2, size=N)}) df.shape # (1000000, 3) df.head()
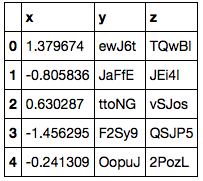
y 列は 100 通りの値を、z 列は 10000 通りの値をとる。
chars1[:10] # array(['SV1ad', '7dNjt', 'vYKxg', 'yym6b', 'MNxUy', 'rLzni', 'juZqZ', # 'fpVas', 'JyXZD', 'ttoNG'], dtype=object) len(np.unique(chars1)) # 100 len(np.unique(chars2)) # 10000
1. 行に対するループ / DataFrame.apply は 使わない
pandas.DataFrame は列ごとに異なる型を持つことができる。DataFrame は内部的に 同じ型の列をまとめて np.ndarray として保持している。列ごとに連続したデータを持つことになるため、そもそも行に対するループには向かない。また、DataFrame.iterrows でのループの際には 異なる型を持つ列の値を Series として取り出すため、そのインスタンス化にも時間がかかる。
また 行ごと / 列ごとに 関数を適用するメソッドに DataFrame.apply があるが、このメソッドでは Python の関数を繰り返し呼び出すためのコストがかかる。apply は利便性を重視したメソッドのため、パフォーマンスを気にする場合は避けたほうがよい。
参考 Python pandas データのイテレーションと関数適用、pipe - StatsFragments
上記ふたつの処理の組み合わせである、各行への関数適用 DataFrame.apply(axis=1) について処理時間を %timeit で計測する。まずは 単純に y 列と z 列の値を文字列結合する場合。
def f1(s): return s['y'] + s['z'] %timeit df.apply(f1, axis=1) # 1 loops, best of 3: 13.7 s per loop
この処理は、Series として列を取り出し ベクトルとして行うほうが格段に速い。
%timeit df['y'] + df['z'] # 10 loops, best of 3: 74.9 ms per loop
ただ、関数中に条件分岐を含む場合など、そのままベクトル化できない場合もある。x 列の値によって 結合の順序を変える例を考える。
def f2_1(s): if s['x'] > 0: return s['y'] + s['z'] else: return s['z'] + s['y'] %timeit df.apply(f2_1, axis=1) # 1 loops, best of 3: 16 s per loop
こういった場合は np.vectorize で関数をベクトル化し、引数として各列を Series (もしくは np.ndarray ) として渡したほうが apply よりは速い。
def f2_2(x, y, z): if x > 0: return y + z else: return z + y %timeit pd.Series(np.vectorize(f2_2)(df['x'], df['y'], df['z']), index=df.index) # 1 loops, best of 3: 334 ms per loop
補足 numpy のドキュメント に記載されているとおり、np.vectorize もパフォーマンスを最重視した方法ではない。さらに速くしたい場合は個別にベクトル化した関数を用意する。参考として、Cython で書いた場合の処理時間は以下となる。np.vectorize の 4-5 倍は速いようだ。
%load_ext cython
%%cython import numpy as np from numpy cimport ndarray def f2_3(ndarray[double, ndim=1] x, ndarray[object, ndim=1] y, ndarray[object, ndim=1] z): cdef: int i, length = len(x) double xval object yval, zval ndarray[object, ndim=1] result = np.empty(length, dtype=object) for i in range(length): xval = x[i] yval = y[i] zval = z[i] if xval > 0: result[i] = yval + zval else: result[i] = zval + yval return result
%timeit pd.Series(f2_3(df['x'].values, df['y'].values, df['z'].values), index=df.index) # 10 loops, best of 3: 67.5 ms per loop
補足 Cython については最近以下の書籍が出ていた。翻訳はわからないが、原著はわかりやすく要点がまとまっていたのでおすすめ。

- 作者: Kurt W. Smith,中田秀基,長尾高弘
- 出版社/メーカー: オライリージャパン
- 発売日: 2015/06/19
- メディア: 大型本
- この商品を含むブログ (3件) を見る
2. object 型は使わない
pandas には、グループ化 ( DataFrame.groupby ) や 結合 ( DataFrame.merge ) など、データの値をキーにして行われる処理がある。こういった操作を行う場合は object 型を避けたほうが速くなることが多い。
pandas では文字列を含む列は object 型になることに注意する。numpy の文字列型とならない理由は簡単にいうと欠損値 ( NaN ) の処理のため。例えば文字列で保存された商品コードをキーにしてグループ化 / 結合する場合など、そのままでは object 型として処理されてしまう。
df.dtypes # x float64 # y object # z object # dtype: object
object 型の列をキーとしてグループ化 / 集約したときの処理時間は以下。
%timeit df.groupby('y').mean() # 10 loops, best of 3: 52.3 ms per loop
処理速度をあげるためには、キーとなる値を カテゴリ型 ( pd.Categorical ) に変換しておくとよい。これは R でいう factor にあたる型。
df['y'] = df['y'].astype('category') %timeit df.groupby('y').mean() # 100 loops, best of 3: 6.89 ms per loop
カテゴリ型では、カテゴリのラベル (pd.Categorical.categories ) と ラベルの位置 (pd.Categorical.codes ) を分けて扱う。内部処理は int 型で保存されたラベルの位置について行われるため、object に対する処理と比較すると速くなる。
# カテゴリ型の内部データを表示 df['y'].values # [ewJ6t, JaFfE, ttoNG, F2Sy9, OopuJ, ..., dS9oG, juZqZ, 5gvFn, 9brWJ, 9fxRG] # Length: 1000000 # Categories (100, object): [0dmK0, 1DdJN, 1IZE1, 1m5Qu, ..., x7c5I, ydsVd, yym6b, zYGBj] # カテゴリのラベル df['y'].cat.categories # Index([u'0dmK0', u'1DdJN', u'1IZE1', u'1m5Qu', u'219tH', u'2aMtU', u'403al', # ... # u'yym6b', u'zYGBj'], # dtype='object') # カテゴリのラベルの位置 df['y'].cat.codes.head() # 0 66 # 1 30 # 2 90 # 3 23 # 4 38 # dtype: int8
列がとりうる値の数が増えた場合も、カテゴリ型にしたほうが速い。
# object 型 %timeit df.groupby('z').mean() # 10 loops, best of 3: 78.4 ms per loop # カテゴリ型 df['z'] = df['z'].astype('category') %timeit df.groupby('z').mean() # 100 loops, best of 3: 17.9 ms per loop
列の型 dtype 以外に処理に影響を与えうるのは、欠損値 ( NaN ) の有無 (無いほうが速い)、object 型内での型の混在 (文字列と数値が混ざっている) など。
3. ユニークでない / ソートされていない index は使わない
最後。index についても、上のとおり object 型は避ける。また、特に理由がない場合 値を ユニークにし、かつソートしておく。上記のサンプルデータ作成時点では、特に index を指定していないため int 型の index が昇順で振られている。
df.index # Int64Index([ 0, 1, 2, 3, 4, 5, 6, 7, # 8, 9, # ... # 999990, 999991, 999992, 999993, 999994, 999995, 999996, 999997, # 999998, 999999], # dtype='int64', length=1000000)
この index で 結合 ( DataFrame.join ) した場合の処理時間は以下 (自身との結合のため意味のない処理だが)。
%timeit df.join(df, rsuffix='right_') # 10 loops, best of 3: 70.6 ms per loop
このとき、 DataFrameに対して前処理 (スライス / サンプリングなど) を行うと、処理に応じて index も並びかわる。例えば サンプリング ( DataFrame.sample ) を行った場合、
df2 = df.sample(n=len(df)) df2.shape # (1000000, 3) df2.index # Int64Index([372149, 955220, 320961, 244572, 254656, 74192, 143279, 246307, # 764579, 96091, # ... # 827030, 681861, 492455, 894210, 758153, 327280, 245717, 952466, # 26440, 532620], # dtype='int64', length=1000000)
このような DataFrame で結合を行うと、上と比較して処理時間が長くなっていることがわかる。
%timeit df2.join(df, rsuffix='right_') # 1 loops, best of 3: 185 ms per loop
index の値がユニークかどうか / ソートされているかは 以下のプロパティで確認できる。このプロパティによって一部の内部処理が分かれる。
# ユニークかどうか df.index.is_unique # True # 昇順でソートされているか df.index.is_monotonic_increasing # True df2.index.is_unique # True df2.index.is_monotonic_increasing # False
まとめ
pandas にその本来のパフォーマンスを発揮させるためには、以下 3 点の処理を避けるとよい。
- 行に対するループ /
DataFrame.applyは 使わない object型は使わない- ユニークでない / ソートされていない
indexは使わない
上記を行った上で処理速度に不満がある場合は Cython や Numba で高速化する。もしくは、Dask を使って並列化を考える。
9/24 追記 Dask についてはこちらを。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (10件) を見る
Chainer で Deep Learning: Bokeh で Live Monitoring したい
概要
Deep Learning の学習には時間がかかるため、進捗が都度 確認できるとうれしい。その際、テキストのログ出力では味気ないので、リアルタイムでプロットを眺めたい。
いくつかの Deep Learning パッケージではそういった機能 (Live Monitoring) が提供されている。
- Undefined Intelligence: Monitoring Experiments in Pylearn2
- Live plotting — Blocks 0.0.1 documentation
- fchollet/hualos · GitHub
同じことを Chainer でやりたい。自分は EC2 を使うことが多いので、リモート環境でも利用できるものがいい。そのため、ここでは Bokeh を使うことにした。
Bokeh とは
Bokeh とは、D3.js を利用したブラウザベースのインタラクティブな可視化を実現するパッケージ。どんなものかは公式の Gallery が充実しているのでそちらを。
補足 R 用のパッケージ {rbokeh} もある。
インストール
pip で。
$ pip install bokeh
準備
環境は EC2 上に作成する。Bokeh からは、ファイル、IPython、Bokeh 組み込みのWebサーバ ( bokeh-server ) 上の画面 の 3種類に対して出力することができるが、今回は Ipython を使うことにする。
補足 もっとも、常に IPython を使うわけではないため、 bokeh-server 上でも描画できるようにしたい。少し試したが、EC2 上に Bokeh を置くと bokeh-server の画面は開くが個々のプロットが表示できなかったため諦めた (ローカルでは問題ない)。できた方いたらやり方教えてください。
bokeh-server はプロットの描画画面だけでなく、動的なデータ更新のためのAPIも提供している。この例では 学習の進捗をリアルタイムで描画するために bokeh-server の機能を利用する。bokeh-server はシェルから以下のコマンドで起動できる。このとき、外部からの接続を受け入れるには自身のホスト名 / IP を引数 ip として指定する。
$ bokeh-server --ip=ec2-xxx-xxx-xxx-xxx.ap-northeast-1.compute.amazonaws.com
補足 EC2 では (特に設定していなければ) グローバル/プライベートの IP が異なるため、IP 指定では正しくデータが更新されないようだ。そのため、パブリックDNSを指定した。
Live Monitoring
以降は IPython Notebook から。Live Monitoring のためのプロットを行うクラスを定義する。
import numpy as np import chainer import bokeh.plotting as plotting from bokeh.models import GlyphRenderer class LiveMonitor(object): def __init__(self, server='chainer', url='http://localhost:5006/', **kwargs): # 出力先に IPython Notebook を指定 plotting.output_notebook(url=url) # トレーニング/テストデータの loss, accuracy を描画する figure を定義 self.train_loss = self._initialize_figure(title='Train loss', color='#FF0000', **kwargs) self.train_acc = self._initialize_figure(title='Train accuracy', color='#0000FF', **kwargs) self.test_loss = self._initialize_figure(title='Test loss', color='#FF0000', **kwargs) self.test_acc = self._initialize_figure(title='Test accuracy', color='#0000FF', **kwargs) # figure を 2 x 2 のグリッド (サブプロット) として配置 self.grid = plotting.gridplot([[self.train_loss, self.test_loss], [self.train_acc, self.test_acc]]) # グリッドを描画 plotting.show(self.grid) def _initialize_figure(self, color=None, line_width=2, title=None, title_text_font_size='9pt', plot_width=380, plot_height=280): """ figure の初期化用のメソッド""" figure = plotting.figure(title=title, title_text_font_size=title_text_font_size, plot_width=plot_width, plot_height=plot_height) # 空のデータで折れ線グラフを作成 x = np.array([]) y = np.array([]) figure.line(x, y, color=color, line_width=line_width) return figure def update(self, train_loss=None, train_accuracy=None, test_loss=None, test_accuracy=None): """ プロットを更新するためのメソッド 指定したキーワード引数に対応する figure が更新される """ self._maybe_update(self.train_loss, train_loss) self._maybe_update(self.train_acc, train_accuracy) self._maybe_update(self.test_loss, test_loss) self._maybe_update(self.test_acc, test_accuracy) def _maybe_update(self, figure, value): """ figure の値を更新するメソッド""" if value is not None: # Variable から np.array に戻す if isinstance(value, chainer.Variable): value = chainer.cuda.to_cpu(value.data) # figure が利用している data_source を取得 renderer = figure.select(dict(type=GlyphRenderer)) ds = renderer[0].data_source # data_source 中の値を更新 y = np.append(ds.data['y'], value) ds.data['y'] = y ds.data['x'] = np.arange(len(y)) # session へ返す (とプロットが更新される) plotting.cursession().store_objects(ds)
このクラスをインスタンスにする。URL としては、bokeh-server の起動時に指定したものを 以下 URL の形式で指定する。
monitor = LiveMonitor(url="http://ec2-xxx-xxx-xxx-xxx.ap-northeast-1.compute.amazonaws.com:5006/")
あとは 学習 / テスト中に、monitor.update に適当な引数を渡せばよい。chainer/examples/mnist/train_mnist.py を例にすると、
# ... 略 optimizer.zero_grads() loss, acc = forward(x_batch, y_batch) loss.backward() optimizer.update() monitor.update(train_loss=loss, train_accuracy=acc) # ... 略 loss, acc = forward(x_batch, y_batch, train=False) monitor.update(test_loss=loss, test_accuracy=acc)
IPython 上 ( LiveMonitor インスタンスを作成した直後) のセルに、以下のようなプロットが表示される。プロットは update が呼ばれるたびに更新される。
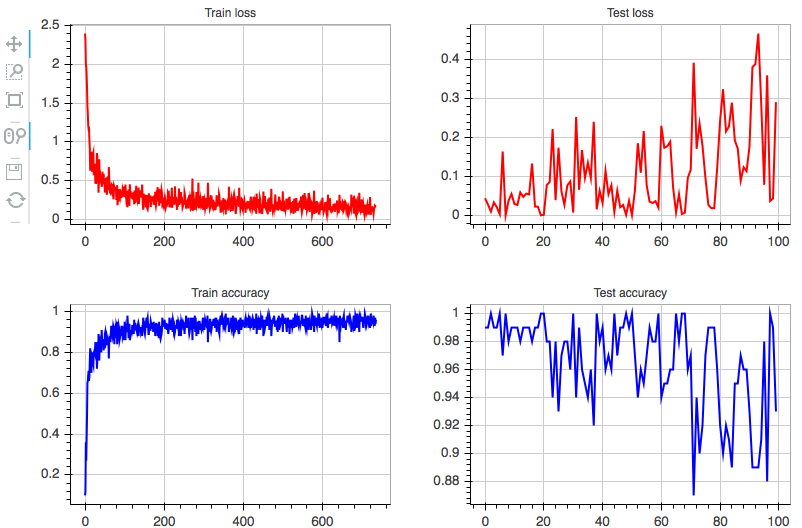
また、各プロットはインタラクティブにズーム/パンといった操作ができる (左上)。
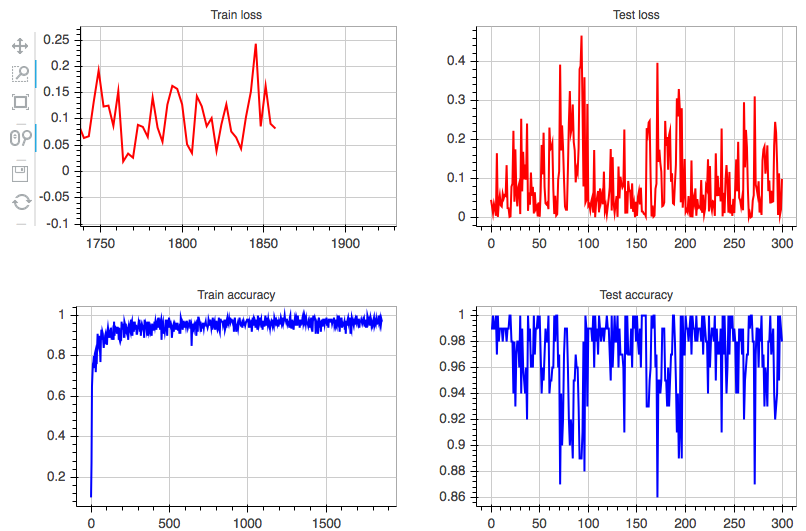
まとめ
これでリアルタイムに進捗を眺めて楽しむことができる。

- 作者: 岡谷貴之
- 出版社/メーカー: 講談社
- 発売日: 2015/04/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (4件) を見る
Chainer で Deep Learning: model zoo で R-CNN やりたい
ニューラルネットワークを使ったオブジェクト検出の手法に R-CNN (Regions with CNN) というものがある。簡単にいうと、R-CNN は以下のような処理を行う。
- 入力画像中からオブジェクトらしい領域を検出し切り出す。
- 各領域を CNN (畳み込みニューラルネットワーク) にかける。
- 2での特徴量を用いて オブジェクトかどうかをSVMで判別する。
R-CNN については 論文著者の方が Caffe (Matlab) での実装 (やその改良版) を公開している。
が、自分は Matlab のライセンスを持っていないので Python でやりたい。Python でやるなら 今 流行りの Chainer を使ってみたい。その試行の記録。
準備
とりあえず論文の再現は目指さず、R-CNN っぽい処理 (オブジェクト検出 -> CNN) を Python から回せるようにしたい。
まずは オブジェクト検出について調べてみると、上述の論文 & 実装で利用されている Selective Search という手法は ( Python の wrapper はあるが) Matlab なしでは利用できないようだ。Python から使える代替手法として GOP (Geodesic Object Proposals) という方法を見つけた。著者がパッケージも作っているのだが、どうもオブジェクト検出の方法がよくわからない、、、が矩形を切り取ることはできそうだ。
ということで今回は以下の処理が流せるようにしたい。モデルの修正やファインチューニングはせず、 caffenet をそのまま使う(済まぬ、、)。環境は EC2 の GPU インスタンス上に作成する。
- オブジェクトっぽい矩形をクロップ (Geodesic K-means)
- クロップした領域を CNN で判別 (
Chainermodel zoo で caffenet を利用) - 判別した結果を描画
都合上、 2 -> 1 -> 3 の順で記載する。
Chainer model zoo の利用
本日時点で PyPI にリリースされている v1.0.1 標準にはない機能のため、GitHub からダウンロードする。自分が利用したのは 本日時点 5222fe572b のリビジョン。
model zoo のダウンロード
chainer/examples/modelzoo から以下を実行してモデル と mean ファイルをダウンロードする。
$ python download_model.py caffenet $ python download_mean_file.py
また、データセットの ID / ラベルを含む synset_words.txt を別途ダウンロードしておく。これは以下のファイルに含まれている。
$ wget http://dl.caffe.berkeleyvision.org/caffe_ilsvrc12.tar.gz $ mkdir ilsvrc $ tar zxvf caffe_ilsvrc12.tar.gz -C ilsvrc
model zoo の読み込み
以降は IPython Notebook で。最初に必要なパッケージをロードする。
%matplotlib inline import os import sys import numpy as np import chainer from chainer import cuda import chainer.functions as F from chainer.functions import caffe import matplotlib.pyplot as plt
次に、先ほどダウンロードした caffenet のパスを指定し、Chainer のモデルとして読み込む。
また、synset_words.txt を np.array として読み込む。
base = os.path.join('chainer', 'examples', 'modelzoo') model = os.path.join(base, 'bvlc_reference_caffenet.caffemodel') func = caffe.CaffeFunction(model) cuda.init(0) func.to_gpu() synset_words = np.array([line[:-1] for line in open(os.path.join(base, 'ilsvrc', 'synset_words.txt'), 'r')]) synset_words[:10] # array(['n01440764 tench, Tinca tinca', # 'n01443537 goldfish, Carassius auratus', # 'n01484850 great white shark, white shark, man-eater, man-eating shark, Carcharodon carcharias', # 'n01491361 tiger shark, Galeocerdo cuvieri', # 'n01494475 hammerhead, hammerhead shark', # 'n01496331 electric ray, crampfish, numbfish, torpedo', # 'n01498041 stingray', 'n01514668 cock', 'n01514859 hen', # 'n01518878 ostrich, Struthio camelus'], # dtype='|S131')
続けて、画像に対する caffenet の判別結果を返す関数を準備する。画像のリストを入力とし、一度に入力された画像群に対してはバッチで処理するようにした。
in_size = 224 cropwidth = 256 - in_size start = cropwidth // 2 stop = start + in_size # caffenet meannpy = os.path.join(base, 'ilsvrc_2012_mean.npy') mean_image = np.load(meannpy) mean_image = mean_image[:, start:stop, start:stop].copy() def predict(images): """画像のリストに対する判別を行う""" global mean_image, in_size, cropwidth, start, stop def swap(x): x = np.array(x)[:, :, ::-1] x = np.swapaxes(x, 0, 2) x = np.swapaxes(x, 1, 2) return x if not isinstance(images, list): images = [images] batch_size = len(images) x_data = np.ndarray((batch_size, 3, in_size, in_size), dtype=np.float32) for i, image in enumerate(images): image = swap(image) image = image[:, start:stop, start:stop].copy().astype(np.float32) image -= mean_image x_data[i] = image x_data = cuda.to_gpu(x_data) x = chainer.Variable(x_data, volatile=True) y, = func(inputs={'data': x}, outputs=['fc8'], train=False) y.data = cuda.to_cpu(y.data) indexer = y.data.argmax(axis=1) return synset_words[indexer], y.data.max(axis=1)
動作確認
自分は ImageNet へのアクセス権を持っていないため、wikimedia で適当な画像をみつくろって試す。そのため、URL を指定して画像をダウンロードする関数、画像を caffenet へ入力するためにリサイズする関数を作成する。
def get_image(url): """URLから画像をダウンロード""" import urllib import StringIO import PIL.Image as Image return Image.open(StringIO.StringIO(urllib.urlopen(url).read())).convert("RGB") def resize(image): """画像をリサイズ""" import PIL.Image as Image return image.resize((256, 256), Image.ANTIALIAS)
いくつか結果を添付する。
url1 = "https://upload.wikimedia.org/wikipedia/commons/0/0e/BIRD_PARK_8_0189.jpg"
img = get_image(url1)
plt.imshow(img)

img = resize(img) plt.imshow(img)

predict(img) # (array(['n01806143 peacock'], # dtype='|S131'), array([ 26.1235218], dtype=float32))
別の画像。
url2 = "https://upload.wikimedia.org/wikipedia/commons/thumb/f/f4/Ostrich_Ngorongoro_05.jpg/640px-Ostrich_Ngorongoro_05.jpg"
img = get_image(url2)
plt.imshow(img)

img = resize(img) plt.imshow(img)

predict(img) # (array(['n01518878 ostrich, Struthio camelus'], # dtype='|S131'), array([ 22.44941521], dtype=float32))
定量的に測った訳ではないが、魚類は正解しにくい気がする。
url3 = "https://upload.wikimedia.org/wikipedia/commons/thumb/f/ff/Peixe010eue.jpg/640px-Peixe010eue.jpg"
img = get_image(url3)
plt.imshow(img)

img = resize(img) plt.imshow(img)

predict(img) # (array(['n01443537 goldfish, Carassius auratus'], # dtype='|S131'), array([ 24.29219818], dtype=float32))
複数画像を処理する場合は、画像のリストを渡す。
images = [resize(get_image(url1)), resize(get_image(url2)), resize(get_image(url3))] labels, scores = predict(images) labels # array(['n01806143 peacock', 'n01518878 ostrich, Struthio camelus', # 'n01443537 goldfish, Carassius auratus'], # dtype='|S131') scores # array([ 26.1235218 , 22.44941521, 24.29219818], dtype=float32)
矩形のクロップ (Geodesic K-means)
Geodesic Object Proposals 著者のサイトから Code, Data をダウンロードする。README が少しわかりにくいが、以下の方法でインストールできた。
# eigen のダウンロード $ wget http://bitbucket.org/eigen/eigen/get/3.2.5.zip $ unzip 3.2.5.zip # GOP のダウンロード $ wget http://googledrive.com/host/0B6qziMs8hVGieFg0UzE0WmZaOW8/code/gop_1.3.zip $ unzip gop_1.3.zip # GOP の解凍ディレクトリ/external/eigen に Eigen ディレクトリをコピー $ mkdir gop_1.3/external/eigen $ cp -r eigen-eigen-bdd17ee3b1b3/Eigen/ gop_1.3/external/eigen/ $ ls gop_1.3/external/eigen # Eigen $ cd gop_1.3/build # -DUSE_PYTHON で Python 2.x とのリンクを指定 $ cmake .. -DCMAKE_BUILD_TYPE=Release -DUSE_PYTHON=2 $ sudo make install
また、ダウンロードした Data は gop_1.3/data ディレクトリの中に入れておく。
$ unzip gop_data.zip
GOP を IPython Notebook から使ってみる。GOP は site-packages にインストールされないため、ロードするには sys に対して path を追加してやる必要がある。矩形を選択する方法のうち、今のところ動かせたのは Geodesic K-means のみなのでそれを使う。GOP では Geodesic K-means の後にオブジェクト検出を行うようなのだが、その結果を利用して矩形選択する方法はまだわかってない (そのため、オブジェクト検出はまだできていないという理解だが Proposal の処理を追っていないので自信はない)。
sys.path.append('./gop_1.3/src/') from gop import * from util import * prop = proposals.Proposal(setupLearned(200, 4, 0.8)) detector = contour.MultiScaleStructuredForest() detector.load("./gop_1.3/data/sf.dat") def get_boundaries(image): # 画像データを直接入力とする方法が不明のため、一時ファイルに保存 img.save('tmp.png') s = segmentation.geodesicKMeans(imgproc.imread('tmp.png'), detector, 100) b = prop.propose(s) boxes = s.maskToBox(b) return boxes
以下の画像を使って確認してみる。
url = "https://upload.wikimedia.org/wikipedia/commons/8/8f/Dogs_playing_on_the_beach_in_the_sand.jpg"
img = get_image(url)
plt.imshow(img)

fig, ax = plt.subplots(1, 1) ax.imshow(img) boxes = get_boundaries(img) def plot_boxes(ax, boxes, labels=None): """ax への矩形とラベルの追加""" if labels is None: labels = [None] * len(boxes) history = [] from matplotlib.patches import FancyBboxPatch for box, label in zip(boxes, labels): coords = (box[0], box[1]) b = FancyBboxPatch(coords, box[2]-box[0], box[3]-box[1], boxstyle="square,pad=0.", ec="b", fc="none", lw=0.5) mindist = 100000 if len(history) > 0: mindist = min([sum((box - h) ** 2) for h in history]) # ほぼ重なる矩形は描画しない if mindist > 30000: if label is not None: ax.text(coords[0], coords[1], label, color='b') ax.add_patch(b) history.append(box) plot_boxes(ax, boxes)

クロップされた画像はこんな感じ。
cropped = img.crop(boxes[13])
plt.imshow(cropped)

リサイズして caffenet にかける。犬という意味では近いが、品種は当たってない。
predict(resize(cropped)) # (array(['n02105412 kelpie'], # dtype='|S131'), array([ 10.38668251], dtype=float32))
各領域への CNN の適用
Geodesic K-means で抽出された各領域をクロップした画像のリストを作り、caffenet に渡す。
images = [resize(img.crop(box)) for box in boxes] labels = [] scores = [] nunit = len(images) / 5 unit = len(images) / nunit for i in range(nunit+1): l, s = predict(images[i*unit:min(i*unit+unit, len(images))]) labels.extend(l.tolist()) scores.extend(s.tolist()) import pandas as pd df = pd.DataFrame({'Labels': labels, 'Scores': scores}) df = df.sort('Scores', ascending=False) df.head()

判別結果のうちスコアがよい部分をラベル付きで描画する。やはりうまくオブジェクトが切り出せていない感じがする。
fig, ax = plt.subplots(1, 1) ax.imshow(img) heads = df.head(n=50) labels = [l.split(' ', 1)[1] for l in heads['Labels'].tolist()] plot_boxes(ax, boxes[heads.index], labels=labels)

まとめ
今回、Python から以下の処理をまわすことはできた。
- オブジェクトっぽい矩形をクロップ
- クロップした領域を CNN で判別
- 判別した結果を描画
が、判別結果自体は微妙な感じなので、以下2つについてはもう少し調べたい。
同日追記
パラメータ調整したら矩形抽出は多少ましになったような、、(画像末端まで選択されてしまうことが減っている)。元論文では候補を 2000 個抽出しているため、同程度以上にしておけばよさそう。


- 作者: 岡谷貴之
- 出版社/メーカー: 講談社
- 発売日: 2015/04/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (4件) を見る
Python pandas データのイテレーションと関数適用、pipe
pandas ではデータを 列 や 表形式のデータ構造として扱うが、これらのデータから順番に値を取得 (イテレーション) して何か操作をしたい / また 何らかの関数を適用したい、ということがよくある。このエントリでは以下の 3 つについて整理したい。
- イテレーション
- 関数適用
pipe(0.16.2 で追加)
それぞれ、Series、DataFrame、GroupBy (DataFrame.groupbyしたデータ) で可能な操作が異なるため、順に記載する。
まずは必要なパッケージを import する。
import numpy as np import pandas as pd
イテレーション
Series
Series は以下 2つのイテレーション用メソッドを持つ。各メソッドの挙動は以下のようになる。
図で表すとこんな感じ。矢印が処理の方向、枠内が 1 処理単位。
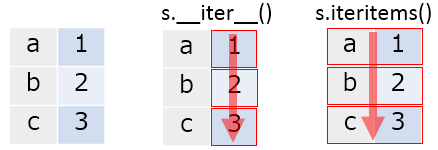
s = pd.Series([1, 2, 3], index=['a', 'b', 'c']) for v in s: print(v) # 1 # 2 # 3 for i, v in s.iteritems(): print(i) print(v) print('') # a # 1 # # b # 2 # # c # 3
DataFrame
DataFrame は以下 3つのイテレーション用メソッドを持つ。同様に挙動を示す。
__iter__:DataFrameの列名 (columns) のみをイテレーションiteritems:DataFrameの列名と 列の値 (Series) からなるtupleをイテレーションiterrows:DataFrameの行名と 行の値 (Series) からなるtupleをイテレーション

df = pd.DataFrame({'A': [1, 2, 3], 'B': [4, 5, 6]}, index=['a', 'b', 'c'])
df
# A B
# a 1 4
# b 2 5
# c 3 6
for col in df:
print(col)
# A
# B
for key, column in df.iteritems():
print(key)
print(column)
print('')
# A
# a 1
# b 2
# c 3
# Name: A, dtype: int64
#
# B
# a 4
# b 5
# c 6
# Name: B, dtype: int64
for key, row in df.iterrows():
print(key)
print(row)
print('')
# a
# A 1
# B 4
# Name: a, dtype: int64
#
# b
# A 2
# B 5
# Name: b, dtype: int64
#
# c
# A 3
# B 6
# Name: c, dtype: int64
GroupBy
__iter__:GroupByのグループ名と グループ (DataFrameもしくはSeries) からなるtupleをイテレーション

df = pd.DataFrame({'group': ['g1', 'g2', 'g1', 'g2'],
'A': [1, 2, 3, 4], 'B': [5, 6, 7, 8]},
columns=['group', 'A', 'B'])
df
# group A B
# 0 g1 1 5
# 1 g2 2 6
# 2 g1 3 7
# 3 g2 4 8
grouped = df.groupby('group')
for name, group in grouped:
print(name)
print(group)
print('')
# g1
# group A B
# 0 g1 1 5
# 2 g1 3 7
#
# g2
# group A B
# 1 g2 2 6
# 3 g2 4 8
関数適用
Series
Series の各値に対して 関数を適用する方法は以下の 2 つ。挙動はほぼ一緒だが、関数適用する場合は apply を使ったほうが意図が明確だと思う
Series.apply:Seriesの各値に対して関数を適用。Series.map:Seriesの各値を、引数を用いてマッピングする。引数として、dictやSeriesも取れる。

s = pd.Series([1, 2, 3], index=['a', 'b', 'c']) s.apply(lambda x: x * 2) # a 2 # b 4 # c 6 # dtype: int64 # apply の引数には、Series の値そのものが渡っている s.apply(type) # a <type 'numpy.int64'> # b <type 'numpy.int64'> # c <type 'numpy.int64'> # dtype: object # 関数が複数の返り値を返す場合、結果は tuple の Series になる s.apply(lambda x: (x, x * 2)) # a (1, 2) # b (2, 4) # c (3, 6) # dtype: object # 結果を DataFrame にしたい場合は、返り値を Series として返す s.apply(lambda x: pd.Series([x, x * 2], index=['col1', 'col2'])) # col1 col2 # a 1 2 # b 2 4 # c 3 6 # map の挙動は apply とほぼ同じ (map では結果を DataFrame にすることはできない) s.map(lambda x: x * 2) # a 2 # b 4 # c 6 # dtype: int64 s.map(type) # a <type 'numpy.int64'> # b <type 'numpy.int64'> # c <type 'numpy.int64'> # dtype: object # map は 関数以外に、 mapping 用の dict や Series を引数として取れる s.map(pd.Series(['x', 'y', 'z'], index=[1, 2, 3])) # a x # b y # c z # dtype: object
DataFrame
DataFrame に対して 関数を適用する方法は以下の 2 つ。
DataFrame.apply:DataFrameの各列もしくは各行に対して関数を適用。行 / 列の指定はaxisキーワードで行う。DataFrame.applymap:DataFrameの各値に対して関数を適用。
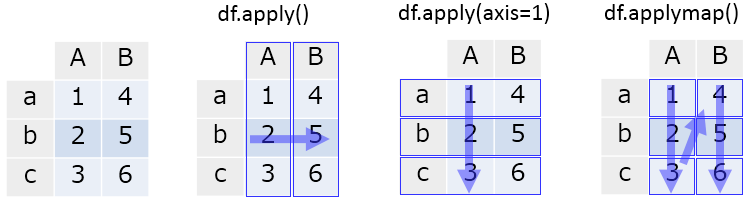
df = pd.DataFrame({'A': [1, 2, 3], 'B': [4, 5, 6]}, index=['a', 'b', 'c'])
df
# A B
# a 1 4
# b 2 5
# c 3 6
# 各列に対して関数適用
df.apply(lambda x: np.sum(x))
A 6
B 15
dtype: int64
# 各行に対して関数適用
df.apply(lambda x: np.sum(x), axis=1)
a 5
b 7
c 9
dtype: int64
# 各値に対して関数適用
df.applymap(lambda x: x * 2)
# A B
# a 2 8
# b 4 10
# c 6 12
# apply で適用される関数には、各列もしくは各行が Series として渡される
df.apply(type)
# A <class 'pandas.core.series.Series'>
# B <class 'pandas.core.series.Series'>
# dtype: object
# applymap で適用される関数には、値そのものが引数として渡される
df.applymap(type)
# A B
# a <type 'numpy.int64'> <type 'numpy.int64'>
# b <type 'numpy.int64'> <type 'numpy.int64'>
# c <type 'numpy.int64'> <type 'numpy.int64'>
GroupBy
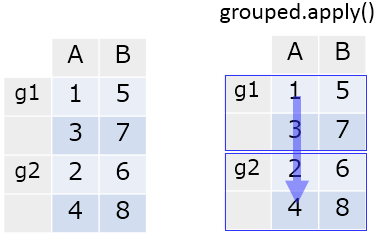
GroupBy については、GroupBy.apply で各グループに関数を適用できる。
df = pd.DataFrame({'group': ['g1', 'g2', 'g1', 'g2'],
'A': [1, 2, 3, 4], 'B': [5, 6, 7, 8]},
columns=['group', 'A', 'B'])
df
# group A B
# 0 g1 1 5
# 1 g2 2 6
# 2 g1 3 7
# 3 g2 4 8
grouped = df.groupby('group')
grouped.apply(np.mean)
# A B
# group
# g1 2 6
# g2 3 7
補足 処理最適化のため、対象となるグループの数 == 関数適用の実行回数とはならないので注意。関数中で破壊的な処理を行うと意図しない結果になりうる。
# 適用される関数 def f(x): print('called') return x # グループ数は 2 grouped.ngroups # 2 # f の実行は 3 回 grouped.apply(f) # called # called # called
pipe
先日 リリースされた v0.16.2 にて pipe メソッドが追加された。これは R の {magrittr} というパッケージからインスパイアされたもので、データへの連続した操作を メソッドチェイン (複数のメソッドの連続した呼び出し) で記述することを可能にする。
Series.pipe、DataFrame.pipe それぞれ、x.pipe(f, *args, **kwargs) は f(x, *args, **kwargs) と同じ。つまり、データ全体に対する関数適用になる。

補足 GroupBy.pipe は v0.16.2 時点では存在しない。
# 渡される型は呼び出し元のインスタンス s.pipe(type) # pandas.core.series.Series df.pipe(type) # pandas.core.frame.DataFrame np.random.seed(1) df = pd.DataFrame(np.random.randn(10, 10)) # DataFrame を引数として heatmap を描く関数を定義 def heatmap(df): import matplotlib.pyplot as plt fig, ax = plt.subplots() return ax.pcolor(df.values, cmap='Greens') # heatmap(df) と同じ。 df.pipe(heatmap)
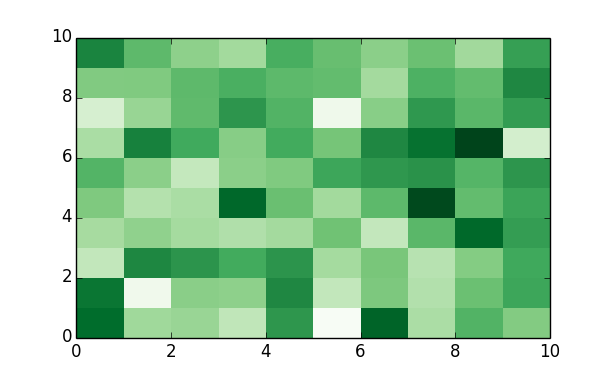
まとめ
イテレーション、関数適用、pipe について整理した。特に関数適用は データの前処理時に頻出するため、パターンを覚えておくと便利。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る
Python pandas + folium + Jupyter でリーフレット / コロプレス図を描きたい
引き続き、 R の可視化を Python に持ってくるシリーズ。R には以下のようなパッケージがあり、地図上へのリーフレット配置やコロプレス図の描画がカンタンにできる。それぞれの概要はリンク先を。
{leaflet}: リーフレット配置{choroplethr}: コロプレス図の描画
これを Python でやりたい。調べてみると folium というパッケージが上記 両方をサポートしているようなので使ってみる。
インストール
pip で。
pip install folium
準備
以降の操作は Jupyter Notebook から行う。まずはパッケージをロードする。
import numpy as np import pandas as pd import folium
folium は プロット結果を html としてエクスポートすることを想定して作成されているようだ。そのため、結果を Jupyter 上に埋め込みたい場合は 以下のような関数を定義する必要がある。
from IPython.display import HTML def inline_map(m): # 中間生成される json が必要なプロットがあるため、一度 html として書き出し m.create_map(path='tmp.html') iframe = '<iframe srcdoc=\"{srcdoc}\" style=\"width: 100%; height: 400px; border: none\"></iframe>' return HTML(iframe.format(srcdoc=m.HTML.replace('\"', '"')))
リーフレット
手順は以下のようになる。
folium.Mapで地図を描画する範囲を緯度経度/ズームにより指定Map.simple_markerで緯度経度を指定してリーフレットを配置 (複数配置する場合は繰り返し)Map.create_mapで地図を含むhtmlを生成 (Jupyter上に描画する場合は上で定義した関数inline_mapを呼ぶ )
m = folium.Map(location=[33.763, -84.392], zoom_start=17) m.simple_marker([33.763006, -84.392912], popup='World of Coca-Cola') inline_map(m)

補足 Jupyter 上ではスクロール / 拡大縮小できる。
既定では OpenStreetMap が利用されるが、Mapbox や maps.stamen を使うこともできる。また、リーフレットのマーカーとしては以下のものが利用できる。
- Simple Markers: シンプルなマーカー (上のもの)
- Circle Markers: 円形のマーカー
- Polygon Markers: 多角形のマーカー
- Lat/Lng Popups: 緯度経度を表示するマーカー
- Click-for-Marker: クリックで配置可能なマーカー
- Vincent Popups: Vincent によるプロットを埋め込めるマーカー
それぞれの描画サンプルは README で確認することができる。
コロプレス図
コロプレス図を描くには以下2つのデータソースが必要である。
GeoJSONもしくはTopoJSON形式のファイル- コロプレス図を色分けするための値を含む
pandasのDataFrame
サンプルとして 国別のマクドナルドの店舗数 をプロットする。
GeoJSON ファイルの準備
国別にプロットするため、国別の GeoJSON ファイルがほしい。以下リポジトリの countries.geo.json をローカルに保存して使う。
中身は 以下のように ISO 3166-1 alpha-3 の国コードを id としたデータとなっている。
{"type":"FeatureCollection",
"features":[{"type":"Feature","id":"AFG",
"properties":{"name":"Afghanistan"},
"geometry":{"type":"Polygon","coordinates":[[[61.210817,35.650072],[62.230651,35.270664],...
DataFrame の準備
DataFrame は 上で準備した GeoJSON と紐づけるためのキー (一般には GeoJSON の id ) と値の 2 列をもつ必要がある。 国別のマクドナルドの店舗数 データを pd.read_html で読み込む。
url = "https://en.wikipedia.org/wiki/List_of_countries_with_McDonald%27s_restaurants" df = pd.read_html(url, header=0, index_col=0)[0] # 列名を変更 df.columns = ['Country', 'Date', 'First outlet location', 'Number', 'Source', 'Note', 'CEO'] df[['Country', 'Number']].head() # Country Number # # # 1 United States 14267 # 2 Canada 1427 # 3 Puerto Rico 108 # 4 U.S. Virgin Islands 6 # 5 Costa Rica 54
こちらのデータには国名が入っているため、GeoJSON と紐づけるためには ISO 国コードに変換する必要がある。変換には pycountry を使う。インストールしていない方は pip で。
import pycountry pycountry.countries.get(name='Japan').alpha3 # 'JPN' # データ中の国名が pycountry のものと違う場合の mapper countries = {'United Kingdom': 'United Kingdom', 'Russia': 'Russian Federation', 'South Korea': 'Korea, Republic of', 'Taiwan': 'Taiwan, Province of China', 'Vietnam': 'Viet Nam', } def f(x): for k, v in pd.compat.iteritems(countries): if k in x: x = v try: return pycountry.countries.get(name=x).alpha3 except KeyError: return np.nan # 国コードへの変換 df.loc[:, 'Code'] = df['Country'].apply(f) # 国コードに変換できなかったデータは除外 df = df.dropna(subset=['Code']) # 欠損値を 0 でパディング df.loc[:, 'Number'] = df['Number'].fillna('0') # 数値に変換できない文字列があるため、余計な文字を削除 df.loc[:, 'Number'] = df['Number'].str.replace('[+,]', '') # 数値 (float) 型に変換 df.loc[:, 'Number'] = df['Number'].astype(float) df[['Code', 'Country', 'Number']].sort('Number', ascending=False).head() # Code Country Number # # # 1 USA United States 14267 # 8 JPN Japan 3164 # 49 CHN China 2000 # 11 DEU Germany 1468 # 2 CAN Canada 1427
これで、国コード / プロットする値をもつ DataFrame が準備できた。アメリカすごいな、、、。
コロプレス図の描画
Map.geo_json で コロプレス図を描画するレイヤーを Map に追加できる。ここで使っている引数の意味は以下。
geo_path:GeoJSONファイルのパスdata: 色分けのための値をもつDataFramecolumns:data中GeoJSONと紐づけるキー / 値 を含む列名key_on:GeoJSON側で紐付けに使うキーthreshold_scale: 色分けをする際の閾値fill_color色分けに使う color-brewer の名前reset: 既存のレイヤがある場合に削除する
m = folium.Map(location=[10, 35], zoom_start=1.5) geojson = r'countries.geo.json' m.geo_json(geo_path=geojson, data=df, columns=['Code', 'Number'], key_on='feature.id', threshold_scale=[1, 100, 500, 1000, 2000, 4000], fill_color='BuPu', reset=True) inline_map(m)

まとめ
folium を使えば リーフレット / コロプレス図の描画がカンタンにできる。Jupyter 上で Javascript を使用してのデータ可視化、結構使えるのでは。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る
Python pandas のデータを Highcharts/Highstock + Jupyter でプロットしたい
R を使っている方はご存知だと思うが、R には {htmlwidgets} というパッケージがあり、R 上のデータを任意の Javascript ライブラリを使ってプロットすることが比較的カンタンにできる。{htmlwidgets} って何?という方には こちらの説明がわかりやすい。
同じことを Python + pandas を使ってやりたい。サンプルとして利用する Javascript ライブラリは 上の資料と同じく Highcharts、Highstock にする。
補足 pandas-highcharts という Python パッケージもあるが、このエントリでは任意の Javascript ライブラリで使えるであろう方法を記載する。
Highcharts でのプロット
以降の操作は Jupyter Notebook 上で行う。まずは必要パッケージをロードする。
import numpy as np import pandas as pd from IPython.display import HTML
続けて、Highcharts、Highstock を読み込む。これは %%html magic を使うのが楽。
%%html
<script src="http://code.highcharts.com/highcharts.js"></script>
<script src="http://code.highcharts.com/stock/highstock.js"></script>
<script src="http://code.highcharts.com/modules/exporting.js"></script>
Highcharts でプロットする準備ができたため、%%html magic を使って サンプル Your first chart に記載のサンプルをプロットしてみる。これでプロット時のスクリプト / データ構造が確認できる。
%%html
<div id="container" style="width:100%; height:400px;"></div>
<script>
plot = function () {
$('#container').highcharts({
chart: {
type: 'bar'
},
title: {
text: 'Fruit Consumption'
},
xAxis: {
categories: ['Apples', 'Bananas', 'Oranges']
},
yAxis: {
title: {
text: 'Fruit eaten'
}
},
series: [{
name: 'Jane',
data: [1, 0, 4]
}, {
name: 'John',
data: [5, 7, 3]
}]
});
};
plot();
</script>
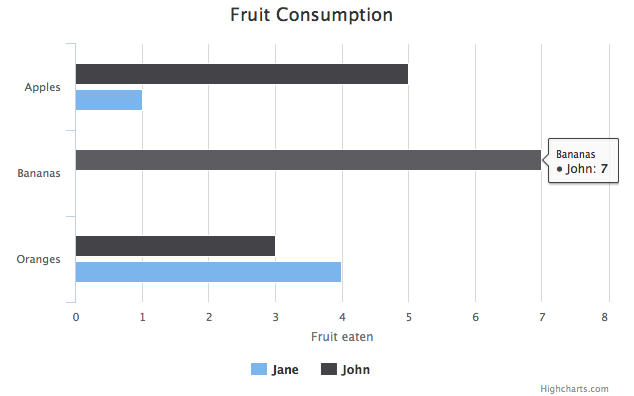
補足 Jupyter から実行すればアニメーションする。
pandas のデータをプロットしたい場合は、上のスクリプトと同じように .highcharts() の引数にあわせた形式でデータを渡し、HTML としてレンダリングしてやればうまくいきそうだ。pandas で元データとなる DataFrame を定義して、上のスクリプトの形式に変換していく。
df = pd.DataFrame({'Jane': [1, 0, 4], 'John': [5, 7, 3]},
index=['Apples', 'Bananas', 'Oranges'])
df
# Jane John
# Apples 1 5
# Bananas 0 7
# Oranges 4 3
スクリプトは文字列結合で作ってもよいが、引数となる json 形式に対応する辞書型のデータをつくってからjson.dumps したほうが簡単だろう。DataFrame.to_json でデータを直接 変換できると楽なのだが、フォーマットが違うため無理そうだ。
# NG! df.to_json() # '{"Jane":{"Apples":1,"Bananas":0,"Oranges":4}, # "John":{"Apples":5,"Bananas":7,"Oranges":3}}'
そのため、個々の要素ごとに変換を考えていく。うまいこと辞書ができたら json.dumps する。
[{'name': c, 'data': col.tolist()} for c, col in df.iteritems()]
# [{'data': [1, 0, 4], 'name': 'Jane'}, {'data': [5, 7, 3], 'name': 'John'}]
chartdict = {'chart': {'type': 'bar'},
'title': {'text': 'Fruit Consumption'},
'xAxis': {'categories': df.index.tolist()},
'yAxis': {'title': {'text': 'Fruit eaten'}},
'series': [{'name': c, 'data': col.tolist()} for c, col in df.iteritems()]
}
import json
json.dumps(chartdict)
# '{"series": [{"data": [1, 0, 4], "name": "Jane"}, {"data": [5, 7, 3], "name": "John"}],
# "yAxis": {"title": {"text": "Fruit eaten"}}, "chart": {"type": "bar"},
# "xAxis": {"categories": ["Apples", "Bananas", "Oranges"]},
# "title": {"text": "Fruit Consumption"}}'
あとは 必要な HTML / Javascript のテンプレートを文字列として作って format すればよい。
template = """ <script src="http://code.highcharts.com/highcharts.js"></script> <script src="http://code.highcharts.com/modules/exporting.js"></script> <div id="{chart}" style="width:100%; height:400px;"></div> <script type="text/javascript"> plot = function () {{ $("#{chart}").highcharts({data}); }}; plot(); </script> """ HTML(template.format(chart='container2', data=json.dumps(chartdict))) # 略
Highstock でのプロット
同様に、Highstock へプロットすることもできる。サンプル Single line series を pandas のデータを使ってプロットしてみる。
補足 株価の取得には以下のパッケージを使う。
import japandas as jpd toyota = jpd.DataReader(7203, 'yahoojp', start='2015-01-01') toyota.head() # 始値 高値 安値 終値 出来高 調整後終値* # 日付 # 2015-01-05 7565 7575 7416 7507 9515300 7507 # 2015-01-06 7322 7391 7300 7300 12387900 7300 # 2015-01-07 7256 7485 7255 7407 11465400 7407 # 2015-01-08 7500 7556 7495 7554 10054500 7554 # 2015-01-09 7630 7666 7561 7609 10425400 7609
Highstock に渡すデータの形式は以下のサイトがわかりやすい。引数としては [UNIX時間(ミリ秒), 始値, 高値, 安値, 終値] を入れ子のリストにして渡せばよいようだ。
時刻は UNIX時間で渡す必要があるので、少し操作が必要だ。上で取得した DataFrame は日時型の Index である DatetimeIndex を持っている。DatetimeIndex はUNIXエポックを基準とした現在時刻をナノ秒で保存しているため、int 型に変換して 1000000 で割ればUNIX時間(ミリ秒)となる。したがって、Highstock へ渡すデータは以下のようにして作れる。
toyota.index.astype(int) / 1000000 # array([1420416000000, 1420502400000, 1420588800000, 1420675200000, # 1420761600000, 1421107200000, 1421193600000, 1421280000000, # .... # 1433721600000, 1433808000000, 1433894400000, 1433980800000, # 1434067200000]) toyota['time'] = toyota.index.astype(int) / 1000000 toyota[['time', u'始値', u'高値', u'安値', u'終値']].values.tolist() # [[1420416000000, 7565, 7575, 7416, 7507], # [1420502400000, 7322, 7391, 7300, 7300], # ... # [1433980800000, 8250, 8326, 8241, 8322], # [1434067200000, 8387, 8394, 8329, 8394]]
描画する。アニメーションも含め、うまく動いているようだ。
chartdict = {'rangeSelector': {'selected': 1},
'title': {'text' : 'Stock Price'},
'series': [{'name' : u'トヨタ',
'data': toyota[['time', u'始値', u'高値', u'安値', u'終値']].values.tolist(),
'tooltip': {'valueDecimals': 2}}]}
template = """
<div id="{chart}" style="width:100%; height:400px;"></div>
<script type="text/javascript">
plot = function () {{
$('#{chart}').highcharts('StockChart', {data});
}};
plot();
</script>
HTML(template.format(chart='container3', data=json.dumps(chartdict)))
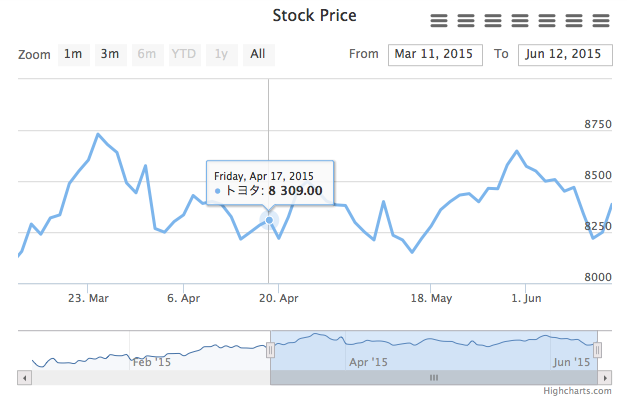
まとめ
pandas のデータを任意の Javascript ライブラリでプロットする際には、
- 当該の Javascript ライブラリが利用するデータ構造を確認する
- そのデータ構造にあうように
pandasのデータを変換し、辞書型を作る json.dumpsでスクリプトに渡す
とやればだいたいできると思う。
補足 ここで今 話題の spyre 上でもプロットしたいな?と思ったのだが、spyre で普通に HTML としてレンダリングするとうまく動かない。spyre が内部で使っている cherrypy も DOM を書き換えているのが原因かと思っているのだが、自分にはそのあたりの知識がまったくないのでよくわからない。できた方がいれば教えてください。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る