pandas 0.16.0/0.16.1 の主要な新機能
先日 5/11 に pandas 0.16.1 がリリースされた。前バージョンである 0.16.0 とあわせて、主要な変更点である以下3点の概要をまとめたい。各見出しの括弧内には対応したバージョンを記載した。
- 簡単な列追加 /
DataFrame.assign(0.16.0) - 文字列処理の強化 (0.16.0/0.16.1)
- ランダムサンプリング
DataFrame.sample(0.16.1)
変更点全体はリリースノートを参照。上記3点以外にも便利な変更はあるのだが、 Categorical や Frequency など 元機能の説明がないとわからない箇所なので別途、、。
簡単な列追加 / DataFrame.assign (0.16.0)
DataFrame への列追加をより簡潔に行うためのメソッドとして、DataFrame.assign が追加された。R {dplyr} の mutate 関数に似た書式で複数列の追加ができる。
これまで列追加の際には カラムを指定して代入し、元データを破壊的に変更する必要があった。
import pandas as pd df = pd.DataFrame({'A':[1, 2, 3], 'B':[4, 5, 6]}) df # A B # 0 1 4 # 1 2 5 # 2 3 6 # これまでの列追加 df['C'] = [7, 8, 9] df # A B C # 0 1 4 7 # 1 2 5 8 # 2 3 6 9
assign を使うと、作成したい列名をキーワード引数として以下のように書ける。また、戻り値は元データに列追加したコピーとなり、元データ自体は変更されない。
# D列, E列を追加 df.assign(D=[10, 11, 12], E=[13, 14, 15]) # A B C D E # 0 1 4 7 10 13 # 1 2 5 8 11 14 # 2 3 6 9 12 15 # 元データはそのまま df # A B C # 0 1 4 7 # 1 2 5 8 # 2 3 6 9
キーワード引数の値には通常の列作成時と同じ型が渡せる。各列の値を組み合わせた列 + ダミー列を同時に作りたければ、
df.assign(AB=df['A'] * df['B'], AC=df['A'] + df['C'], dummy=1) # A B C AB AC dummy # 0 1 4 7 4 8 1 # 1 2 5 8 10 10 1 # 2 3 6 9 18 12 1
キーワード引数として渡せない列名、例えば記号や日本語を含む列名は、一度 辞書にしてから渡せばよい。
keys = {'A*B': df['A'] * df['B'], 'A+B':df['A'] + df['B']}
df.assign(**keys)
# A B A*B A+B
# 0 1 4 4 5
# 1 2 5 10 7
# 2 3 6 18 9
補足: メソッド自体は 0.16.0 で追加され、0.16.1 でキーワード引数の処理順序がアルファベット順に固定された。
文字列処理の強化 (0.16.0/0.16.1)
pandas での文字列処理について過去に以下の記事を書いたことがあるのだが、当時はアクセサから利用できるメソッドが限定されていて、少し手間がかかるところがあった。
0.16.0, 0.16.1 で、.str アクセサに以下のメソッド群が追加された。それぞれ、 Python 標準の文字列メソッドと同一の処理を Series 内の値に対して適用するもの。
| Methods | ||||
|---|---|---|---|---|
isalnum |
isalpha |
isdigit |
isspace |
islower |
isupper |
istitle |
isnumeric |
isdecimal |
find |
rfind |
ljust |
rjust |
zfill |
capitalize |
swapcase |
normalize |
partition |
rpartition |
index |
rindex |
translate |
補足 str.normalize は ユニコード正規化 ( unicodedata.normalize ) を値に対して適用するもの。
補足 .str アクセサから利用可能なメソッド全体はこちら。
df = pd.DataFrame({'A': ['xxx', '3', 'yyy'], 'B': [1, 2, 3]})
df
# A B
# 0 xxx 1
# 1 3 2
# 2 yyy 3
# 先頭を大文字に
df['A'].str.capitalize()
# 0 Xxx
# 1 3
# 2 Yyy
# Name: A, dtype: object
# 文字列が数値がどうかを調べる
df['A'].str.isdigit()
# 0 False
# 1 True
# 2 False
# Name: A, dtype: bool
# 5桁分を 0 パディング
df['A'].str.zfill(5)
# 0 00xxx
# 1 00003
# 2 00yyy
# Name: A, dtype: object
やりたいことに応じて適当に組み合わせると、かなり柔軟な処理がかける。例えば 数値の文字列のみを 0 パディングしたければ、
df.loc[df['A'].str.isdigit(), 'A'] = df['A'].str.zfill(5) df # A B # 0 xxx 1 # 1 00003 2 # 2 yyy 3
補足 DataFrame.loc の意味はこちらの記事参照。
また、0.16.1 から .str アクセサを Index からも呼び出せるようになった。これまでは 列名/行名に対する文字列処理は .map を使って関数適用する必要があったが、.str を使えば以下のように書ける。
df # A B # 0 xxx 1 # 1 00003 2 # 2 yyy 3 df.columns.str.lower() # Index([u'a', u'b'], dtype='object') # 列名を小文字に変更 df.columns = df.columns.str.lower() df # a b # 0 xxx 1 # 1 00003 2 # 2 yyy 3
str.split のように複数の値を返しうる処理については、expand オプションを利用して 返り値の型を制御できる。互換性維持のため、既定値はメソッドにより異なる。APIドキュメントを参照。
expand=False: 返り値の次元を増やさない = 返り値はSeriesもしくはIndexexpand=True: 返り値の次元を増やす = 返り値はDataFrameもしくはMultiIndex
補足 これまで同様の制御をおこなっていた return_type オプションは deprecate されており、将来のバージョンで削除される。
s = pd.Series(['a,b', 'a,c', 'b,c']) # 返り値は Series s.str.split(',') # 0 [a, b] # 1 [a, c] # 2 [b, c] # dtype: object # 返り値は DataFrame s.str.split(',', expand=True) # 0 1 # 0 a b # 1 a c # 2 b c idx = pd.Index(['a,b', 'a,c', 'b,c']) # 返り値は 1 レベルの Index idx.str.split(',') # Index([[u'a', u'b'], [u'a', u'c'], [u'b', u'c']], dtype='object') # 返り値は 2 レベルの MultiIndex idx.str.split(',', expand=True) # MultiIndex(levels=[[u'a', u'b'], [u'b', u'c']], # labels=[[0, 0, 1], [0, 1, 1]])
ランダムサンプリング DataFrame.sample (0.16.1)
Series, DataFrame, Panel から適当なデータをサンプリングするためのメソッドとして、.sample が追加された。
ここでは DataFrame.sample を例としてその処理を記載する。まず以下のようなデータを用意した。
df = pd.DataFrame({'A': [1, 2 ,3, 4, 5], 'B': ['a', 'b', 'c', 'd', 'e']})
df
# A B
# 0 1 a
# 1 2 b
# 2 3 c
# 3 4 d
# 4 5 e
DataFrame.sample は 既定では 1 行をランダムにサンプリングする。
n: サンプルサイズを指定する。既定は 1。axis: サンプリングするレコードの方向を指定する。0(既定) で行のサンプリング、1で列のサンプリング。
# 1 行の抽出 df.sample() # A B # 3 4 d # 3 行の抽出 df.sample(3) # A B # 0 1 a # 4 5 e # 2 3 c # 1 列の抽出 df.sample(axis=1) # A # 0 1 # 1 2 # 2 3 # 3 4 # 4 5
また、サンプルサイズでなく抽出比を指定する場合は frac を指定する。
df.sample(frac=0.4) # A B # 3 4 d # 1 2 b
最後に、replace オプションを利用して 抽出方法を変更することができる。
replace=False(既定): 非復元抽出。サンプリングされた各要素の重複を許さない。replace=True: 復元抽出。各要素の重複を許す。
# 復元抽出。2行目、4行目が重複 df.sample(n=4, replace=True) # A B # 1 2 b # 2 3 c # 0 1 a # 2 3 c # 非復元抽出では 元のデータ数以上のサンプルサイズは取れない df.sample(n=6) # ValueError: Cannot take a larger sample than population when 'replace=False' df.sample(n=6, replace=True) # A B # 1 2 b # 4 5 e # 0 1 a # 1 2 b # 2 3 c # 4 5 e
補足 これまでは index の要素をサンプリングして選択する必要があった。以下の記事参照。
まとめ
0.16.0 / 0.16.1 の変更点のうち、以下 3 点の概要をまとめた。
- 簡単な列追加 /
DataFrame.assign(0.16.0) - 文字列処理の強化 (0.16.0/0.16.1)
- ランダムサンプリング
DataFrame.sample(0.16.1)
他にも、バグ修正 / 挙動・仕様の統一など 様々な改善が入っているので使ってみてください。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る
簡単な集約/変換処理を PySpark & pandas の DataFrame で行う
こちらの続き。
準備
サンプルデータは iris 。今回は HDFS に csv を置き、そこから読み取って DataFrame を作成する。
# HDFS にディレクトリを作成しファイルを置く $ hadoop fs -mkdir /data/ $ hadoop fs -put iris.csv /data/ $ hadoop fs -ls / Found 1 items drwxr-xr-x - ec2-user supergroup 0 2015-04-28 20:01 /data # Spark のパスに移動 $ echo $SPARK_HOME /usr/local/spark $ cd $SPARK_HOME $ pwd /usr/local/spark $ bin/pyspark
補足 前回同様に pandas から直接 PySpark の DataFrame を作成した場合、groupBy 時に java.lang.OutOfMemoryError: Java heap space エラーが発生してシェルごと落ちる。
CSV ファイルの読み込み
pandas では前回同様 read_csv。
import numpy as np import pandas as pd # 表示する行数を設定 pd.options.display.max_rows=10 names = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth', 'Species'] # pandas pdf = pd.read_csv('~/iris.csv', header=None, names=names) pdf # 略
PySpark は標準では csv から直接 DataFrame を作成できないため、一度 Row のリストを作成して DataFrame に変換する。
from pyspark.sql import Row lines = sc.textFile("hdfs://127.0.0.1:9000/data/iris.csv") cells = lines.map(lambda l: l.split(",")) rows = cells.map(lambda x: Row(SepalLength=float(x[0]), SepalWidth=float(x[1]), PetalLength=float(x[2]), PetalWidth=float(x[3]), Species=x[4])) sdf = sqlContext.createDataFrame(rows) sdf.show() # 略
グルーピング/集約
ある列の値ごとに集計
pandas, PySpark で多少 文法は異なる。
列の値でグループ分けし、一列の合計を取得する場合:
# pandas pdf.groupby('Species')['SepalLength'].sum() # Species # setosa 250.3 # versicolor 296.8 # virginica 329.4 # Name: SepalLength, dtype: float64 # PySpark sdf.groupBy('Species').sum('SepalLength').show() # Species SUM(SepalLength) # virginica 329.3999999999999 # versicolor 296.8 # setosa 250.29999999999998
指定した複数列の合計を取得する場合:
# pandas pdf.groupby('Species')[['PetalWidth', 'PetalLength']].sum() # PetalWidth PetalLength # Species # setosa 12.2 73.2 # versicolor 66.3 213.0 # virginica 101.3 277.6 # PySpark sdf.groupBy('Species').sum('PetalWidth', 'PetalLength').show() # Species SUM(PetalWidth) SUM(PetalLength) # virginica 101.29999999999998 277.59999999999997 # versicolor 66.30000000000001 213.0 # setosa 12.199999999999996 73.2
全列の合計を取得する場合:
# pandas pdf.groupby('Species').sum() # SepalLength SepalWidth PetalLength PetalWidth # Species # setosa 250.3 170.9 73.2 12.2 # versicolor 296.8 138.5 213.0 66.3 # virginica 329.4 148.7 277.6 101.3 # PySpark sdf.groupBy('Species').sum().show() # Species SUM(PetalLength) SUM(PetalWidth) SUM(SepalLength) SUM(SepalWidth) # virginica 277.59999999999997 101.29999999999998 329.3999999999999 148.7 # versicolor 213.0 66.30000000000001 296.8 138.5 # setosa 73.2 12.199999999999996 250.29999999999998 170.90000000000003
補足 pandas では グループ化したデータも DataFrame と同じようにスライシングできたりする。
一方、PySpark の GroupedData は集約系のAPI しか持っていない。
# pandas pdf.groupby('Species')['PetalWidth'] # <pandas.core.groupby.SeriesGroupBy object at 0x7f62f4218d50> # PySpark (NG!) sdf.groupBy('Species')[['Species']] # TypeError: 'GroupedData' object has no attribute '__getitem__' sdf.groupBy('Species').select('PetalWidth') # AttributeError: 'GroupedData' object has no attribute 'select'
また、pandas では apply で自作の集約関数 (UDAF) を利用することができるが、PySpark 1.3.1 時点 では非対応らしい。PySpark の udf を利用して定義した自作関数を集約時に使うと以下のエラーになる。
# pandas pdf.groupby('Species')[['PetalWidth', 'PetalLength']].apply(np.sum) # PetalWidth PetalLength # Species # setosa 12.2 73.2 # versicolor 66.3 213.0 # virginica 101.3 277.6 # PySpark (NG!) import pyspark.sql.functions np_sum = pyspark.sql.functions.udf(np.sum, pyspark.sql.types.FloatType()) sdf.groupBy('Species').agg(np_sum(sdf.PetalWidth)) # py4j.protocol.Py4JJavaError: An error occurred while calling o334.agg. # : org.apache.spark.sql.AnalysisException: expression 'pythonUDF' is neither present in the group by, nor is it an aggregate function. Add to group by or wrap in first() if you don't care which value you get.;
行持ち / 列持ち変換
複数列持ちの値を行持ちに展開 (unpivot / melt)
pandas では pd.melt。 DataFrame.melt ではないので注意。
# pandas pmelted = pd.melt(pdf, id_vars=['Species'], var_name='variable', value_name='value') pmelted # Species variable value # 0 setosa SepalLength 5.1 # 1 setosa SepalLength 4.9 # 2 setosa SepalLength 4.7 # 3 setosa SepalLength 4.6 # 4 setosa SepalLength 5.0 # .. ... ... ... # 595 virginica PetalWidth 2.3 # 596 virginica PetalWidth 1.9 # 597 virginica PetalWidth 2.0 # 598 virginica PetalWidth 2.3 # 599 virginica PetalWidth 1.8 # # [600 rows x 3 columns]
同様の処理を PySpark でやるには、DataFrame.flatMap。1行の入力に対して複数行 (この例では4行) のデータを返すことができる。fratMap の返り値は RDD インスタンスになるため、必要なら再度 DataFrame 化する。
# PySpark def mapper(row): return [Row(Species=row[4], variable='PetalLength', value=row[0]), Row(Species=row[4], variable='PetalWidth', value=row[1]), Row(Species=row[4], variable='SepalLength', value=row[2]), Row(Species=row[4], variable='SepalWidth', value=row[3])] smelted = sqlContext.createDataFrame(sdf.flatMap(mapper)) smelted.show() # Species value variable # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.1 SepalLength # setosa 3.5 SepalWidth # ... .. ... # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.0 SepalLength # setosa 3.6 SepalWidth smelted.count() # 600L
複数行持ちの値を列持ちに変換 (pivot)
pandas では DataFrame.pivot。pivotするデータは列にする値 (以下では Species ) と行にする値 (以下では variable ) の組がユニークになっている必要がある。そのため、まず pivot 用データを作成 -> その後 pivot する。
# pandas # pivot 用データを作成 punpivot = pmelted.groupby(['Species', 'variable']).sum() punpivot = punpivot.reset_index() punpivot # Species variable value # 0 setosa PetalLength 73.2 # 1 setosa PetalWidth 12.2 # 2 setosa SepalLength 250.3 # 3 setosa SepalWidth 170.9 # 4 versicolor PetalLength 213.0 # .. ... ... ... # 7 versicolor SepalWidth 138.5 # 8 virginica PetalLength 277.6 # 9 virginica PetalWidth 101.3 # 10 virginica SepalLength 329.4 # 11 virginica SepalWidth 148.7 # # [12 rows x 3 columns] # pivot punpivot.pivot(index='variable', columns='Species', values='value') # Species setosa versicolor virginica # variable # PetalLength 73.2 213.0 277.6 # PetalWidth 12.2 66.3 101.3 # SepalLength 250.3 296.8 329.4 # SepalWidth 170.9 138.5 148.7
PySpark の DataFrame のままでは同じ処理はできないようなので、一度 RDD に変換してから、 groupBy -> map
# PySpark # pivot 用データを作成 sunpivot = smelted.groupBy('Species', 'variable').sum() sunpivot.show() # Species variable SUM(value) # versicolor SepalWidth 138.5 # versicolor SepalLength 296.8 # setosa PetalLength 73.2 # virginica PetalWidth 101.29999999999998 # versicolor PetalWidth 66.30000000000001 # setosa SepalWidth 170.90000000000003 # virginica PetalLength 277.59999999999997 # setosa SepalLength 250.29999999999998 # versicolor PetalLength 213.0 # setosa PetalWidth 12.199999999999996 # virginica SepalWidth 148.7 # virginica SepalLength 329.3999999999999 def reducer(obj): # variable : value の辞書を作成 result = {o[1]:o[2] for o in obj[1]} return Row(Species=obj[0], **result) # pivot spivot = sunpivot.rdd.groupBy(lambda x: x[0]).map(reducer) spivot.collect() # [Row(PetalLength=277.59999999999997, PetalWidth=101.29999999999998, SepalLength=329.3999999999999, SepalWidth=148.7, Species=u'virginica'), # Row(PetalLength=73.2, PetalWidth=12.199999999999996, SepalLength=250.29999999999998, SepalWidth=170.90000000000003, Species=u'setosa'), # Row(PetalLength=213.0, PetalWidth=66.30000000000001, SepalLength=296.8, SepalWidth=138.5, Species=u'versicolor')] sqlContext.createDataFrame(spivot).show() # PetalLength PetalWidth SepalLength SepalWidth Species # 277.59999999999997 101.29999999999998 329.3999999999999 148.7 virginica # 73.2 12.199999999999996 250.29999999999998 170.90000000000003 setosa # 213.0 66.30000000000001 296.8 138.5 versicolor
列の分割 / 結合
列の値を複数列に分割
ある列の値を適当に文字列処理して、新しい列を作成したい。pandas には 文字列処理用のアクセサがあるため、 assign と組み合わせて以下のように書ける。
# pandas psplitted = pmelted.assign(Parts=pmelted['variable'].str.slice(0, 5), Scale=pmelted['variable'].str.slice(5)) psplitted # Species variable value Parts Scale # 0 setosa SepalLength 5.1 Sepal Length # 1 setosa SepalLength 4.9 Sepal Length # 2 setosa SepalLength 4.7 Sepal Length # 3 setosa SepalLength 4.6 Sepal Length # 4 setosa SepalLength 5.0 Sepal Length # .. ... ... ... ... ... # 595 virginica PetalWidth 2.3 Petal Width # 596 virginica PetalWidth 1.9 Petal Width # 597 virginica PetalWidth 2.0 Petal Width # 598 virginica PetalWidth 2.3 Petal Width # 599 virginica PetalWidth 1.8 Petal Width # # [600 rows x 5 columns]
PySparkには上記のようなメソッドはないので map で処理する。
# PySpark def splitter(row): parts = row[2][:5] scale = row[2][5:] return Row(Species=row[0], value=row[1], Parts=parts, Scale=scale) ssplitted = sqlContext.createDataFrame(smelted.map(splitter)) ssplitted.show() # Parts Scale Species value # Petal Length setosa 1.4 # Petal Width setosa 0.2 # Sepal Length setosa 5.1 # Sepal Width setosa 3.5 # Petal Length setosa 1.4 # .. .. ... .. # Petal Length setosa 1.4 # Petal Width setosa 0.2 # Sepal Length setosa 5.0 # Sepal Width setosa 3.6
複数列の値を一列に結合
pandas では普通に文字列結合すればよい。
# pandas psplitted['variable2'] = psplitted['Parts'] + psplitted['Scale'] psplitted # Species variable value Parts Scale variable2 # 0 setosa SepalLength 5.1 Sepal Length SepalLength # 1 setosa SepalLength 4.9 Sepal Length SepalLength # 2 setosa SepalLength 4.7 Sepal Length SepalLength # 3 setosa SepalLength 4.6 Sepal Length SepalLength # 4 setosa SepalLength 5.0 Sepal Length SepalLength # .. ... ... ... ... ... ... # 595 virginica PetalWidth 2.3 Petal Width PetalWidth # 596 virginica PetalWidth 1.9 Petal Width PetalWidth # 597 virginica PetalWidth 2.0 Petal Width PetalWidth # 598 virginica PetalWidth 2.3 Petal Width PetalWidth # 599 virginica PetalWidth 1.8 Petal Width PetalWidth # # [600 rows x 6 columns]
PySpark では map。
# PySpark def unite(row): return Row(Species=row[2], value=row[3], variable=row[0] + row[1]) sqlContext.createDataFrame(splitted.map(unite)).show() # Species value variable # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.1 SepalLength # setosa 3.5 SepalWidth # .. .. .. # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.0 SepalLength # setosa 3.6 SepalWidth
補足 withColumn の場合、オペレータは 数値の演算として扱われてしまうようなのでここでは使えない。
# PySpark (NG!) ssplitted.withColumn('variable', splitted.Parts + splitted.Scale).show() # Parts Scale Species value variable # Petal Length setosa 1.4 null # Petal Width setosa 0.2 null # .. .. .. .. ..
まとめ
PySpark と pandas のデータ集約/変形処理を整理した。
データ分析用途で利用したい場合、(ごく当たり前だが) データ量が少なく手元でさっといろいろ試したい場合は pandas、データ量が比較的多く 単純な処理を全体にかけたい場合は Spark がよい。
Spark は map 系の API が充実するとさらに使いやすくなりそうだ。が、小回りの効く文法/機能が充実していくことは考えにくいので 完全に Spark だけでデータ分析をする、、という状態には将来もならないのではないかと思う。小さいデータは pandas 使いましょう。

Learning Spark: Lightning-Fast Big Data Analysis
- 作者: Holden Karau,Andy Konwinski,Patrick Wendell,Matei Zaharia
- 出版社/メーカー: O'Reilly Media
- 発売日: 2015/01/28
- メディア: Kindle版
- この商品を含むブログを見る
Python pandas 関連エントリの目次
このブログ中の pandas 関連のエントリをまとめた目次です。
最近 pandas 開発チーム と PyData グループ の末席に加えていただき、パッケージ自体の改善にもより力を入れたいと思います。使い方についてご質問などありましたら Twitter で @ ください。
目次につけた絵文字は以下のような意味です。
- 🔰: 最初に知っておけば一通りの操作ができそうな感じのもの。
- 🚧: v0.16.0 時点で少し情報が古く、機能の改善を反映する必要があるもの。
- 🚫: 当該の機能が deprecate 扱いとなり、将来的に 代替の方法が必要になるもの。
基本
また、上記に対応した比較エントリ:
R {dplyr}, {tidyr}
PySpark
Julia
機能別
データ選択
- Python pandas データ選択処理をちょっと詳しく <前編>
- Python pandas データ選択処理をちょっと詳しく <中編>
- Python pandas データ選択処理をちょっと詳しく <後編>
グルーピング/集約/集計/データ変形
- Python pandas の算術演算 / 集約関数 / 統計関数まとめ
- Python pandas アクセサ / Grouperで少し高度なグルーピング/集計
- Python pandas データのイテレーションと関数適用、pipe
- Python pandas 図でみる データ連結 / 結合処理
- Python pandas 欠損値/外れ値/離散化の処理
そのほかの前処理
データ操作
文字列、日付など、各データ型に固有の操作。
入出力
可視化
- Python pandas プロット機能を使いこなす
- Python Jupyter + pandas で DataFrame 表示をカスタマイズする
- Python spyre によるデータ分析結果のWebアプリ化
地理情報の可視化
その他
- Python pandas のデータを scikit-learn でうまいこと処理したい
- Python pandas で日本の株価情報取得とローソク足チャート描画
- Python pandas パフォーマンス維持のための 3 つの TIPS
- Dask で 並列 DataFrame 処理
変更点
自作パッケージ
- Python pandas / scikit-learn 向けのちょっとしたパッケージ作った <前編>
- Python pandas / scikit-learn 向けのちょっとしたパッケージ作った <後編>
- Python pandas 日本語環境向けのちょっとしたパッケージ作った
発表資料

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (13件) を見る
簡単なデータ操作を PySpark & pandas の DataFrame で行う
Spark v1.3.0 で追加された DataFrame 、結構いいらしいという話は聞いていたのだが 自分で試すことなく時間が過ぎてしまっていた。ようやく PySpark を少し触れたので pandas との比較をまとめておきたい。内容に誤りや よりよい方法があればご指摘 下さい。
過去に基本的なデータ操作について 以下 ふたつの記事を書いたことがあるので、同じ処理のPySpark 版を加えたい。今回は ひとつめの "簡単なデータ操作〜" に相当する内容。
pandas 版
準備
環境は EC2 に作る。Spark のインストールについてはそのへんに情報あるので省略。サンプルデータは iris を csv でダウンロードしてホームディレクトリにおいた。以降の操作は PySpark のコンソールで行う。
# Spark のパスに移動 $ echo $SPARK_HOME /usr/local/spark $ cd $SPARK_HOME $ pwd /usr/local/spark # 既定だとログの出力が邪魔なので抑制 $ cp conf/log4j.properties.template conf/log4j.properties $ vi conf/log4j.properties # INFO をすべて WARN に変える $ bin/pyspark # Welcome to # ____ __ # / __/__ ___ _____/ /__ # _\ \/ _ \/ _ `/ __/ '_/ # /__ / .__/\_,_/_/ /_/\_\ version 1.3.1 # /_/ # # Using Python version 2.7.9 (default, Apr 1 2015 19:28:03) # SparkContext available as sc, HiveContext available as sqlContext.
ここでは HDFS は使わず、pandas の read_csv で pandas.DataFrame を作成する。列名に "." が入っていると PySpark でうまく動かないようだったため以下のカラム名にした。
import pandas as pd # 表示する行数を設定 pd.options.display.max_rows=10 names = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth', 'Species'] # pandas pdf = pd.read_csv('~/iris.csv', header=None, names=names) pdf # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns]
これを PySpark の DataFrame に変換する。pandas と PySpark の基本的な違いとして、
PySparkにはpandas.IndexにあたるものがないPySparkにはpandas.Seriesにあたるものがない ( 代わり? にRowがあるが、どの程度 柔軟な操作ができるかは未知 )
# PySpark sdf = sqlContext.createDataFrame(pdf) sdf # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double, Species: string] # データの中身を表示するには DataFrame.show() sdf.show() # SepalLength SepalWidth PetalLength PetalWidth Species # 5.1 3.5 1.4 0.2 setosa # 4.9 3.0 1.4 0.2 setosa # 4.7 3.2 1.3 0.2 setosa # 4.6 3.1 1.5 0.2 setosa # .. ... ... ... ... # 5.4 3.9 1.3 0.4 setosa # 5.1 3.5 1.4 0.3 setosa # 5.7 3.8 1.7 0.3 setosa # 5.1 3.8 1.5 0.3 setosa
以降、pdf が pandas、sdf が PySpark の DataFrame をあらわす。
type(pdf) # <class 'pandas.core.frame.DataFrame'> type(sdf) # <class 'pyspark.sql.dataframe.DataFrame'>
基本
まず基本的な操作を。先頭いくつかのデータを確認するには head。
PySpark での返り値は Row インスタンスのリストになる。
# pandas pdf.head(5) # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # PySpark sdf.head(5) # [Row(SepalLength=5.1, SepalWidth=3.5, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.9, SepalWidth=3.0, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.7, SepalWidth=3.2, PetalLength=1.3, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.6, SepalWidth=3.1, PetalLength=1.5, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=5.0, SepalWidth=3.6, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa')] # pandas type(pdf.head(5)) # <class 'pandas.core.frame.DataFrame'> # PySpark type(sdf.head(5)) # <type 'list'>
各カラムのデータ型の確認には dtypes。
# pandas pdf.dtypes # SepalLength float64 # SepalWidth float64 # PetalLength float64 # PetalWidth float64 # Species object # dtype: object # PySpark sdf.dtypes # [('SepalLength', 'double'), ('SepalWidth', 'double'), # ('PetalLength', 'double'), ('PetalWidth', 'double'), # ('Species', 'string')]
要約統計量の確認は describe。
# pandas pdf.describe() # SepalLength SepalWidth PetalLength PetalWidth # count 150.000000 150.000000 150.000000 150.000000 # mean 5.843333 3.054000 3.758667 1.198667 # std 0.828066 0.433594 1.764420 0.763161 # min 4.300000 2.000000 1.000000 0.100000 # 25% 5.100000 2.800000 1.600000 0.300000 # 50% 5.800000 3.000000 4.350000 1.300000 # 75% 6.400000 3.300000 5.100000 1.800000 # max 7.900000 4.400000 6.900000 2.500000 # PySpark sdf.describe().show() # summary SepalLength SepalWidth PetalLength PetalWidth # count 150 150 150 150 # mean 5.843333333333334 3.0540000000000003 3.758666666666666 1.1986666666666668 # stddev 0.8253012917851317 0.4321465800705415 1.7585291834055206 0.760612618588172 # min 4.3 2.0 1.0 0.1 # max 7.9 4.4 6.9 2.5
列操作
列名操作
参照と変更。PySpark ではプロパティへの代入は不可。
# pandas pdf.columns # Index([u'SepalLength', u'SepalWidth', u'PetalLength', u'PetalWidth', u'Species'], dtype='object') # PySpark sdf.columns # [u'SepalLength', u'SepalWidth', u'PetalLength', u'PetalWidth', u'Species'] # pandas pdf.columns = [1, 2, 3, 4, 5] pdf.columns # Int64Index([1, 2, 3, 4, 5], dtype='int64') # PySpark sdf.columns = [1, 2, 3, 4, 5] # AttributeError: can't set attribute
マッピングによって列名を変更するには、pandas では DataFrame.rename。
# pandas pdf.columns # Int64Index([1, 2, 3, 4, 5], dtype='int64') pdf = pdf.rename(columns={1:'SepalLength', 2:'SepalWidth', 3:'PetalLength', 4:'PetalWidth', 5:'Species'}) pdf # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns]
PySpark では DataFrame.withColumnRenamed。
# PySpark sdf.withColumnRenamed('Species', 'xxx') # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double, xxx: string]
補足 非破壊的な処理のため、反映には代入が必要。ここでは列名変更したくないので代入しない。
列名による列選択
pandas.DataFrame の列選択では 当該の列のデータを含む Series が返ってくる。
# pandas pdf.Species # 0 setosa # 1 setosa # 2 setosa # 3 setosa # 4 setosa # ... # 145 virginica # 146 virginica # 147 virginica # 148 virginica # 149 virginica # Name: Species, dtype: object pdf['Species'] # 略
PySpark では、列の属性を表現する Column インスタンスが返ってくる。
# PySpark sdf.Species # Column<Species> sdf['Species'] # Column<Species>
pandas、PySpark いずれも、文字列ではなくリストを渡せば その列を DataFrame としてスライシングする。
# pandas pdf[['PetalWidth']] # PetalWidth # 0 0.2 # 1 0.2 # 2 0.2 # 3 0.2 # 4 0.2 # .. ... # 145 2.3 # 146 1.9 # 147 2.0 # 148 2.3 # 149 1.8 # # [150 rows x 1 columns] # PySpark sdf[['PetalWidth']] # DataFrame[PetalWidth: double] # pandas pdf[['PetalWidth', 'PetalLength']] # PetalWidth PetalLength # 0 0.2 1.4 # 1 0.2 1.4 # 2 0.2 1.3 # 3 0.2 1.5 # 4 0.2 1.4 # .. ... ... # 145 2.3 5.2 # 146 1.9 5.0 # 147 2.0 5.2 # 148 2.3 5.4 # 149 1.8 5.1 # # [150 rows x 2 columns] # PySpark sdf[['PetalWidth', 'PetalLength']] # DataFrame[PetalWidth: double, PetalLength: double]
PySpark では DataFrame.select でもよい。
# PySpark sdf.select(sdf['PetalWidth'], sdf['PetalLength']) # DataFrame[PetalWidth: double, PetalLength: double]
真偽値リストによる列選択
自分はあまり使わないのだが、R {dplyr} との比較という観点で。
indexer = [False, False, True, True, False] # pandas pdf.loc[:, indexer] # PetalLength PetalWidth # 0 1.4 0.2 # 1 1.4 0.2 # 2 1.3 0.2 # 3 1.5 0.2 # 4 1.4 0.2 # .. ... ... # 145 5.2 2.3 # 146 5.0 1.9 # 147 5.2 2.0 # 148 5.4 2.3 # 149 5.1 1.8 # # [150 rows x 2 columns] # PySpark sdf[[c for c, i in zip(sdf.columns, indexer) if i is True]] # DataFrame[PetalLength: double, PetalWidth: double]
列の属性による列選択
列の型が pandas の場合は float、PySpark の場合は double の列のみ取り出す。
# pandas pdf.loc[:, pdf.dtypes == float] # SepalLength SepalWidth PetalLength PetalWidth # 0 5.1 3.5 1.4 0.2 # 1 4.9 3.0 1.4 0.2 # 2 4.7 3.2 1.3 0.2 # 3 4.6 3.1 1.5 0.2 # 4 5.0 3.6 1.4 0.2 # .. ... ... ... ... # 145 6.7 3.0 5.2 2.3 # 146 6.3 2.5 5.0 1.9 # 147 6.5 3.0 5.2 2.0 # 148 6.2 3.4 5.4 2.3 # 149 5.9 3.0 5.1 1.8 # # [150 rows x 4 columns] # PySpark sdf[[c for c, type in sdf.dtypes if type == 'double']] # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double]
行操作
値の条件による行選択
# pandas pdf[pdf['Species'] == 'virginica'] # SepalLength SepalWidth PetalLength PetalWidth Species # 100 6.3 3.3 6.0 2.5 virginica # 101 5.8 2.7 5.1 1.9 virginica # 102 7.1 3.0 5.9 2.1 virginica # 103 6.3 2.9 5.6 1.8 virginica # 104 6.5 3.0 5.8 2.2 virginica # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [50 rows x 5 columns] # PySpark sdf[sdf['Species'] == 'virginica'].show() # SepalLength SepalWidth PetalLength PetalWidth Species # 6.3 3.3 6.0 2.5 virginica # 5.8 2.7 5.1 1.9 virginica # 7.1 3.0 5.9 2.1 virginica # 6.3 2.9 5.6 1.8 virginica # .. ... ... ... ... # 6.5 3.0 5.5 1.8 virginica # 7.7 3.8 6.7 2.2 virginica # 7.7 2.6 6.9 2.3 virginica # 6.0 2.2 5.0 1.5 virginica
PySpark では DataFrame.filter でもよい。
# PySpark sdf.filter(sdf['Species'] == 'virginica').show() # 略
行番号による行選択
PySpark では 冒頭の n 行を抽出したりはできるが、適当な行選択ではできない、、、と思う。PySpark でどうしてもやりたければ index にあたる列を追加 -> 列の値で選択。
4/27追記 DataFrame.collect で Row のリストに変換 -> 再度 DataFrame 化すればスキーマ変更せずにできる、、、が、マスタでデータを処理することになるのでよい方法ではなさそう。
# pandas pdf.loc[[2, 3, 4]] # SepalLength SepalWidth PetalLength PetalWidth Species # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # PySpark [c for i, c in enumerate(sdf.collect()) if i in (2, 3 ,4)] # [Row(SepalLength=4.7, SepalWidth=3.2, PetalLength=1.3, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.6, SepalWidth=3.1, PetalLength=1.5, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=5.0, SepalWidth=3.6, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa')] sqlContext.createDataFrame([c for i, c in enumerate(sdf.collect()) if i in (2, 3 ,4)]).show() # SepalLength SepalWidth PetalLength PetalWidth Species # 4.7 3.2 1.3 0.2 setosa # 4.6 3.1 1.5 0.2 setosa # 5.0 3.6 1.4 0.2 setosa
ランダムサンプリング
pandas ではindex をサンプリングしてスライス。
import random # pandas pdf.loc[random.sample(pdf.index, 5)] # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 64 6.1 2.9 4.7 1.4 versicolor # 17 5.4 3.9 1.3 0.4 setosa # 14 4.3 3.0 1.1 0.1 setosa # 4 4.6 3.1 1.5 0.2 setosa # 146 6.7 3.0 5.2 2.3 virginica
PySpark では DataFrame.sample。この関数、fraction の確率で行ごとに選択しているようで、サンプリングするたびに抽出される行数が変わる ( 行数指定でランダムサンプリング、という処理はできなさそう )。
# PySpark sdf.sample(False, 5.0 / sdf.count()).show() # SepalLength SepalWidth PetalLength PetalWidth Species # 5.4 3.4 1.7 0.2 setosa # 5.7 2.8 4.5 1.3 versicolor # 6.2 2.8 4.8 1.8 virginica # 6.1 3.0 4.9 1.8 virginica
補足 今後、pandas にもサンプリングメソッド DataFrame.sample が実装予定 #9666。
列の追加
pandas では DataFrame に対して 直接 新しい列を追加できるが、PySpark ではできない。
# 破壊的な処理になるのでコピー pdf_temp = pdf.copy() # pandas pdf_temp['PetalMult'] = pdf_temp['PetalWidth'] * pdf_temp['PetalLength'] pdf_temp # SepalLength SepalWidth PetalLength PetalWidth Species PetalMult # 0 5.1 3.5 1.4 0.2 setosa 0.28 # 1 4.9 3.0 1.4 0.2 setosa 0.28 # 2 4.7 3.2 1.3 0.2 setosa 0.26 # 3 4.6 3.1 1.5 0.2 setosa 0.30 # 4 5.0 3.6 1.4 0.2 setosa 0.28 # .. ... ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica 11.96 # 146 6.3 2.5 5.0 1.9 virginica 9.50 # 147 6.5 3.0 5.2 2.0 virginica 10.40 # 148 6.2 3.4 5.4 2.3 virginica 12.42 # 149 5.9 3.0 5.1 1.8 virginica 9.18 # # [150 rows x 6 columns] # PySpark sdf['PetalMult'] = sdf['PetalWidth'] * sdf['PetalLength'] # TypeError: 'DataFrame' object does not support item assignment
PySpark での列追加は DataFrame.withColumn。
# PySpark sdf.withColumn('PetalMult', sdf.PetalWidth * sdf.PetalLength).show() # SepalLength SepalWidth PetalLength PetalWidth Species PetalMult # 5.1 3.5 1.4 0.2 setosa 0.27999999999999997 # 4.9 3.0 1.4 0.2 setosa 0.27999999999999997 # 4.7 3.2 1.3 0.2 setosa 0.26 # 4.6 3.1 1.5 0.2 setosa 0.30000000000000004 # .. .. ... ... ... ... # 5.4 3.9 1.3 0.4 setosa 0.52 # 5.1 3.5 1.4 0.3 setosa 0.42 # 5.7 3.8 1.7 0.3 setosa 0.51 # 5.1 3.8 1.5 0.3 setosa 0.44999999999999996
補足 pandas v0.16.0 以降では 類似のメソッド DataFrame.assign が追加。
# pandas pdf.assign(PetalMult=pdf['PetalWidth'] * pdf['PetalLength']) # 略
まとめ
PySpark と pandas のデータ操作を整理した。
PySpark、上のような基本的な処理は pandas と似たやり方で直感的に使える感じだ。
大規模処理は PySpark、細かい取り回しが必要なものは pandas でうまく併用できるとよさそう。
4/29追記 続きはこちら。

Learning Spark: Lightning-Fast Big Data Analysis
- 作者: Holden Karau,Andy Konwinski,Patrick Wendell,Matei Zaharia
- 出版社/メーカー: O'Reilly Media
- 発売日: 2015/01/28
- メディア: Kindle版
- この商品を含むブログを見る
Python pandas / scikit-learn 向けのちょっとしたパッケージ作った <前編>
こちらの続き。
pandas のデータを scikit-learn でうまく処理するためのパッケージを作ったのでその使い方を書きたい。今回は 適当なデータをファイルから読み込み -> 前処理してクラスタリングする、という例を書く。
このパッケージ 基本的には pandas と使い方は同じなので、以下 追加機能にあたる部分を中心に記載する。
- データは 説明変数 / 目的変数の定義をもち、それぞれプロパティから参照 / 上書きできる。
scikit-learnの各サブパッケージにプロパティからアクセスできる。また、メソッド呼び出し時の引数を省略できる。
インストール
pip install pandas_ml
補足 今後 別パッケージへの拡張を考えているため、scikit-learn への依存は optional にしている。そのため、scikit-learn をインストールしていない方は別途インストールを。
データの読み込み
こういう例を書くとき、 iris 以外のデータがぱっと出てくる人ってかっこいいと思う。
# おまじない import numpy as np import pandas as pd pd.options.display.max_rows = 8 state = np.random.RandomState(1) # csv からデータ読み込み # http://aima.cs.berkeley.edu/data/iris.csv names = ['Sepal.Length', 'Sepal.Width', 'Petal.Length', 'Petal.Width', 'Species'] iris = pd.read_csv('iris.csv', header=None, names=names) iris # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # .. ... ... ... ... ... # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns] type(iris) # <class 'pandas.core.frame.DataFrame'>
上で読み込んだデータを pandas_ml で処理するには、一度 pandas_ml.ModelFrame インスタンスに変換する必要がある。ModelFrame を作成するには、
- 第一引数には、
pd.DataFrameに変換できる何か (DataFrame自体, 辞書など) を渡す targetキーワードには、目的変数として扱いたい 列名の文字列、もしくはpd.Seriesに変換できる何か (Series、もしくはリスト-like )を渡す
詳しくは ドキュメント を。
今回は 第一引数には pd.DataFrame、 target には 目的変数として扱いたい列名である "Species" を指定する。
import pandas_ml as pdml df = pdml.ModelFrame(iris, target='Species') type(df) # <class 'pandas_ml.core.frame.ModelFrame'> # ModelFrame は DataFrame のサブクラス isinstance(df, pd.DataFrame) # True
補足 target キーワードを指定しない場合、目的変数をもたない ModelFrame インスタンスが作成される。
作成した ModelFrame は pd.DataFrame と同じように扱える。一部、 pd.DataFrame にないメソッド / プロパティを持つ。
df # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # .. ... ... ... ... ... # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns] # データが目的変数を持っているかを確認 df.has_target() # True # 説明変数は data プロパティで参照 df.data # Sepal.Length Sepal.Width Petal.Length Petal.Width # 0 5.1 3.5 1.4 0.2 # 1 4.9 3.0 1.4 0.2 # 2 4.7 3.2 1.3 0.2 # 3 4.6 3.1 1.5 0.2 # .. ... ... ... ... # 146 6.3 2.5 5.0 1.9 # 147 6.5 3.0 5.2 2.0 # 148 6.2 3.4 5.4 2.3 # 149 5.9 3.0 5.1 1.8 # # [150 rows x 4 columns] # 目的変数は target プロパティで参照 df.target # 0 setosa # 1 setosa # ... # 148 virginica # 149 virginica # Name: Species, Length: 150, dtype: object # 目的変数の列名を取得 df.target_name # 'Species' # これも一緒 (target の name 属性を見ているので) df.target.name # 'Species'
このとき、ModelFrame.data は 目的変数のない ModelFrame インスタンスに、また ModelFrame.target は pd.Series を継承した ModelSeries インスタンスになっている。
type(df.data) # <class 'pandas_ml.core.frame.ModelFrame'> df.data.has_target() # False type(df.target) # <class 'pandas_ml.core.series.ModelSeries'> isinstance(df.target, pd.Series) # True
データの前処理
いくつか前処理を行う。まず、target についているラベル ( "setosa", "versicolor", "virginica" ) を数値 (0, 1, 2) に変換する。この処理は pandas の map がカンタン。
df.target.map({'setosa': 0, 'versicolor': 1, 'virginica': 2}) # 0 0 # 1 0 # ... # 148 2 # 149 2 # Name: Species, Length: 150, dtype: int64 # これをそのまま target に代入すればよい df.target = df.target.map({'setosa': 0, 'versicolor': 1, 'virginica': 2}) df # Species Sepal.Length Sepal.Width Petal.Length Petal.Width # 0 0 5.1 3.5 1.4 0.2 # 1 0 4.9 3.0 1.4 0.2 # 2 0 4.7 3.2 1.3 0.2 # 3 0 4.6 3.1 1.5 0.2 # .. ... ... ... ... ... # 146 2 6.3 2.5 5.0 1.9 # 147 2 6.5 3.0 5.2 2.0 # 148 2 6.2 3.4 5.4 2.3 # 149 2 5.9 3.0 5.1 1.8 # # [150 rows x 5 columns]
次に、(特に理由はないが) "Sepal.Length" と "Sepal.Width" のみを正規化する。これは pandas にはない処理なので、 sklearn.preprocessing.normalize を使う。
ModelFrame は scikit-learn の各サブパッケージに対応したプロパティ (アクセサ) を持っており、サブパッケージをロードしなくても 対応する関数をメソッドとして利用できる。このとき、呼び出し元のデータが自動的に引数として渡されるため データの引数指定は不要。
サブパッケージとプロパティの対応一覧はドキュメント を。
df[['Sepal.Length', 'Sepal.Width']].preprocessing.normalize() # Sepal.Length Sepal.Width # 0 0.824513 0.565842 # 1 0.852851 0.522154 # 2 0.826599 0.562791 # 3 0.829266 0.558853 # .. ... ... # 146 0.929491 0.368846 # 147 0.907959 0.419058 # 148 0.876812 0.480833 # 149 0.891385 0.453247 # # [150 rows x 2 columns] # これもそのまま代入すればよい df[['Sepal.Length', 'Sepal.Width']] = df[['Sepal.Length', 'Sepal.Width']].preprocessing.normalize() df # Species Sepal.Length Sepal.Width Petal.Length Petal.Width # 0 0 0.824513 0.565842 1.4 0.2 # 1 0 0.852851 0.522154 1.4 0.2 # 2 0 0.826599 0.562791 1.3 0.2 # 3 0 0.829266 0.558853 1.5 0.2 # .. ... ... ... ... ... # 146 2 0.929491 0.368846 5.0 1.9 # 147 2 0.907959 0.419058 5.2 2.0 # 148 2 0.876812 0.480833 5.4 2.3 # 149 2 0.891385 0.453247 5.1 1.8 # # [150 rows x 5 columns]
クラスタリング
ModelFrame.cluster.k_means で、sklearn.cluster.k_means を呼び出せる。このメソッドの返り値は centroid, label, inertia の 3つになる。
centroid, label, inertia = df.cluster.k_means(n_clusters=3, random_state=state) centroid # array([[ 0.82602947, 0.56251635, 1.464 , 0.244 ], # [ 0.91055414, 0.41171559, 5.59583333, 2.0375 ], # [ 0.90545279, 0.42259059, 4.26923077, 1.34230769]]) label # 0 0 # 1 0 # ... # 148 1 # 149 1 # Length: 150, dtype: int32 inertia # 31.598367459843072
もしくは、ModelFrame.cluster.KMeans から sklearn.cluster.KMeans インスタンスを作成して fit -> predict する。メソッドの呼び出しを ModelFrame 側から行うことで引数を省略できる。
kmeans = df.cluster.KMeans(n_clusters=3, random_state=state) df.fit(kmeans) # KMeans(copy_x=True, init='k-means++', max_iter=300, n_clusters=3, n_init=10, # n_jobs=1, precompute_distances=True, # random_state=<mtrand.RandomState object at 0x104053410>, tol=0.0001, # verbose=0) df.predict(kmeans) # 0 1 # 1 1 # ... # 148 0 # 149 0 # Length: 150, dtype: int32
直前に利用された estimator は ModelFrame.estimator から参照できる。また、直近のクラスタリング結果は ModelFrame.predicted にキャッシュされている。
df.estimator # KMeans(copy_x=True, init='k-means++', max_iter=300, n_clusters=3, n_init=10, # n_jobs=1, precompute_distances=True, # random_state=<mtrand.RandomState object at 0x104053410>, tol=0.0001, # verbose=0) df.predicted # 0 1 # 1 1 # ... # 148 0 # 149 0 # Length: 150, dtype: int32
ModelFrame.metrics はsklearn.metrics の各メソッドを引数省略して呼び出せる。このとき、内部的には上のキャッシュが利用されている。
df.metrics.completeness_score()
# 0.8643954288752761
引数省略時の内部処理について
scikit-learn の各メソッドと 内部的に渡すデータの対応関係一覧は以下ドキュメントに記載している。何かおかしなことをやっていたら GitHub からご指摘ください。
まとめ
pandas_ml を利用して データの読み込み / 前処理 / クラスタリングを行う方法を記載した。
次は、Pipeline, Cross Validation, Grid Search あたりを。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (10件) を見る
9/12追記 パッケージ名変更。
Python pandas のデータを scikit-learn でうまいこと処理したい
はじめに
Python で機械学習する場合、pandas で前処理したデータを scikit-learn で処理する、というケースが多いと思う。pandas, scikit-learn には それぞれ 簡単にできる / できない処理があるので、うまいこと連携できるとうれしい。
scikit-learn の各メソッドは numpy.ndarray に対する処理を前提にしているため、pandas のデータ形式 (DataFrame や Series) を渡すと 内部で ndarray に変換して結果を返してくる。そのため、結果に対して 直接 pandas の処理を続けることはできない。
ndarray で処理すりゃいいじゃん、、というむきもあるが、自分はどうしても pandas で処理がしたいんだ。とりあえず、pandas のデータをできるだけ維持したい、というモチベーションがあるものとして処理のサンプルを書く。上述のとおり scikit-learn で何か処理をするたびに ndarray が返ってくるので、これを 都度 pandas のデータ形式に戻してやる必要がある。
pandas / scikit-learn 標準での処理
このサンプルで行う処理の流れは、
- データの準備:
sklearn.datasetsのデータセットを読み込み - 前処理:
sklearn.preprocessingで説明変数を二値化し、sklearn.cross_validationでデータを訓練用 / テスト用に分割 - 分類 / 評価:
sklearn.svmで 分類し、sklearn.metricsで評価
# おまじない import numpy as np import pandas as pd pd.options.display.max_rows = 10 pd.options.display.max_columns = 15 state = np.random.RandomState(1)
データの準備
scikit-learn 付属の手書き数字のデータセットを datasets.load_digits で読み込む。
import sklearn.datasets as datasets digits = datasets.load_digits() type(digits) # <class 'sklearn.datasets.base.Bunch'> # 説明変数 digits.data.shape # (1797, 64) # 目的変数 digits.target.shape # (1797,) # 目的変数を Series に変換 target = pd.Series(digits.target) target # 0 0 # 1 1 # 2 2 # ... # 1794 8 # 1795 9 # 1796 8 # Length: 1797, dtype: int64 # 説明変数を DataFrame に変換 data = pd.DataFrame(digits.data) data # 0 1 2 3 4 5 6 ... 57 58 59 60 61 62 63 # 0 0 0 5 13 9 1 0 ... 0 6 13 10 0 0 0 # 1 0 0 0 12 13 5 0 ... 0 0 11 16 10 0 0 # 2 0 0 0 4 15 12 0 ... 0 0 3 11 16 9 0 # 3 0 0 7 15 13 1 0 ... 0 7 13 13 9 0 0 # 4 0 0 0 1 11 0 0 ... 0 0 2 16 4 0 0 # ... .. .. .. .. .. .. .. ... .. .. .. .. .. .. .. # 1792 0 0 4 10 13 6 0 ... 0 2 14 15 9 0 0 # 1793 0 0 6 16 13 11 1 ... 0 6 16 14 6 0 0 # 1794 0 0 1 11 15 1 0 ... 0 2 9 13 6 0 0 # 1795 0 0 2 10 7 0 0 ... 0 5 12 16 12 0 0 # 1796 0 0 10 14 8 1 0 ... 1 8 12 14 12 1 0 # # [1797 rows x 64 columns]
前処理
まずは 説明変数を preprocessing.binarize で二値化して
import sklearn.preprocessing as pp data = pd.DataFrame(pp.binarize(data.values, threshold=5)) data # 0 1 2 3 4 5 6 ... 57 58 59 60 61 62 63 # 0 0 0 0 1 1 0 0 ... 0 1 1 1 0 0 0 # 1 0 0 0 1 1 0 0 ... 0 0 1 1 1 0 0 # 2 0 0 0 0 1 1 0 ... 0 0 0 1 1 1 0 # 3 0 0 1 1 1 0 0 ... 0 1 1 1 1 0 0 # 4 0 0 0 0 1 0 0 ... 0 0 0 1 0 0 0 # ... .. .. .. .. .. .. .. ... .. .. .. .. .. .. .. # 1792 0 0 0 1 1 1 0 ... 0 0 1 1 1 0 0 # 1793 0 0 1 1 1 1 0 ... 0 1 1 1 1 0 0 # 1794 0 0 0 1 1 0 0 ... 0 0 1 1 1 0 0 # 1795 0 0 0 1 1 0 0 ... 0 0 1 1 1 0 0 # 1796 0 0 1 1 1 0 0 ... 0 1 1 1 1 0 0 # # [1797 rows x 64 columns]
cross_validation.train_test_split で 訓練用とテスト用のデータに分割する。
import sklearn.cross_validation as crv train_data, test_data, train_target, test_target = crv.train_test_split(data.values, target.values, random_state=state) train_data = pd.DataFrame(train_data) test_data = pd.DataFrame(test_data) train_target = pd.Series(train_target) test_target = pd.Series(test_target) train_data # 0 1 2 3 4 5 6 ... 57 58 59 60 61 62 63 # 0 0 0 1 1 1 0 0 ... 0 1 1 1 0 0 0 # 1 0 0 0 1 1 0 0 ... 0 0 1 1 1 1 0 # 2 0 0 0 0 1 0 0 ... 0 0 0 1 1 0 0 # 3 0 0 0 1 1 1 1 ... 0 0 1 0 0 0 0 # 4 0 0 1 1 1 0 0 ... 0 0 1 1 1 0 0 # ... .. .. .. .. .. .. .. ... .. .. .. .. .. .. .. # 1342 0 0 0 1 1 0 0 ... 0 0 1 1 1 0 0 # 1343 0 0 0 0 1 0 0 ... 0 0 0 1 0 0 0 # 1344 0 0 1 1 1 0 0 ... 0 1 1 1 1 0 0 # 1345 0 0 0 0 1 1 1 ... 0 0 0 1 1 0 0 # 1346 0 0 1 1 1 1 1 ... 0 1 1 1 0 0 0 # # [1347 rows x 64 columns] test_data # 0 1 2 3 4 5 6 ... 57 58 59 60 61 62 63 # 0 0 0 1 1 1 0 0 ... 0 0 1 1 0 0 0 # 1 0 1 1 1 1 1 1 ... 1 1 1 0 0 0 0 # 2 0 0 0 1 1 0 0 ... 0 0 1 1 1 0 0 # 3 0 0 0 1 1 1 1 ... 0 0 1 0 0 0 0 # 4 0 0 0 0 1 1 0 ... 0 0 0 1 1 0 0 # .. .. .. .. .. .. .. .. ... .. .. .. .. .. .. .. # 445 0 0 1 1 1 0 0 ... 0 1 1 1 1 0 0 # 446 0 0 1 1 1 0 0 ... 0 1 1 1 1 0 0 # 447 0 0 1 1 1 0 0 ... 0 1 1 0 0 0 0 # 448 0 0 1 1 1 0 0 ... 0 1 1 1 0 0 0 # 449 0 0 1 1 1 0 0 ... 0 1 1 1 0 0 0 # # [450 rows x 64 columns]
分類 / 評価
svm.LinearSVC で分類する。
import sklearn.svm as svm svc = svm.LinearSVC(random_state=state) # 訓練データをフィット svc.fit(train_data, train_target) # LinearSVC(C=1.0, class_weight=None, dual=True, fit_intercept=True, # intercept_scaling=1, loss='l2', multi_class='ovr', penalty='l2', # random_state=<mtrand.RandomState object at 0x103f28350>, tol=0.0001, # verbose=0) # テストデータを分類 predicted = svc.predict(test_data) predicted = pd.Series(predicted) predicted # 0 1 # 1 5 # 2 0 # ... # 447 7 # 448 8 # 449 5 # Length: 450, dtype: int64
最後に、得られた結果を metrics で評価する。
metrics.confusion_matrix: 混同行列metrics.accuracy_score: 正答率
import sklearn.metrics as metrics metrics.confusion_matrix(test_target, predicted) # [[51 0 0 0 1 0 0 0 1 0] # [ 0 38 0 0 0 1 0 0 3 0] # [ 0 0 38 1 0 0 1 1 0 0] # [ 0 1 1 45 0 2 1 1 1 0] # [ 0 0 0 0 45 0 1 1 0 0] # [ 0 0 2 0 0 36 0 0 0 1] # [ 0 0 0 0 0 0 43 0 0 0] # [ 0 0 0 0 1 0 0 47 0 0] # [ 0 2 1 1 1 0 0 0 31 1] # [ 0 2 0 1 1 3 0 1 1 39]] metrics.accuracy_score(test_target, predicted) # 0.917777777778
うん、、、めんどくさい。この めんどくささは 以下 3 点に起因していると思う。
scikit-learn返り値のndarrayを 都度pandasのデータ形式に変換する必要がある ( このとき、データの属性、たとえばカラム名を維持したい場合は毎回columnsを引数として渡す必要がある )scikit-learnの個々のモジュールを都度 ロードする必要がある (これはpandas云々は関係ないが)scikit-learnにいちいち似たような引数を渡さなければならない ( 目的変数が変わることはないはずなので、一度 定義したものが使いまわせるとうれしい )
というわけで
作りました。
ドキュメントはこちら。
こちらを使うと 上のサンプルは以下のように書ける。
state = np.random.RandomState(1) import pandas_ml as pdml import sklearn.datasets as datasets # データの読み込み / データ型の作成 (ModelFrame は DataFrame を継承したクラス) df = pdml.ModelFrame(datasets.load_digits()) # 説明変数 df.data # 0 1 2 3 4 5 6 ... 57 58 59 60 61 62 63 # 0 0 0 5 13 9 1 0 ... 0 6 13 10 0 0 0 # 1 0 0 0 12 13 5 0 ... 0 0 11 16 10 0 0 # 2 0 0 0 4 15 12 0 ... 0 0 3 11 16 9 0 # 3 0 0 7 15 13 1 0 ... 0 7 13 13 9 0 0 # 4 0 0 0 1 11 0 0 ... 0 0 2 16 4 0 0 # ... .. .. .. .. .. .. .. ... .. .. .. .. .. .. .. # 1792 0 0 4 10 13 6 0 ... 0 2 14 15 9 0 0 # 1793 0 0 6 16 13 11 1 ... 0 6 16 14 6 0 0 # 1794 0 0 1 11 15 1 0 ... 0 2 9 13 6 0 0 # 1795 0 0 2 10 7 0 0 ... 0 5 12 16 12 0 0 # 1796 0 0 10 14 8 1 0 ... 1 8 12 14 12 1 0 # # [1797 rows x 64 columns] # 目的変数 df.target # 0 0 # 1 1 # 2 2 # ... # 1794 8 # 1795 9 # 1796 8 # Name: .target, Length: 1797, dtype: int64 # 二値化 df.data = df.data.preprocessing.binarize(threshold=5) # 訓練用とテスト用のデータに分割 train_df, test_df = df.cross_validation.train_test_split(random_state=state) train_df # .target 0 1 2 3 4 5 ... 57 58 59 60 61 62 63 # 0 2 0 0 1 1 1 0 ... 0 1 1 1 0 0 0 # 1 6 0 0 0 1 1 0 ... 0 0 1 1 1 1 0 # 2 6 0 0 0 0 1 0 ... 0 0 0 1 1 0 0 # 3 7 0 0 0 1 1 1 ... 0 0 1 0 0 0 0 # 4 1 0 0 1 1 1 0 ... 0 0 1 1 1 0 0 # ... ... .. .. .. .. .. .. ... .. .. .. .. .. .. .. # 1342 8 0 0 0 1 1 0 ... 0 0 1 1 1 0 0 # 1343 4 0 0 0 0 1 0 ... 0 0 0 1 0 0 0 # 1344 9 0 0 1 1 1 0 ... 0 1 1 1 1 0 0 # 1345 1 0 0 0 0 1 1 ... 0 0 0 1 1 0 0 # 1346 5 0 0 1 1 1 1 ... 0 1 1 1 0 0 0 # # [1347 rows x 65 columns] # 訓練データをフィット svc = train_df.svm.LinearSVC(random_state=state) train_df.fit(svc) # LinearSVC(C=1.0, class_weight=None, dual=True, fit_intercept=True, # intercept_scaling=1, loss='l2', multi_class='ovr', penalty='l2', # random_state=<mtrand.RandomState object at 0x103f3e350>, tol=0.0001, # verbose=0) # テストデータを分類 predicted = test_df.predict(svc) predicted # 0 1 # 1 5 # 2 0 # ... # 447 7 # 448 8 # 449 5 # Length: 450, dtype: int64 # 評価 test_df.metrics.confusion_matrix() # Predicted 0 1 2 3 4 5 6 7 8 9 # Target # 0 51 0 0 0 1 0 0 0 1 0 # 1 0 38 0 0 0 1 0 0 3 0 # 2 0 0 38 1 0 0 1 1 0 0 # 3 0 1 1 45 0 2 1 1 1 0 # 4 0 0 0 0 45 0 1 1 0 0 # 5 0 0 2 0 0 36 0 0 0 1 # 6 0 0 0 0 0 0 43 0 0 0 # 7 0 0 0 0 1 0 0 47 0 0 # 8 0 2 1 1 1 0 0 0 31 1 # 9 0 2 0 1 1 3 0 1 1 39 test_df.metrics.accuracy_score() # 0.9177777777777778
先に記載の課題は以下のように改善される。
scikit-learnの処理をデータに対して適用することで、pandasのデータ形式 (を継承したデータ構造) を維持できる- データのプロパティから
scikit-learnの各モジュールへアクセスできる。モジュールは必要になったときにロードされる - メソッド呼び出しの際、目的変数 / 説明変数を省略できる
もう少しまともな解説を別の記事で書きます。
3/19追記 続きはこちら。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (10件) を見る
9/12追記 パッケージ名変更。
Python pandas 日本語環境向けのちょっとしたパッケージ作った
最近の空き時間は GitHub で草植えをしている。まずは pandas を日本語環境で使う場合に たまに必要になる処理をまとめた パッケージを作った。
インストール
pip install japandas
機能
機能の一覧はこちら。
- 日時処理
- 日本語日付のパース
- 日本の祝日カレンダー
- 文字列処理
- Unicode 正規化
- 全角/半角変換
- リモートデータアクセス
詳細はドキュメントを。
日時処理
日本語日付のパース
pandas には 日付らしい入力を適切に処理してくれる pandas.to_datetime があるが、これは日本語の日付 ( "XX年XX月XX日" とか ) に対応していない。例えば 以下のような入力は日時としてパースされず 文字列のまま残ってしまう。
import pandas as pd pd.to_datetime(u'2014年11月30日') # u'2014年11月30日'
japandas.to_datetime は pd.to_datetime を軽くラップし、以下のような日付をパースできるようにする。
'XX年XX月XX日''XX年XX月''XX年XX月XX日XX時XX分''XX年XX月XX日XX時XX分XX秒'
import japandas as jpd jpd.to_datetime(u'2015年3月1日') # Timestamp('2015-03-01 00:00:00') jpd.to_datetime([u'2014年11月30日13時25分', u'2014年11月30日14時38分']) # <class 'pandas.tseries.index.DatetimeIndex'> # [2014-11-30 13:25:00, 2014-11-30 14:38:00] # Length: 2, Freq: None, Timezone: None
補足 リスト-like な入力を処理する場合、日付のフォーマットは全て同じでなければならない。
参考 標準の pd.to_datetime で任意のフォーマットをパースするには、以下のように format オプションで書式を与えてやればよい。pd.to_datetime については、 Python pandas で日時関連のデータ操作をカンタンに - StatsFragments を。
pd.to_datetime(u'2014年11月30日', format=u'%Y年%m月%d日') # Timestamp('2014-11-30 00:00:00')
日本の祝日カレンダー
japandas.JapaneseHolidayCalendar は日本の祝日を定義したクラス。
calendar = jpd.JapaneseHolidayCalendar() calendar.holidays() # <class 'pandas.tseries.index.DatetimeIndex'> # [1970-01-01, ..., 2030-12-23] # Length: 969, Freq: None, Timezone: None
このカレンダー定義を使うと、定義に応じた営業日計算が以下のとおりできる。
参考 Time Series / Date functionality — pandas 0.16.2 documentation
import datetime cday = pd.offsets.CDay(calendar=calendar) # 4/29は祝日(昭和の日)なので無視されて加算 datetime.datetime(2014, 4, 28) + cday # Timestamp('2014-04-30 00:00:00') # 4/26は土曜日, 4/27は日曜日なので無視されて減算 datetime.datetime(2014, 4, 28) - cday # Timestamp('2014-04-25 00:00:00') # 5/4は日曜日, 5/5は祝日(こどもの日), 5/6は祝日(みどりの日/振替休日)なので無視されて加算 datetime.datetime(2014, 5, 3) + cday # Timestamp('2014-05-07 00:00:00') # 5/3は土曜日、前日は営業日なので通常の減算 datetime.datetime(2014, 5, 3) - cday # Timestamp('2014-05-02 00:00:00')
また、作成した Custom Business Day インスタンスは 指定した範囲の営業日を含む Index の生成にも使える。japandas.date_range は pd.date_range の日本語日付対応版。
indexer = jpd.date_range(u'2014年5月1日', u'2014年5月10日', freq=cday) indexer # <class 'pandas.tseries.index.DatetimeIndex'> # [2014-05-01, ..., 2014-05-09] # Length: 5, Freq: C, Timezone: None
この Index を使うと、カレンダー定義に応じてデータを抽出する処理が簡単に書ける。
import numpy as np # 処理したいデータを作成 df = pd.DataFrame(np.random.randn(10, 3), index=jpd.date_range(u'2014年5月1日', u'2014年5月10日', freq='D')) df # 0 1 2 # 2014-05-01 0.762453 -1.418762 -0.150073 # 2014-05-02 0.966500 -0.473888 0.272871 # 2014-05-03 0.473370 -1.282504 0.380449 # 2014-05-04 0.215411 0.220587 -1.088699 # 2014-05-05 0.286348 -1.069165 -1.471871 # 2014-05-06 -0.665438 -0.402046 -1.008051 # 2014-05-07 1.173935 2.080087 -2.279285 # 2014-05-08 -0.957195 0.746798 0.092214 # 2014-05-09 -0.259276 -0.775489 0.572525 # 2014-05-10 -0.910188 0.294136 0.020730 # カレンダー上 営業日のレコードを抽出 df.loc[indexer] # 0 1 2 # 2014-05-01 0.762453 -1.418762 -0.150073 # 2014-05-02 0.966500 -0.473888 0.272871 # 2014-05-07 1.173935 2.080087 -2.279285 # 2014-05-08 -0.957195 0.746798 0.092214 # 2014-05-09 -0.259276 -0.775489 0.572525 # カレンダー上 休日のレコードを抽出 df[~df.index.isin(indexer)] # 0 1 2 # 2014-05-03 0.473370 -1.282504 0.380449 # 2014-05-04 0.215411 0.220587 -1.088699 # 2014-05-05 0.286348 -1.069165 -1.471871 # 2014-05-06 -0.665438 -0.402046 -1.008051 # 2014-05-10 -0.910188 0.294136 0.020730
文字列処理
Unicode 正規化
Series.str.normalize を使うと、標準の unicodedata.normalize と同じ処理を 各値に対して適用できる。
s = pd.Series([u'アイウエオ', u'カキクケコ', u'ガギグゲゴ', u'ABCDE']) s # 0 アイウエオ # 1 カキクケコ # 2 ガギグゲゴ # 3 ABCDE # dtype: object s.str.normalize() # 0 アイウエオ # 1 カキクケコ # 2 ガギグゲゴ # 3 ABCDE # dtype: object
文字列以外の値は np.nan になる。これは str アクセサの他のメソッドと一緒。また、 Python 2.x 系の str ( unicode ではないもの) も処理できる。
s = pd.Series([u'アイウエオ', 23, u'ガギグゲゴ', None, 'AAA']) s.str.normalize() # 0 アイウエオ # 1 NaN # 2 ガギグゲゴ # 3 NaN # 4 AAA # dtype: object
全角/半角変換
正規化があればいらない気もするのだが、全角/半角変換用のメソッドもつけてみた。
Series.str.h2z: 半角 -> 全角へ変換Series.str.z2h: 全角 -> 半角へ変換
s = pd.Series([u'アイウエオ', u'ABC01', u'DE345']) z = s.str.h2z() z # 0 アイウエオ # 1 ABC01 # 2 DE345 # dtype: object z.str.z2h() # 0 アイウエオ # 1 ABC01 # 2 DE345 # dtype: object
参考 文字列処理全般については、 Python pandas で日時関連のデータ操作をカンタンに - StatsFragments を。
補足 全角/半角変換について、既存パッケージの実装は ベクトル化したとき少し遅くなりそうだったので再発明したのだが、速度的にはあまり意味はなかった。今後 既存パッケージに置き換えるか、Cython 化するかしたい 。また、str.normalize は標準にのせたい 。
リモートデータアクセス
以下の記事の内容を pandas.io.data.DataReader と同じ形式でラップしたもの。加えて .plot(kind='ohlc') も可能。
参考 Remote Data Access — pandas 0.16.2 documentation
jpd.DataReader(7203, 'yahoojp', start='2014-10-01', end='2014-10-05') # 始値 高値 安値 終値 出来高 調整後終値* # 日付 # 2014-10-01 6450 6559 6435 6500 14482100 6500 # 2014-10-02 6370 6423 6256 6275 15240200 6275 # 2014-10-03 6231 6309 6217 6290 10280100 6290
補足 ページ取得の際は毎回ウェイトを入れている。あまり長期間のデータ取得には使わないよう。
最後に
不具合、もしくは日本固有の機能要望があれば GitHub からください。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る
Python pandas で日本の株価情報取得とローソク足チャート描画
以下の記事を読んでいて、pandas 標準では日本株式の情報が直接とれないことに気づいたのでやり方をまとめたい。
この記事では以下 2 点の処理について書く。
補足 標準の v0.15.2 で簡単に動きをみているが、ちゃんとテストはしてない。特にローソク足チャートについては pandas の plot クラスにかなり依存しているため、バージョンが変わると動かなくなる可能性がある。
1. 株価の取得
まず、pandas には標準機能として外部サイトのデータを DataFrame で取得する機能 DataReader がある。DataReader では 日本株の情報は直接はとれないが、簡単に動きを書いておく。
DataReader での取得
DataReader では、0.15.2 現在で以下 6 つのデータソースをサポートしている。詳しくは公式ドキュメントを。
- Yahoo! Finance
- Google Finance
- St.Louis FED (FRED)
- Kenneth French’s data library
- World Bank
- Google Analytics
Yahoo! Finance, Google Finance いずれも東京証券取引所ではなく ニューヨーク証券取引所の情報を取ってくるもの。そのため、米国で上場している企業の情報は取れる (ドル建てになるが、、)。Yahoo! Finance 経由でトヨタ自動車の情報を取得してみると、
import numpy as np import pandas as pd import pandas.io.data as web start = '2014-10-01' toyota_nyse = web.DataReader('TM', 'yahoo', start=start) toyota_nyse.head() # Open High Low Close Volume Adj Close # Date # 2014-10-01 117.94 117.99 116.46 116.65 676800 116.65 # 2014-10-02 115.02 115.18 113.64 114.48 507300 114.48 # 2014-10-03 115.70 116.31 115.48 116.16 281500 116.16 # 2014-10-06 116.61 117.22 116.41 116.70 339200 116.70 # 2014-10-07 117.00 117.14 115.80 115.82 398400 115.82
補足 もう少し詳細な情報が取れる Yahoo! Finance Options という機能もある。
補足 今後、この DataReader は別パッケージとして分離される予定。
Yahoo ファイナンス からの取得
東京証券取引所の情報も上のような形で取りたい。が、日本版である Yahoo! ファイナンスには 米国の Yahoo! Finance が提供しているものと同じ API はないようだ。
そのため、DataReader と同じような感じで Yahoo! ファイナンス から情報を取得する get_quote_yahoojp 関数を書いた (コードは最下部に添付)。ロジックを簡単に書くと、
- API がないので スクレイピングで
- 複数の URL にまたがる情報を、順次
pd.read_htmlで読み込み - 読み込んだ 各
DataFrameをpd.concatで結合し - 日付データをパースして
indexに設定
参考 このあたりの機能を使った。
作成した get_quote_yahoojp を使って、同じくトヨタ自動車の情報をとると、
toyota_tse = get_quote_yahoojp(7203, start=start) toyota_tse.head() # Open High Low Close Volume Adj Close # Date # 2014-10-01 6450 6559 6435 6500 14482100 6500 # 2014-10-02 6370 6423 6256 6275 15240200 6275 # 2014-10-03 6231 6309 6217 6290 10280100 6290 # 2014-10-06 6370 6444 6345 6383 10966200 6383 # 2014-10-07 6380 6455 6351 6366 10129100 6366
2. ローソク足チャートの描画
株価をとったらチャートが描きたいので、ローソク足チャートを作成する関数 / クラスを定義した (コードは最下部に添付)。こちらは pandas のプロットフレームワーク上に実装したので、添付のコードを実行した後は DataFrame.plot(kind='ohlc') でローソク足チャートが描けるようになる。
補足 内部的には、matplotlib.finance.candlestick を使っているが、この関数を含む matplotlib.finance モジュールは matplotlib v1.4.0 で deprecate されており、今後どのような扱いになるのかわからない。
# 最下部に添付のコードを実行してから import matplotlib.pyplot as plt toyota_tse.plot(kind='ohlc')
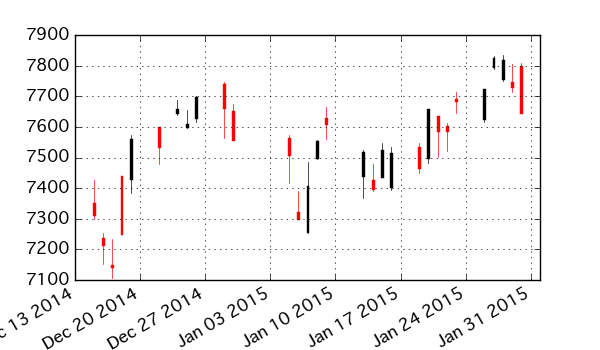
土日の間にスペースが空くのが嫌なら、一度 BusinessDay に変換してからプロット。
toyota_tse.asfreq('B').plot(kind='ohlc')
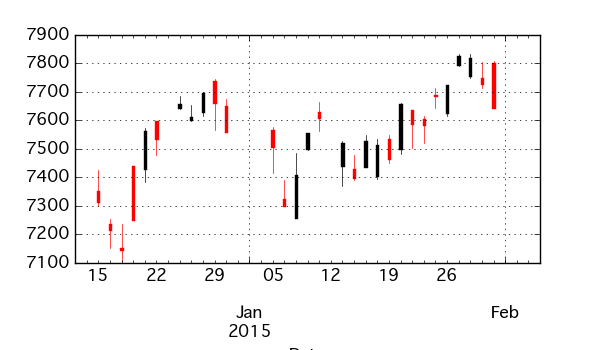
ついでだが、DatetimeIndex をもつ Series から呼び出すと ohlc にリサンプリングしてプロットするようにした。
np.random.seed(1) idx = pd.date_range(start='2015-01-05 09:00', freq='H', periods=1000) values = np.random.randint(low=-500, high=500, size=1000) s = pd.Series(10000 + np.cumsum(values), index=idx) s.head() # 2015-01-05 09:00:00 9537 # 2015-01-05 10:00:00 9272 # 2015-01-05 11:00:00 9680 # 2015-01-05 12:00:00 9252 # 2015-01-05 13:00:00 9519 s.plot(kind='ohlc')
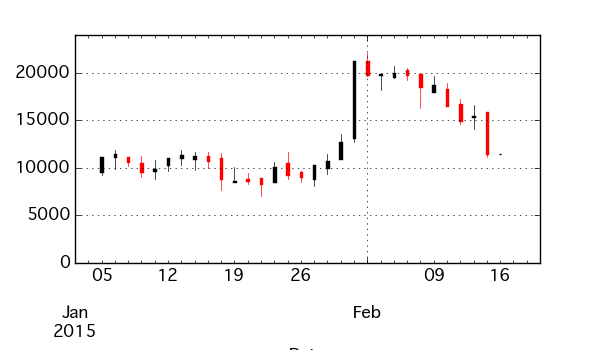
また、ohlc でリサンプリングされた結果 ( DataFrame ) からもプロットできる。
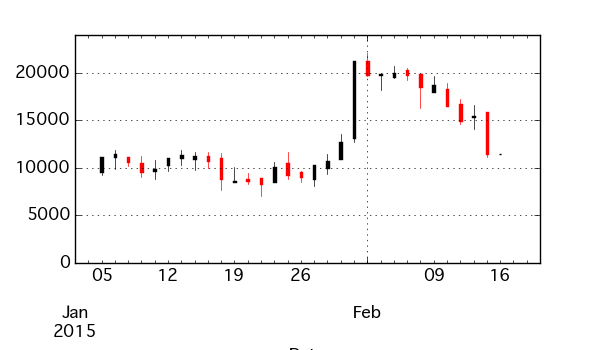
df = s.resample('B', how='ohlc') df.head() # open high low close # 2015-01-05 9537 11168 9252 11168 # 2015-01-06 11176 11872 9912 11458 # 2015-01-07 11099 11099 10174 10652 # 2015-01-08 10465 11236 8980 9539 # 2015-01-09 9655 10805 8797 9910 df.plot(kind='ohlc')
以下、利用したコード。

Python for Finance: Analyze Big Financial Data
- 作者: Yves Hilpisch
- 出版社/メーカー: Oreilly & Associates Inc
- 発売日: 2014/12/27
- メディア: ペーパーバック
- この商品を含むブログを見る
Python pandas 図でみる データ連結 / 結合処理
なんかぼやぼやしているうちにひさびさの pandas エントリになってしまった。基本的な使い方については網羅したい気持ちはあるので、、、。
今回は データの連結 / 結合まわり。この部分 公式ドキュメント がちょっとわかりにくいので改訂したいなと思っていて、自分の整理もかねて書きたい。
公式の方はもう少し細かい使い方も載っているのだが、特に重要だろうというところだけをまとめる。
連結 / 結合という用語は以下の意味で使っている。まず憶えておいたほうがよい関数、メソッドは以下の 4 つだけ。
- 連結: データの中身をある方向にそのままつなげる。
pd.concat,DataFrame.append - 結合: データの中身を何かのキーの値で紐付けてつなげる。
pd.merge,DataFrame.join
連結 (concatenate)
柔軟な連結 pd.concat
ふたつの DataFrame の連結は pd.concat で行う ( DataFrame.concat ではない)。元データとする DataFrame を df1、df2 としてそれぞれ以下のように定義する。
import pandas as pd df1 = pd.DataFrame({'A': ['A0', 'A1', 'A2', 'A3'], 'B': ['B0', 'B1', 'B2', 'B3'], 'C': ['C0', 'C1', 'C2', 'C3'], 'D': ['D0', 'D1', 'D2', 'D3']}, index=[0, 1, 2, 3]) df2 = pd.DataFrame({'A': ['A4', 'A5', 'A6', 'A7'], 'B': ['B4', 'B5', 'B6', 'B7'], 'C': ['C4', 'C5', 'C6', 'C7'], 'D': ['D4', 'D5', 'D6', 'D7']}, index=[4, 5, 6, 7])
基本的な連結
この 2 つのデータに対する pd.concat の結果は下図 Result で示すもの = 縦方向の連結になる。pd.concat の引数は連結したい DataFrame のリスト [df1, df2] になる。Result の上半分が df1, 下半分が df2 に対応している。
pd.concat([df1, df2])

引数には 3つ以上の DataFrame からなるリストを渡してもよい。結果は 各 DataFrame が順に縦方向に連結されたものになる。
df3 = pd.DataFrame({'A': ['A8', 'A9', 'A10', 'A11'],
'B': ['B8', 'B9', 'B10', 'B11'],
'C': ['C8', 'C9', 'C10', 'C11'],
'D': ['D8', 'D9', 'D10', 'D11']},
index=[8, 9, 10, 11])
pd.concat([df1, df2, df3])

列名が異なる場合の連結
上ふたつの例では連結する各 DataFrame の 列名 ( columns )はすべて ['A', 'B', 'C', 'D'] で同一だったので気にする必要はなかったが、内部的には 各列は 列名で紐付けされてから連結されている。
以下のように 列名が異なる場合、元データ両方に存在する 列 ['B', 'D'] はそれそれ紐付けられて縦連結され、片方にしか存在しない列は 空の部分が NaN でパディングされる。
df4 = pd.DataFrame({'B': ['B2', 'B3', 'B6', 'B7'],
'D': ['D2', 'D3', 'D6', 'D7'],
'F': ['F2', 'F3', 'F6', 'F7']},
index=[2, 3, 6, 7])
pd.concat([df1, df4])

補足 連結方向のラベル ( 縦方向の場合 行名 = index ) 同士は紐付けされず 常にそのまま連結される。出力をみると、 df1 の index である [0, 1, 2, 3] と df2 の index である [2, 3, 6, 7] は一部重複しているが、紐付けされず元の順序のまま連結されている。連結方向のラベルを重複させたくない場合の処理は後述。
横方向の連結
横方向に連結したい場合は axis=1 を指定。このとき 直前の例とは逆に 紐付けは 連結方向でないラベル = index について行われる。 連結方向のラベルにあたる columns はそのまま維持される。
pd.concat([df1, df4], axis=1)

連結処理の指定
上でみたとおり、pd.concat による連結では、データを "連結方向でないラベル" で紐付けしてから 連結していた。この紐付けは元データをすべて残す形 = 完全外部結合のような形で行われている。
引数の各データに共通のラベルのみを残して連結したい場合は join='inner' を指定する。下の例では横連結を指定しているため、共通の index である [2, 3] に対応する行のみが残る。
pd.concat([df1, df4], axis=1, join='inner')

また、紐付け時に特定のラベルのみを残したい場合もある。そのときは join_axes で残したいラベルの名前を指定すればよい。
例えば axis=1 横方向に連結するとき、join_axes に ひとつめの DataFrame の index を指定すると、それらだけが残るため 左外部結合のような処理になり、
pd.concat([df1, df4], axis=1, join_axes=[df1.index])

ふたつめの DataFrame の index を指定すると 右外部結合のような処理になる。
pd.concat([df1, df4], axis=1, join_axes=[df4.index])

連結方向のラベルの指定
pd.concat は既定では 連結方向のラベル ( 縦連結の場合は index、横連結の場合は columns ) に対しては特に変更を行わない。そのため、上の例のように 連結結果に同名のラベルが重複してしまうことがある。
こういう場合、連結元のデータごとに keys キーワードで指定したラベルを追加で付与することができる。このとき、結果は 複数のレベルをもつ MultiIndex になる ( MultiIndex については別途)。これは図だけでもわかりにくいので テキストでの出力も添付。
pd.concat([df1, df4], axis=1, keys=['X', 'Y']) # X Y # A B C D B D F # 0 A0 B0 C0 D0 NaN NaN NaN # 1 A1 B1 C1 D1 NaN NaN NaN # 2 A2 B2 C2 D2 B2 D2 F2 # 3 A3 B3 C3 D3 B3 D3 F3 # 6 NaN NaN NaN NaN B6 D6 F6 # 7 NaN NaN NaN NaN B7 D7 F7

もしくは、ignore_index=True を指定して 連結方向のラベルを 0 から振りなおすことができる。これは縦連結のときに index を連番で振りなおす場合に便利。
pd.concat([df1, df4], ignore_index=True)

縦方向のシンプルな連結 DataFrame.append
pd.concat でたいていの連結はできる。うち、よく使う 縦方向の連結については DataFrame.append でよりシンプルに書ける。
df1.append(df2)

引数には DataFrame のリストも渡せる。
df1.append([df2, df4])

また、データに一行追加したい、なんて場合も DataFrame.append。このとき、引数は 追加する行に対応した Series になる。
このとき、連結対象の DataFrame の columns と Series の index どうしが紐付けられて連結される。そのため、行として追加する Series は以下のような形で作る。Series が name 属性を持っている場合は 連結後の行の index は name で指定されたもの (ここでは 10 ) になる。
s1 = pd.Series(['X0', 'X1', 'X2', 'X3'], index=['A', 'B', 'C', 'D'], name=10) df1.append(s1)

name 属性のない Series を連結したい場合は ignore_index=True を指定しないとエラーになる。
s2 = pd.Series(['X0', 'X1', 'X2', 'X3'], index=['A', 'B', 'C', 'D']) # NG! df1.append(s2) # TypeError: Can only append a Series if ignore_index=True or if the Series has a name # OK df1.append(s2, ignore_index=True)

補足 append の際、内部では毎回 元データも含めた全体のコピー処理が走るので、ループで一行ずつ追加するような処理は避けたほうがよい。
結合 (merge)
列の値による結合 pd.merge
ふたつの DataFrame の結合は pd.merge もしくは DataFrame.merge で行う。結合もとの DataFrame を left、right としてそれぞれ以下のように定義する。
left = pd.DataFrame({'key': ['K0', 'K1', 'K2', 'K3'],
'A': ['A0', 'A1', 'A2', 'A3'],
'B': ['B0', 'B1', 'B2', 'B3']})
right = pd.DataFrame({'key': ['K1', 'K3', 'K5', 'K7'],
'C': ['C1', 'C3', 'C5', 'C7'],
'D': ['D1', 'D3', 'D5', 'D7']},
index=[1, 3, 5, 7])
この例では 作成した DataFrame 中の key カラムを結合時のキーとし、この列の値が同じ行どうしを結合したい。結合時のキーとなる列名は on キーワードで指定する。既定では内部結合となり、両方のデータに共通の ['K1', 'K3'] に対応する行どうしが結合されて残る。
pd.merge(left, right, on='key')

結合方法は how キーワードで指定する。指定できるのは、
inner: 既定。内部結合。両方のデータに含まれるキーだけを残す。left: 左外部結合。ひとつめのデータのキーをすべて残す。right: 右外部結合。ふたつめのデータのキーをすべて残す。outer: 完全外部結合。すべてのキーを残す。
それぞれの出力を順に図示する。
pd.merge(left, right, on='key', how='left')

pd.merge(left, right, on='key', how='right')

pd.merge(left, right, on='key', how='outer')

複数のキーによる結合
キーとして複数の列を指定したい場合は、on キーワードにキーとする列名のリストを渡せばよい。
left = pd.DataFrame({'key1': ['K0', 'K0', 'K1', 'K2'],
'key2': ['K0', 'K1', 'K0', 'K1'],
'A': ['A0', 'A1', 'A2', 'A3'],
'B': ['B0', 'B1', 'B2', 'B3']})
right = pd.DataFrame({'key1': ['K0', 'K1', 'K1', 'K2'],
'key2': ['K0', 'K0', 'K0', 'K0'],
'C': ['C0', 'C1', 'C2', 'C3'],
'D': ['D0', 'D1', 'D2', 'D3']})
pd.merge(left, right, on=['key1', 'key2'])

index による結合 DataFrame.join
各データの index をキーとして結合したい場合は、DataFrame.join が便利。既定は左外部結合となり、結合方法は how で変更できる。指定できるオプションは pd.merge と同じ。
left = pd.DataFrame({'A': ['A0', 'A1', 'A2'],
'B': ['B0', 'B1', 'B2']},
index=['K0', 'K1', 'K2'])
right = pd.DataFrame({'C': ['C0', 'C2', 'C3'],
'D': ['D0', 'D2', 'D3']},
index=['K0', 'K2', 'K3'])
left.join(right)

left.join(right, how='inner')

left.join(right, how='right')

left.join(right, how='outer')

補足 pd.merge でも left_index ならびに right_index キーワードによって index をキーとした結合はできる。
まとめ
pandas でのデータ連結 / 結合まわりを整理した。これ以外の データ変形 (行持ち / 列持ち変換とか) は R の {dplyr}、{tidyr} との対比でまとめたやつがあるのだが、列名 や行名が複数のレベルを持つ = MultiIndex の場合など pandas 固有のものもあるのでまた別途。
- Python pandas でのグルーピング/集約/変換処理まとめ - StatsFragments
- R dplyr, tidyr でのグルーピング/集約/変換処理まとめ - StatsFragments
2015/05/12追記
公式ドキュメントに反映した。こちらのほうが網羅的。
Merge, join, and concatenate — pandas 0.16.2 documentation

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (10件) を見る
Python pandas strアクセサによる文字列処理
概要
今週の 週刊 pandas は文字列処理について。やたらと文字数が多くなったのだが、これはデータを都度表示しているせいであって自分の話がムダに長いわけではない、、、と思いたい。
今回はこちらの記事に書いた内容も使うので、適宜ご参照ください。
サンプルデータ
なんか適当な実データないかな?と探していたら 週間少年ジャンプの過去作品の連載作品 / ジャンルなどがまとめられているサイトをみつけた。これを pandas で集計できる形まで整形することをゴールにしたい。
KTR's Comic Room: Weekly Jump Database
データの読み込み
上記リンクの "ジャンプ連載データ表" を、ファイル名 "jump_db.html" としてローカルに保存した。
補足 pd.read_html では引数に URL を渡して 直接ネットワークからファイルを読むこともできる。が、今回は データ元サイトへの負荷をさけるため、ローカルの HTML ファイルを読むことにした。
依存パッケージのインストール
pd.read_html を利用するためには 以下のパッケージが必要。インストールしていない場合はインストールする。
pip install lxml html5lib beautifulsoup4
準備
numpy, pandas をロードする。
# おまじない from __future__ import unicode_literals import numpy as np import pandas as pd # 表示する行数を指定 pd.options.display.max_rows = 10
HTML ファイルの読み込み
pd.read_html では 対象の HTML ファイルに含まれる TABLE 要素 を DataFrame として読み出す。ひとつの html ファイルには複数の TABLE が含まれることもあるため、pd.read_html の返り値は DataFrame のリストになっている。
# read_htmlの返り値は DataFrame のリストになる dfs = pd.read_html('jump_db.html', header=0) type(dfs) # list # 最初のDataFrameを取り出す df = dfs[0] df # 開始 終了 回数 タイトル 作者 原作者・その他 ジャンル # 0 6801 7144 131 父の魂 貝塚ひろし NaN NaN # 1 6801 6811 11 くじら大吾 梅本さちお NaN NaN # 2 6804 6913 21 おれはカミカゼ 荘司としお NaN NaN # 3 6808 7030 60 漫画コント55号 榎本有也 NaN NaN # 4 6810 6919 21 男の条件 川崎のぼる 原作/梶原一騎 NaN # .. ... ... ... ... ... ... ... # 667 1442 連載中 12+ ハイファイクラスタ 後藤逸平 NaN 近未来,SF,刑事,一芸 # 668 1443 連載中 11+ Sporting Salt 久保田ゆうと NaN スポーツ医学,学園 # 669 1451 連載中 3+ 卓上のアゲハ 古屋樹 NaN 卓球,エロ,ミステリ # 670 1452 連載中 2+ E−ROBOT 山本亮平 NaN エロ,ロボット # 671 1501 連載中 1+ 学糾法廷 小畑健 原作/榎伸晃 学園,裁判 # # [672 rows x 7 columns] # 後続処理でのエラーチェックのため、最初のレコード数を保存しておく original_length = len(df)
最近の新連載はエロばっかりなの、、、??ちょっと心惹かれるがまあそれはいい。
各カラムのデータ型を確認するには df.dtypes 。文字列が含まれるカラムは object 型になっている。
# カラムのデータ型を表示 df.dtypes # 開始 float64 # 終了 object # 回数 object # タイトル object # 作者 object # 原作者・その他 object # ジャンル object # dtype: object
str アクセサ
以降の処理は pandas の str アクセサを中心に行う。pandas では内部のデータ型が文字列 ( str もしくは unicode ) 型のとき、 str アクセサを使って データの各要素に対して文字列メソッドを適用することができる。str アクセサから使えるメソッドの一覧はこちら。
補足 v0.15.1 時点では str アクセサからは使えない Python 標準の文字列メソッドもある。そんなときは apply。apply についてはこちら。
例えば、文字列を小文字化する lower メソッドを各要素に対して適用する場合はこんな感じ。
# 各要素が大文字の Series を作成 d.Series(['AAA', 'BBB', 'CCC']) # 0 AAA # 1 BBB # 2 CCC # dtype: object # str アクセサを通じて、各要素に lower メソッドを適用 pd.Series(['AAA', 'BBB', 'CCC']).str.lower() # 0 aaa # 1 bbb # 2 ccc # dtype: object # str アクセサを使わないと AttributeError になり NG! pd.Series(['AAA', 'BBB', 'CCCC']).lower() # AttributeError: 'Series' object has no attribute 'lower'
また、str アクセサに対するスライシングは各要素へのスライシングになる。各要素の最初の二文字を取り出す場合、
pd.Series(['AAA', 'BBB', 'CCC']).str[0:2] # 0 AA # 1 BB # 2 CC # dtype: object
データの処理
ここからサンプルデータへの処理を始める。
連載継続中かどうかのフラグ立て
元データでは、各作品が現在も連載中かどうかは以下いずれかでわかる。
- "終了" カラムの値が "連載中" である
- "回数" カラムの末尾に
+がついている
# 回数 カラムの値を確認 df['回数'] # 0 131 # 1 11 # 2 21 # ... # 669 3+ # 670 2+ # 671 1+ # Name: 回数, Length: 672, dtype: object
が、このままでは機械的に扱いにくいため、連載中かどうかで bool のフラグをたてたい。そのためには、以下どちらかの処理を行えばよい。
- "終了" カラムに対する論理演算を行う
- "回数" カラムの末尾を
str.endswithで調べる
# 終了カラムの値で判別する場合 df['終了'] == '連載中' # 0 False # 1 False # 2 False # ... # 669 True # 670 True # 671 True # Name: 終了, Length: 672, dtype: bool # 回数カラムの値で判別する場合 df['回数'].str.endswith('+') # 0 False # 1 False # 2 False # ... # 669 True # 670 True # 671 True # Name: 回数, Length: 672, dtype: bool # 結果を 連載中 カラムとして代入 df['連載中'] = df['回数'].str.endswith('+')
連載回数のパース
続けて、現在は文字列 ( object ) 型として読み込まれている連載回数を数値型として扱いたい。が、上のとおり 連載中のレコードには末尾に +, 確実な連載回数が取得できているレコードは [ ] で囲われているため、そのままでは数値型へ変換できない。
# そのまま変換しようとすると NG! df['回数'].astype(float) # ValueError: invalid literal for float(): 1+
数値に変換するためには、文字列中の数値部分だけを切り出してから数値に変換すればよい。str.extract を使うとデータに対して指定した正規表現とマッチする部分を抽出できる。抽出した数値部分に対して astype を使って float 型へ変換する。
# 数値部分だけを抽出 (この時点では各要素は文字列) ['回数'].str.extract('([0-9]+)') # 0 131 # 1 11 # 2 21 # ... # 669 3 # 670 2 # 671 1 # Name: 回数, Length: 672, dtype: object # float 型へ変換 (表示の dtype 部分で型がわかる) df['回数'].str.extract('([0-9]+)').astype(float) # 0 131 # 1 11 # 2 21 # ... # 669 3 # 670 2 # 671 1 # Name: 回数, Length: 672, dtype: float64
補足 1件 元データが "??" のものがある。この文字列は数値を含まないため、str.extract の結果は NaN になる。NaN を含むデータは int 型へは変換できない。
# NG! NaN があるため int 型へは変換できない df['回数'].str.extract('([0-9]+)').astype(int) # ValueError: cannot convert float NaN to integer
上記の結果を元のカラムに代入して上書きする。その後、dropna で NaN のデータを捨てる。dropna についてはこちら。
# 結果を上書き df['回数'] = df['回数'].str.extract('([0-9]+)').astype(float) # 回数 が NaN のデータを捨てる df = df.dropna(axis=0, subset=['回数']) # 1レコードがフィルタされたことを確かめる assert len(df) + 1 == original_length df.shape # (671, 8)
連載開始時のタイムスタンプ取得
"開始" カラムの値から 年度, 週を取得。元データは、通常は "9650" のように年下二桁 + 週 の4桁、合併号では "9705.06" のようなドット区切りの値が入っている。そのため、read_heml では float 型としてパースされている。これを datetime 型に変換したい。
ここでは N 号は その年の N 週 月曜に発行されたものとして日付を埋める。実際には号数と週番号はずれる / 合併号は発行曜日がずれる / 昔は発行曜日が違っていた、と様々な問題があるがここでは無視する。
素直にやるなら apply を使ってこんな感じで書ける。pd.offsets についてはこちら。
import datetime def parse_date(x): # 小数点を切り捨て x = np.floor(x) y, w = divmod(x, 100) # y が50より大きければ 1900年代, それ以外なら2000年代として扱う y += 1900 if y > 50 else 2000 d = datetime.datetime(int(y), 1, 1) # 週次のオフセットを追加 d += pd.offsets.Week(int(w), weekday=0) return d df['開始'].apply(parse_date) # 0 1968-01-08 # 1 1968-01-08 # 2 1968-01-29 # ... # 669 2014-12-22 # 670 2014-12-29 # 671 2015-01-05 # Name: 開始, Length: 671, dtype: datetime64[ns]
今回は文字列処理の記事なのであえて文字列として処理する。最初の手順は以下。
- 処理対象のカラムを
astypeで文字列型に変換し、適当な変数に代入する。 str.splitで小数点前後で文字列を分割。返り値をDataFrameで受け取るためreturn_type='frame'を指定。- 小数点の前の文字列のみ = 1列目のみ を処理対象の変数に代入しなおす。
# 処理対象のカラムを文字列型に変換 dt = df['開始'].astype(str) dt # 0 6801.0 # 1 6801.0 # 2 6804.0 # ... # 669 1451.0 # 670 1452.0 # 671 1501.0 # Name: 開始, Length: 671, dtype: object # str.split で小数点前後を分割 dt.str.split('.', return_type='frame') # 0 1 # 0 6801 0 # 1 6801 0 # 2 6804 0 # 3 6808 0 # 4 6810 0 # .. ... .. # 667 1442 0 # 668 1443 0 # 669 1451 0 # 670 1452 0 # 671 1501 0 # # [671 rows x 2 columns] # 小数点の前の文字列のみを処理対象の変数に代入 dt = dt.str.split('.', return_type='frame')[0] dt # 0 6801 # 1 6801 # 2 6804 # ... # 669 1451 # 670 1452 # 671 1501 # Name: 0, Length: 671, dtype: object
ここで、values プロパティを使って内部の値を確認する。00年代の値は数値の桁数が異なるため、文字列長も異なっていることがわかる。このままではパースしにくいため、以下の処理を行う。
str.padで指定した長さまで空白文字でパディングする。str.replaceで空白文字を "0" に置き換える。
# 変数内部の値を確認 dt.values # array([u'6801', u'6801', u'6804', u'6808', u'6810', u'6811', u'6811', # ..... # u'9933', u'9934', u'9943', u'9943', u'9944', u'1', u'2', u'12', # u'13', u'23', u'24', u'32', u'33', u'34', u'38', u'47', u'48', # ..... # u'1451', u'1452', u'1501'], dtype=object) # str.pad で指定した長さまでパディング dt.str.pad(4).values # array([u'6801', u'6801', u'6804', u'6808', u'6810', u'6811', u'6811', # ..... # u'9933', u'9934', u'9943', u'9943', u'9944', u' 1', u' 2', # u' 12', u' 13', u' 23', u' 24', u' 32', u' 33', u' 34', # ..... # u'1441', u'1442', u'1443', u'1451', u'1452', u'1501'], dtype=object) # str.replace で空白文字を "0" に置換 dt.str.pad(4).str.replace(' ', '0').values # array([u'6801', u'6801', u'6804', u'6808', u'6810', u'6811', u'6811', # ..... # u'0012', u'0013', u'0023', u'0024', u'0032', u'0033', u'0034', # ..... # u'1441', u'1442', u'1443', u'1451', u'1452', u'1501'], dtype=object) # ここまでの処理結果を代入 dt = dt.str.pad(4).str.replace(' ', '0')
ここまでで、変数 dt の中身は "年2桁 + 週2桁" の4文字からなる文字列になった。こいつに対して、
- スライシングによって文字列を 年, 週の部分文字列に分割する。
- 年については下2桁しかないため、
whereで場合分けして 西暦4桁の文字列にする。whereについてはこちら。これでpd.to_datetimeで処理できる形になる。 pd.to_datetimeで日付型にパース。入力は年のみのため、format='%Y'を指定する。pd.to_datetimeについてはこちら。
# スライシングで年, 週に分割 year = dt.str[0:2] week = dt.str[2:4] # 西暦4桁の文字列を作成 year = ('19' + year).where(year > '50', '20' + year) year # 0 1968 # 1 1968 # 2 1968 # ... # 669 2014 # 670 2014 # 671 2015 # Name: 0, Length: 671, dtype: object # pd.to_datetime で日付型にパース year = pd.to_datetime(year, format='%Y') year # 0 1968-01-01 # 1 1968-01-01 # 2 1968-01-01 # ... # 669 2014-01-01 # 670 2014-01-01 # 671 2015-01-01 # Name: 0, Length: 671, dtype: datetime64[ns]
ここまでできれば、あと必要な処理は、
- 週の文字列から、
pd.offsets.Weekインスタンスを作る - 年から生成したタイムスタンプを
Week分ずらす
# 週の文字列から、pd.offsets.Week インスタンスを作る week = [pd.offsets.Week(w, weekday=0) for w in week.astype(int)] week # [<Week: weekday=0>, # <Week: weekday=0>, # <4 * Weeks: weekday=0>, # ..... # <52 * Weeks: weekday=0>, # <Week: weekday=0>] # 10. 年から生成したタイムスタンプを 週番号分ずらす dt = [y + w for y, w in zip(year, week)] dt # [Timestamp('1968-01-08 00:00:00'), # Timestamp('1968-01-08 00:00:00'), # Timestamp('1968-01-29 00:00:00'), # ..... # Timestamp('2014-12-29 00:00:00'), # Timestamp('2015-01-05 00:00:00')] # 元の DataFrame に対して代入 df.loc[:, '開始'] = dt
原作有無でのフラグ立て
元データの "原作者・その他" カラムをみると、ここには原作ほか関係者 (監修とか) の名前も入っている。
df[~pd.isnull(df['原作者・その他'])]['原作者・その他'] # 4 原作/梶原一騎 # 9 原作/スドウテルオ # 28 原作/藤井冬木 # ... # 660 原作/成田良悟 # 661 原作/下山健人 # 671 原作/榎伸晃 # Name: 原作者・その他, Length: 111, dtype: object
"原作者・その他" カラムの値から 原作者がいる場合だけフラグを立てたい。具体的には、カラムに "原作/"を含む値が入っているときは 原作ありとして扱いたい。
そんな場合は str.contains。
補足 元データでは 原作者は常に先頭に来ているので、str.startswith でもよい。
df['原作者・その他'].str.contains('原作/') # 0 NaN # ... # 671 True # Name: 原作者・その他, Length: 672, dtype: object # 結果をカラムに代入 df['原作あり'] = df['原作者・その他'].str.contains('原作/')
重複データの削除
一部のシリーズでは 全体と 各部(第一部、二部など) で重複してデータが取得されている。目視でそれっぽいシリーズを抽出して表示してみる。
# 重複しているシリーズ候補 series = ['コブラ', 'ジョジョの奇妙な冒険', 'BASTARD!!', 'みどりのマキバオー', 'ONE PIECE', 'ボボボーボ・ボーボボ', 'DEATH NOTE', 'スティール・ボール・ラン', 'トリコ', 'NARUTO'] for s in series: # 重複しているシリーズ候補を タイトルに含むデータを取得 dup = df[df['タイトル'].str.contains(s)] print(dup[['開始', '回数', 'タイトル']]) # 開始 回数 タイトル # 299 8701.02 593 ジョジョの奇妙な冒険 # 300 8701.02 44 ジョジョの奇妙な冒険(第1部) # 314 8747.00 69 ジョジョの奇妙な冒険(第2部) # 335 8916.00 152 ジョジョの奇妙な冒険(第3部) # 378 9220.00 174 ジョジョの奇妙な冒険(第4部) # 433 9552.00 154 ジョジョの奇妙な冒険(第5部) # 489 1.00 [158] ジョジョの奇妙な冒険第6部・ストーンオーシャン # .....後略
上の例で言うと、"ジョジョの奇妙な冒険" シリーズの回数として 1部 〜 5部の回数分が入っているようだ。同様にシリーズ全体の連載回数に含まれていると思われるレコードを目視で抽出、削除。
# 削除対象タイトル dups = ['ジョジョの奇妙な冒険(第1部)', 'ジョジョの奇妙な冒険(第2部)', 'ジョジョの奇妙な冒険(第3部)', 'ジョジョの奇妙な冒険(第4部)', 'ジョジョの奇妙な冒険(第5部)', 'BASTARD!!〜暗黒の破壊神〜(闇の反逆軍団編)', 'BASTARD!!〜暗黒の破壊神〜(地獄の鎮魂歌編)', 'みどりのマキバオー(第1部)', 'みどりのマキバオー(第2部)', 'ONE PIECE《サバイバルの海 超新星編》', 'ONE PIECE《最後の海 新世界編》', 'ボボボーボ・ボーボボ(第1部)', '真説ボボボーボ・ボーボボ', 'DEATH NOTE(第1部)', 'DEATH NOTE(第2部)', 'スティール・ボール・ラン 1st Stage', 'スティール・ボール・ラン 2nd Stage', 'トリコ〜人間界編〜', 'トリコ〜グルメ界編〜', 'NARUTO(第一部)', 'NARUTO(第二部)'] # タイトルの値が dups に含まれるレコードを除外 df = df[~df['タイトル'].isin(dups)] # dups 分のレコードがフィルタされたことを確かめる assert len(df) + len(dups) + 1 == original_length df.shape # (652, 9)
ジャンルデータの作成
"ジャンル" カラムには複数のジャンルが カンマ区切りで含まれている。
df['ジャンル'] # 0 NaN # 1 NaN # 2 NaN # ... # 669 卓球,エロ,ミステリ # 670 エロ,ロボット # 671 学園,裁判 # Name: ジャンル, Length: 652, dtype: object
これを集計しやすい形にするため、各ジャンルに該当するかどうかを bool でフラグ立てしたい。
このカラムには "秘密警察" とか "ゴム人間" なんて値も入っているため、ユニークな件数をカウントして上位ジャンルのみフィルタする。
まずは、カンマ区切りになっている値を分割して、ジャンル個々の値からなる Series を作る。
# NaN をフィルタ genres = df['ジャンル'].dropna() # カンマ区切りの値を split して、一つのリストとして結合 genres = reduce(lambda x, y: x + y, genres.str.split(',')) # genres = pd.Series(genres) genres # 0 硬派 # 1 不良 # 2 アニメ化 # ... # 1358 ロボット # 1359 学園 # 1360 裁判 # Length: 1361, dtype: object
Series 内の要素がそれぞれいくつ含まれるかをカウントするには value_counts()。
genres.value_counts() # ギャグ 76 # 学園 75 # アニメ化 74 # ... # 演歌 1 # 学園ショートギャグ 1 # スポーツ? 1 # Length: 426, dtype: int64 # 上位10件を表示 genres.value_counts()[:10] # ギャグ 76 # 学園 75 # アニメ化 74 # 格闘 36 # 一芸 32 # ファンタジー 32 # ゲーム化 30 # 変身 28 # ラブコメ 28 # 不良 23 # dtype: int64
とりあえず 以下の3ジャンルをみることにする。各レコードについてジャンルは複数あてはまりうる。そのため、それぞれのフラグについて dummy のカラムを作成し、ジャンルに該当する場合に True を入れる。
genres = ['ギャグ', '格闘', 'ラブコメ'] # str アクセサを利用するため NaN 値をパディング df['ジャンル'] = df['ジャンル'].fillna('') for genre in genres: df[genre] = df['ジャンル'].str.contains(genre)
できあがり
# 必要なカラムのみにフィルタ df = df[['開始', '回数', 'タイトル', '連載中', '原作あり', 'ギャグ', '格闘', 'ラブコメ']] df
できたデータはこんな感じ。俺たちの分析はここからだ!
| index | 開始 | 回数 | タイトル | 連載中 | 原作あり | ギャグ | 格闘 | ラブコメ |
|---|---|---|---|---|---|---|---|---|
| 0 | 1968-01-08 | 131 | 父の魂 | False | NaN | False | False | False |
| ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 667 | 2014-10-20 | 12 | ハイファイクラスタ | True | NaN | False | False | False |
| 668 | 2014-10-27 | 11 | Sporting Salt | True | NaN | False | False | False |
| 669 | 2014-12-22 | 3 | 卓上のアゲハ | True | NaN | False | False | False |
| 670 | 2014-12-29 | 2 | E−ROBOT | True | NaN | False | False | False |
| 671 | 2015-01-05 | 1 | 学糾法廷 | True | True | False | False | False |
まとめ
pandas で文字列処理する場合は str アクセサ。
補足 また、以下のサイトには毎週の掲載順位を含むデータがある。こちらのほうが興味深いが、前処理グレードがちょっと高かったのであきらめた。
おまけ: かんたんに集計
自分としては 想定の前処理ができた時点でもういいかって感じなのだが、簡単に集計してみる。
新連載数
各年 (年度ではなく calendar year) の新連載開始数を時系列でプロット。69年10月に週刊化されたらしく、直後の新連載数が特に多い。
import matplotlib.pyplot as plt summarised = df.groupby(df['開始'].dt.year)['タイトル'].count() ax = summarised.plot(figsize=(7, 3)) ax.set_title('新連載数')

連載回数
連載回数の頻度分布をみてみると、80年より前には10週以下で短期連載 or 連載終了した作品が比較的 多そう。80年代以降の分布が現在の感覚に近いと思う。
# 連載終了したデータのみにフィルタ fin = df[~df['連載中']] fin1 = fin[fin['開始'].dt.year < 1980] fin2 = fin[fin['開始'].dt.year >= 1980] fig, axes = plt.subplots(2, figsize=(7, 4)) bins = np.arange(0, 1000, 5) fin1['回数'].plot(kind='hist', ax=axes[0], bins=bins, xlim=(0, 100), label='80年より前に連載開始', legend=True) fin2['回数'].plot(kind='hist', ax=axes[1], bins=bins, xlim=(0, 100), label='80年以降に連載開始', legend=True, color='green') fig.suptitle('連載回数の頻度分布')

連載の継続率
連載の継続率 = 生存率とみて lifelines を使って Kaplan-Meier 曲線を引く。横軸が時間経過 (週)、縦軸がその時点まで連載継続している確率になる。
lifelines についてはこちら。
from lifelines import KaplanMeierFitter def to_pct(y, position): s = str(100 * y) return s + '%' from matplotlib.ticker import FuncFormatter pctformatter = FuncFormatter(to_pct) df2 = df[df['開始'].dt.year >= 1980] kmf = KaplanMeierFitter() kmf.fit(df2['回数'], event_observed=~df2['連載中']) ax = kmf.plot(figsize=(7, 3), xlim=(0, 200)) ax.yaxis.set_major_formatter(pctformatter)
50週 = 1年 連載継続できるのは 30 % くらいか、、、厳しい世界だホント。

ジャンル別の連載の継続率
上と同じ、でジャンル別。
ax = None for genre in genres: group = df2[df2[genre]] kmf = KaplanMeierFitter() kmf.fit(group['回数'], event_observed=~group['連載中'], label='ジャンル=' + genre) # 描画する Axes を指定。None を渡すとエラーになるので場合分け if ax is None: ax = kmf.plot(figsize=(7, 3), xlim=(0, 200), ci_show=False) else: ax = kmf.plot(ax=ax, figsize=(7, 3), xlim=(0, 200), ci_show=False) ax.yaxis.set_major_formatter(pctformatter)
もう少し偏るかと思ったがそうでもなかった。ラブコメは 150 回くらいに壁がありそう。ニセコイがこの壁を乗り越えてくれることを切に願う。


Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る