R で 状態空間モデル: {dlm} で単変量モデルの状態空間表現を利用する
こちらの続きで、状態空間時系列分析入門の第8章の内容。

- 作者: J.J.F.コマンダー,S.J.クープマン,Jacques J.F. Commandeur,Sime Jan Koopman,和合肇
- 出版社/メーカー: シーエーピー出版
- 発売日: 2008/09
- メディア: 単行本
- 購入: 2人 クリック: 4回
- この商品を含むブログを見る
状態空間モデルでは、トレンド、季節効果、説明変数効果などさまざまな要因を考慮することができる。また これらの要因を単一の状態空間表現で統一的に扱える。時刻 における観測値
と状態
は以下の式で書ける (前回と同じ)。
この式であらわされたモデルを {dlm} で利用する際にどうすればよいのかをまとめたい。
vignette にあるとおり、{dlm} ではモデルを dlm::dlmModPoly などのモデル記述関数の組み合わせで定義できる。これらの関数は便利だが、上の式との関係は外側からはよくわからない。
上式から {dlm} のモデルを作成する際には dlm::dlm を直接使うのがよさそうだ。dlm::dlm の引数は上式にほぼ対応しているため、式表現されたモデルをそのまま実装できる。また、モデル記述関数も内部的には dlm::dlm を使っているため、これら関数の理解にも役立つ。dlm::dlm の引数と上式の対応は前回書いたとおり。
FF:に対応。
V:に対応。
GG:に対応。
W:に対応。
m0:の事前分布の平均。
C0:の事前分布の分散。
J...: 各パラメータが時間変化するとき利用。
参考 サイト 「状態空間時系列分析入門」をRで再現する はこのテキストを {dlm}, {KFAS} で再現している。こちらは dlm::dlm ではなく dlm::dlmModPoly などの関数を使ってモデル作成している。
準備
まずデータを読み込む。データは著者のサポートサイト、An Introduction to State Space Time Series Analysis から入手できる。
library(dlm) # install_github('sinhrks/ggfortify') library(ggfortify) ukdrivers <- read.table('UKdriversKSI.txt', skip = 1) ukdrivers <- ts(ukdrivers[[1]], start = c(1969, 1), frequency = 12) ukdrivers <- log(ukdrivers)
以降、テキストに記載のモデル順に dlm::dlm から利用する場合のプログラムを列挙する。値 / 推定するパラメータを変更したい場合は buildObj 内の対応する要素を適当に修正すればよい。
1. ローカルレベルモデル (確定的レベル)
それぞれのモデルについて 状態 として何が考慮されているかを書いておく。
:
は確定的レベル (経時変化しない)。
補足 それぞれのモデルの式表現を書こうとしたのだが、はてなマークダウン記法だと TeX 記法中 の "&" がうまく処理できないようなので諦めた。詳細はテキストと見比べてください。
y <- ukdrivers parm<- log(var(y)) buildObj <- function(parm) { dlm(FF = matrix(1, 1, 1), V = matrix(exp(parm), 1, 1), GG = matrix(1, 1, 1), W = matrix(0, 1, 1), m0 = 0, C0 = matrix(1e+07, 1, 1)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] # [1,] 1 # # $V # [,1] # [1,] 0.02935256 # # $GG # [,1] # [1,] 1 # # $W # [,1] # [1,] 0 # # $m0 # [1] 0 # # $C0 # [,1] # [1,] 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

赤破線がカルマンフィルタ、青破線が平滑化した値。以降もプロットするが、確率的状態を使うと 実データにほぼフィットするため、グラフからでは差がよくわからない。
2. ローカル線形トレンドモデル (確率的レベルと確率的傾き)
:
は確率的レベル (経時変化する)、
は確率的傾き(経時変化する)。
parm <- log(c(var(y), 0.001, 0.001)) buildObj <- function(parm) { dlm(FF = matrix(c(1, 0), 1, 2), V = matrix(exp(parm[1]), 1, 1), GG = matrix(c(1, 1, 0, 1), 2, 2, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, exp(parm[3])), 2, 2, byrow = TRUE), m0 = c(0, 0), C0 = matrix(c(1e+07, 0, 0, 1e+07), 2, 2, byrow = TRUE)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] # [1,] 1 0 # # $V # [,1] # [1,] 0.002118549 # # $GG # [,1] [,2] # [1,] 1 1 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.01212741 0.00000e+00 # [2,] 0.00000000 1.92431e-10 # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

3. 季節要素のあるローカルレベルモデル (確率的レベルと確率的季節)
:
は確率的レベル、
は 四半期単位の確率的季節のダミー項。
ukinflation <- read.table('UKinflation.txt', skip = 1) ukinflation <- ts(ukinflation[[1]], start = c(1950, 1), frequency = 4) y <- ukinflation parm <- log(c(var(y), 0.00001, 0.00001)) buildObj <- function(parm) { dlm(FF = matrix(c(1, 1, 0, 0), 1, 4), V = matrix(c(exp(parm[1])), 1, 1), GG = matrix(c(1, 0, 0, 0, 0, -1, -1, -1, 0, 1, 0, 0, 0, 0, 1, 0), 4, 4, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0, 0, exp(parm[3]), 0, 0, 0, 0, 0, 0, 0, 0, 0, 0), 4, 4, byrow = TRUE), m0 = c(0, 0, 0, 0), C0 = matrix(c(1e+07, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 1e+07), 4, 4, byrow = TRUE)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] [,3] [,4] # [1,] 1 1 0 0 # # $V # [,1] # [1,] 3.37127e-05 # # $GG # [,1] [,2] [,3] [,4] # [1,] 1 0 0 0 # [2,] 0 -1 -1 -1 # [3,] 0 1 0 0 # [4,] 0 0 1 0 # # $W # [,1] [,2] [,3] [,4] # [1,] 2.124158e-05 0.000000e+00 0 0 # [2,] 0.000000e+00 4.345176e-07 0 0 # [3,] 0.000000e+00 0.000000e+00 0 0 # [4,] 0.000000e+00 0.000000e+00 0 0 # # $m0 # [1] 0 0 0 0 # # $C0 # [,1] [,2] [,3] [,4] # [1,] 1e+07 0e+00 0e+00 0e+00 # [2,] 0e+00 1e+07 0e+00 0e+00 # [3,] 0e+00 0e+00 1e+07 0e+00 # [4,] 0e+00 0e+00 0e+00 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

4. 説明変数のあるローカルレベルモデル (確率的レベル)
:
は確率的レベル、
は説明変数の係数。
説明変数は dlm::dlm へ引数 X として渡す。
ukpetrol <- read.table('logUKpetrolprice.txt', skip = 1) ukpetrol <- ts(ukpetrol, start = start(ukdrivers), frequency = frequency(ukdrivers)) y <- ukdrivers x <- ukpetrol parm <- c(var(y), 0.001) buildObj <- function(parm) { dlm(FF = matrix(c(1, 0), 1, 2), V = matrix(exp(parm[1]), 1, 1), GG = matrix(c(1, 0, 0, 1), 2, 2, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0), 2, 2, byrow = TRUE), m0 = c(0, 0), C0 = matrix(c(1e+07, 0, 0, 1e+07), 2, 2, byrow = TRUE), JFF = matrix(c(0, 1), 1, 2), X = as.matrix(x)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] # [1,] 1 1 # # $V # [,1] # [1,] 0.002347965 # # $GG # [,1] [,2] # [1,] 1 0 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.01166743 0 # [2,] 0.00000000 0 # # $JFF # [,1] [,2] # [1,] 0 1 # # $X # V1 # [1,] -2.273 # [2,] -2.279 # [3,] ... # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07 filtered <- dlmFilter(y, dlmModObj)$m smoothed <- dlmSmooth(y, dlmModObj)$s p <- autoplot(y) p <- autoplot(filtered[, 1] + filtered[, 2] * x, colour = 'red', linetype = 'dashed', p = p) autoplot(smoothed[, 1] + smoothed[, 2] * x, colour = 'blue', p = p)

5. 干渉変数のあるローカルレベルモデル (確率的レベル)
:
は確率的レベル、
は干渉変数の係数。
変数名は異なるが、式は説明変数のあるローカルレベルモデルと同一。
ukseats <- c(rep(0, (1982 - 1968) * 12 + 1), rep(1, (1984 - 1982) * 12 - 1)) ukseats <- ts(ukseats, start = start(ukdrivers), frequency = frequency(ukdrivers)) x <- ukseats parm <- c(var(y), 0.001) buildObj <- function(parm) { dlm(FF = matrix(c(1, 0), 1, 2), V = matrix(exp(parm[1]), 1, 1), GG = matrix(c(1, 0, 0, 1), 2, 2, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0), 2, 2, byrow = TRUE), m0 = c(0, 0), C0 = matrix(c(1e+07, 0, 0, 1e+07), 2, 2, byrow = TRUE), JFF = matrix(c(0, 1), 1, 2), X = as.matrix(x)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] # [1,] 1 0 # # $V # [,1] # [1,] 0.002692686 # # $GG # [,1] [,2] # [1,] 1 0 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.01041175 0 # [2,] 0.00000000 0 # # $JFF # [,1] [,2] # [1,] 0 1 # # $X # [,1] # [1,] 0 # [2,] 0 # [3,] ... # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07 filtered <- dlmFilter(y, dlmModObj)$m smoothed <- dlmSmooth(y, dlmModObj)$s p <- autoplot(y) p <- autoplot(filtered[, 1] + filtered[, 2] * x, colour = 'red', linetype = 'dashed', p = p) autoplot(smoothed[, 1] + smoothed[, 2] * x, colour = 'blue', p = p)

6. 季節要素と干渉変数のあるローカルレベルモデル (確率的レベルと確率的季節)
:
は確率的レベル、
は確率的季節、
は干渉変数の係数。
補足 テキスト記載の数式は3項の季節要素、説明変数、干渉変数を含む 6 状態が記載されている。季節要素が3項のため英国インフレーションモデルについて記載していると思われるが、このモデルには説明変数がないため状態数があわない。そのため、テキスト中の式を英国インフレーションモデルに合うよう書き換えている。
y <- ukinflation ukpulse <- rep(0, length.out = length(ukinflation)) ukpulse[4*(1975-1950)+2] <- 1 ukpulse[4*(1979-1950)+3] <- 1 ukpulse <- ts(ukpulse, start = start(ukinflation), frequency = frequency(ukinflation)) x <- ukpulse parm <- log(c(var(y), 0.00001, 0.00001)) buildObj <- function(parm) { dlm(FF = matrix(c(1, 1, 0, 0, 1), 1, 5), V = matrix(c(exp(parm[1])), 1, 1), GG = matrix(c(1, 0, 0, 0, 0, 0, -1, -1, -1, 0, 0, 1, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1), 5, 5, byrow = TRUE), W = matrix(c(exp(parm[2]), 0, 0, 0, 0, 0, exp(parm[3]), 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0), 5, 5, byrow = TRUE), m0 = c(0, 0, 0, 0, 0), C0 = matrix(c(1e+07, 0, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 0, 1e+07, 0, 0, 0, 0, 0, 1e+07), 5, 5, byrow = TRUE), JFF = matrix(c(0, 0, 0, 0, 1), 1, 5), X = as.matrix(x)) } dlmFitObj <- dlmMLE(y, parm = parm, build = buildObj) (dlmModObj <- buildObj(dlmFitObj$par)) # $FF # [,1] [,2] [,3] [,4] [,5] # [1,] 1 1 0 0 1 # # $V # [,1] # [1,] 2.27569e-05 # # $GG # [,1] [,2] [,3] [,4] [,5] # [1,] 1 0 0 0 0 # [2,] 0 -1 -1 -1 0 # [3,] 0 1 0 0 0 # [4,] 0 0 1 0 0 # [5,] 0 0 0 0 1 # # $W # [,1] [,2] [,3] [,4] [,5] # [1,] 1.783797e-05 0.000000e+00 0 0 0 # [2,] 0.000000e+00 4.310206e-07 0 0 0 # [3,] 0.000000e+00 0.000000e+00 0 0 0 # [4,] 0.000000e+00 0.000000e+00 0 0 0 # [5,] 0.000000e+00 0.000000e+00 0 0 0 # # $JFF # [,1] [,2] [,3] [,4] [,5] # [1,] 0 0 0 0 1 # # $X # [,1] # [1,] 0 # [2,] 0 # [3,] ... # # $m0 # [1] 0 0 0 0 0 # # $C0 # [,1] [,2] [,3] [,4] [,5] # [1,] 1e+07 0e+00 0e+00 0e+00 0e+00 # [2,] 0e+00 1e+07 0e+00 0e+00 0e+00 # [3,] 0e+00 0e+00 1e+07 0e+00 0e+00 # [4,] 0e+00 0e+00 0e+00 1e+07 0e+00 # [5,] 0e+00 0e+00 0e+00 0e+00 1e+07 p <- autoplot(dlmFilter(y, dlmModObj), fitted.colour = 'red', fitted.linetype = 'dashed') autoplot(dlmSmooth(y, dlmModObj), colour = 'blue', p = p)

モデルの作り方
とはいえ、最初から上式のような形でモデルが定義できていることはないと思う。まずは簡単なモデルを dlm::dlmModPoly などで作って、それをベースに書き換えていくのが良さそう。作ったモデルに適当なパラメータを与えて初期化すれば、モデルの次元 / 中身を確認できる。
y <- ukdrivers parm<- log(c(var(y), 0.001)) buildFun <- function(parm){ dlmModPoly(order = 2, dV = exp(parm[1]), dW = c(exp(parm[2]), 0)) } buildFun(parm) # $FF # [,1] [,2] # [1,] 1 0 # # $V # [,1] # [1,] 0.02935256 # # $GG # [,1] [,2] # [1,] 1 1 # [2,] 0 1 # # $W # [,1] [,2] # [1,] 0.001 0 # [2,] 0.000 0 # # $m0 # [1] 0 0 # # $C0 # [,1] [,2] # [1,] 1e+07 0e+00 # [2,] 0e+00 1e+07
R で 状態空間モデル: {dlm} の対数尤度計算について
状態空間モデルを扱う R パッケージである {dlm} では、モデルのパラメータ推定を最尤法で行なう。その際に使われる対数尤度の計算式はパッケージによって違いがあるようで、推定結果が自分の参照しているテキストとずれる。その違いを確かめたい。
当該のテキストはこちら。今回はこれを持っている方向けの内容。

- 作者: J.J.F.コマンダー,S.J.クープマン,Jacques J.F. Commandeur,Sime Jan Koopman,和合肇
- 出版社/メーカー: シーエーピー出版
- 発売日: 2008/09
- メディア: 単行本
- 購入: 2人 クリック: 4回
- この商品を含むブログを見る
参考 サイト 「状態空間時系列分析入門」をRで再現する はこのテキストを {dlm}, {KFAS} で再現しており、対数尤度の差異についても記載がある。
テキストでは単変量状態空間モデルの 時刻 における観測値
と状態
を以下の式であらわしている。
1期先の状態を更新するカルマンフィルタは、
: カルマンゲイン
: 状態推定誤差分散
: カルマンフィルタでの1期先予測誤差分散
これらを用いて、対数尤度関数は式 (8.7) で以下のように定義されている。
: 観測時系列の長さ
: 推定する状態数
: カルマンフィルタの1期先予測誤差
この式で計算される対数尤度は {dlm} のものと異なる。差異がある理由として記載する 2 点がわかったが、これらを解消してもまだ結果はあわない。
1. テキストの記載による差異
まず、テキストの各モデルに対数尤度として記載されている数値は 上式の計算結果そのものでない。P162 に添付されているプログラムに 対数尤度を系列の長さ で割る処理がしれっと入っており、処理後の値が対数尤度として記載されている。そのため、対数尤度に戻すためには各モデルに記載されている数値に 系列の長さ
をかける必要がある。
例えば 2.1 ローカルレベルモデル (確定的レベルモデル) に対数尤度として記載されている数値 0.3297597 に、系列の長さ 192 をかけた 63.31386 が 式 (8.7) の計算結果にあたる。
2. {dlm}の対数尤度計算式との差異
テキストで利用しているデータ利用して、{dlm} との差異を確かめたい。まずデータを読み込む。データは著者のサポートサイト、An Introduction to State Space Time Series Analysis から入手できる。
ukdrivers <- read.table('UKdriversKSI.txt', skip = 1) ukdrivers <- ts(ukdrivers[[1]], start = c(1969, 1), frequency = 12) ukdrivers <- log(ukdrivers) y <- ukdrivers
パラメータを受け取ってローカルレベルモデルを返す関数 buildFun を定義し、dlm::dlmMLE で最尤推定する。結果は リストになり、推定されたパラメータ par や 対数尤度 (を代替するもの/後述) value がプロパティとして含まれている。
library(dlm) parm <- c(var(y)) buildFun <- function(x) { dlmModPoly(order = 1, dV = parm, dW = 0) } dlmFit <- dlmMLE(y, parm = parm, build = buildFun) class(dlmFit) # [1] "list" names(dlmFit) # [1] "par" "value" "counts" "convergence" "message" dlmFit$par # [1] 0.02935256
続けて、最尤推定したパラメータ dlmFit$par を使ってモデルを作成する。プロパティの意味は vignette の記載がわかりやすい。テキストと式/添字をあわせると
FF:に対応。
V:に対応。
GG:に対応。
W:に対応。
m0:の事前分布の平均。
C0:の事前分布の分散。
J...: 各パラメータが時間変化するとき利用。
dlmMod <- buildFun(dlmFit$par) class(dlmMod) # [1] "dlm" names(dlmMod) # [1] "m0" "C0" "FF" "V" "GG" "W" "JFF" "JV" "JGG" "JW" V(dlmMod) # [,1] # [1,] 0.02935256
ここで、{dlm} の内部で最尤推定がどのように行なわれているのか確かめたい。
dlm::dlmMLE の定義をみると、最尤推定は dlm::dlmLL を optim で最適化することで行っている。
dlmMLE # function (y, parm, build, method = "L-BFGS-B", ..., debug = FALSE) # { # logLik <- function(parm, ...) { # mod <- build(parm, ...) # return(dlmLL(y = y, mod = mod, debug = debug)) # } # out <- optim(parm, logLik, method = method, ...) # return(out) # } # <environment: namespace:dlm>
dlm::dlmLL はその名前から対数尤度のようにも見えるが、対数尤度とは符号が逆 ( optim で最適化するため、尤もらしいほど値が小さくなる )。
dlmLL(y = y, mod = buildFun(dlmFit$par)) # [1] -230.7721
補足 パラメータ推定時の dlm::dlmLL の値は dlm::dlmMLE の結果にも含まれている。
dlmFit$value # [1] -230.7721
dlm::dlmLL の処理は少し条件分岐が多いため省略するが、上のモデルについて必要な処理は以下のようなものであることがわかる。カルマンフィルタ dlm::dlmFilter を適用した結果から予測誤差、予測誤差標準偏差を求めて 対数尤度の代替となる値を算出している。
# 予測誤差、予測誤差標準偏差を計算 residuals_dlmcompat <- function (object) { mod <- object$mod # yは観測値 (入力), a は状態の予測値 res <- drop(object$y - object$a %*% t(mod$FF)) f <- function(i) { # 予測誤差標準偏差 diag(crossprod(object$D.R[i, ] * t(mod$FF %*% object$U.R[[i]])) + mod$V) } SD <- drop(t(sqrt(sapply(seq(along = object$U.R), f)))) return(list(res = res, sd = SD)) } # dlmLLと同じ値を計算 LL_dlmcompat <- function(y, mod) { filtered <- dlmFilter(y, mod) resid <- residuals_dlmcompat(filtered) # 予測誤差分散 F <- as.numeric(resid$sd)^2 v <- as.numeric(resid$res) return(0.5 * sum(log(F) + v^2/F)) } LL_dlmcompat(y = y, mod = buildFun(dlmFit$par)) # [1] -230.7721
これを上式 (8.7) と比べると以下の差異がある。
- 符号が逆 (
optimでの処理のため ) - 第1項目がない ( パラメータ非依存のため
optimには影響ない ) - 第2項目の総和の範囲が異なる
これをテキストに準じて書き換えると、
LL_ckbookcompat <- function(y, mod) { n <- length(y) filtered <- dlmFilter(y, mod) resid <- residuals_dlmcompat(filtered) F <- (as.numeric(resid$sd)^2)[2:n] v <- as.numeric(resid$res)[2:n] return(-0.5 * (n * log(2 * pi) + sum(log(F) + v^2/F))) } LL_ckbookcompat(y = y, mod = buildFun(dlmFit$par)) # [1] 62.39492
となり、テキストの対数尤度 63.31386 に結構 近づいた (が、あわない、、)。対数尤度の計算結果はパッケージによって異なる、ということは覚えておいたほうがよさそうだ。
補足: このモデルにおいては、 {KFAS} の結果も上記修正後の結果と一致している。
library(KFAS) kfasMod <- SSModel(y ~ SSMtrend(degree=1, Q=list(matrix(0))), H = matrix(NA)) kfasFit <- fitSSM(kfasMod, inits = parm, method = "BFGS") kfasFit$optim.out$value # [1] -62.39492 KFS(kfasFit$model)$logLik # [1] 62.39492
pandas 日時まわりのリサンプリング/オフセット処理
こちらの続き。
今回のサンプルデータには自分の歩数のデータを使いたい。インスパイヤ元は以下のサイトだ。
データの読み込み
歩数データは iPhone の Health アプリから Export できる。形式は XML なので、そのままでは pandas で読み込めない。一度 XML から必要な属性を辞書のリストとして取り出した後、pandas に読み込ませる。
import pandas as pd from xml.etree import ElementTree tree = ElementTree.parse('export.xml') root = tree.getroot() # 属性の辞書のリストを作る data = [e.attrib for e in root.findall('.//Record')] data[0] # {'endDate': '20150511220900+0900', # 'recordCount': '104', # 'source': u'xxx', # 'startDate': '20150511210900+0900', # 'type': 'HKQuantityTypeIdentifierStepCount', # 'unit': 'count', # 'value': '531'} df = pd.DataFrame(data) # 必要な列のみにフィルタ df = df[['startDate', 'type', 'unit', 'value']] df.head() # startDate type unit value # 0 20150511210900+0900 HKQuantityTypeIdentifierStepCount count 531 # 1 20150512000900+0900 HKQuantityTypeIdentifierStepCount count 6 # 2 20150512060900+0900 HKQuantityTypeIdentifierStepCount count 629 # 3 20150512070900+0900 HKQuantityTypeIdentifierStepCount count 312 # 4 20150512080900+0900 HKQuantityTypeIdentifierStepCount count 1483
Series.value_counts() を使うと、ある列で出現する要素の個数を集計できる。"type" 列を集計すると、Export したデータには歩数以外のデータも含まれていることがわかる。歩数以外は不要なので削除する。
df['type'].value_counts() # HKQuantityTypeIdentifierStepCount 375 # HKQuantityTypeIdentifierDistanceWalkingRunning 374 # HKQuantityTypeIdentifierFlightsClimbed 216 # dtype: int64 # 歩数のみにフィルタ df = df[df['type'] == 'HKQuantityTypeIdentifierStepCount']
また、XML で読み込んだデータはすべて文字列型になっているため、適宜型変換を行う。
# 歩数データは数値に df['value'] = df['value'].astype(float) # 日付は index として設定し、日付型 (DatetimeIndex) に変換 df = df.set_index('startDate') # この時点で index は文字列型になっている df.index # Index([u'20150511210900+0900', u'20150512000900+0900', u'20150512060900+0900', # u'20150512070900+0900', u'20150512080900+0900', u'20150512090900+0900', # ... # u'20150518165000+0900', u'20150518165900+0900', u'20150518171600+0900', # u'20150518181200+0900'], # dtype='object', name=u'startDate', length=965) # to_datetime で日時型に変換 / タイムゾーンを表すオフセットを適宜設定 pd.to_datetime(df.index, utc=True).tz_convert('Asia/Tokyo') # DatetimeIndex(['2015-05-11 21:09:00+09:00', '2015-05-12 00:09:00+09:00', # '2015-05-12 06:09:00+09:00', '2015-05-12 07:09:00+09:00', # ... # '2015-05-18 16:50:00+09:00', '2015-05-18 16:59:00+09:00', # '2015-05-18 17:16:00+09:00', '2015-05-18 18:12:00+09:00'], # dtype='datetime64[ns]', length=965, freq=None, tz='Asia/Tokyo') # index を上書き df.index = pd.to_datetime(df.index, utc=True).tz_convert('Asia/Tokyo') # タイムゾーンは使わないので削除 df.index = df.index.tz_localize(None) # 日付の昇順にソート df = df.sort_index() df.head() # type unit value # 2014-11-25 21:09:00 HKQuantityTypeIdentifierStepCount count 1396.00 # 2014-11-26 21:09:00 HKQuantityTypeIdentifierStepCount count 7020.37 # 2014-11-27 21:09:00 HKQuantityTypeIdentifierStepCount count 6413.63 # 2014-11-28 21:09:00 HKQuantityTypeIdentifierStepCount count 8396.00 # 2014-11-29 21:09:00 HKQuantityTypeIdentifierStepCount count 12411.00
今の機種に買い替えたのが昨年11末なので、買い替え以降すべてのデータが入っているようだ。
部分文字列によるスライシング
pandas での一般的なデータ選択については以下の記事にまとめた。
加えて、データが 日付型の Index ( DatetimeIndex ) を持つとき、日付-like な文字列で __getitem__ すると、その期間にあてはまるデータを抽出してくれる。例えば 2015-05-17 日分のデータを抽出したければ以下のようにする。この方法を使うと 好きな期間のデータを簡単に確認することができる。詳細は公式ドキュメントを参照。
df['2015-05-17'] # type unit value # 2015-05-17 08:09:00 HKQuantityTypeIdentifierStepCount count 18.0000 # 2015-05-17 11:09:00 HKQuantityTypeIdentifierStepCount count 180.0000 # 2015-05-17 12:09:00 HKQuantityTypeIdentifierStepCount count 934.0000 # 2015-05-17 13:09:00 HKQuantityTypeIdentifierStepCount count 469.0000 # ... ... ... ... # 2015-05-17 21:16:00 HKQuantityTypeIdentifierStepCount count 88.5087 # 2015-05-17 21:17:00 HKQuantityTypeIdentifierStepCount count 23.1214 # 2015-05-17 21:18:00 HKQuantityTypeIdentifierStepCount count 99.3283 # 2015-05-17 21:19:00 HKQuantityTypeIdentifierStepCount count 41.6284 # # [14 rows x 3 columns]
リサンプリング
データは日時で処理したいので、まずは 1日ごとの合計を算出したい。こういうときは DataFrame.resample。リサンプリングする期間や集約関数は引数として指定できる。
s = df.resample('D', how='sum') # value # 2014-11-25 1396.000000 # 2014-11-26 7020.370000 # 2014-11-27 6413.630000 # 2014-11-28 8396.000000 # ... ... # 2015-05-15 7561.998000 # 2015-05-16 10615.000000 # 2015-05-17 9208.000000 # 2015-05-18 8085.994898 # # [175 rows x 1 columns]
日次に集約した結果をプロットしてみる。
s.plot()
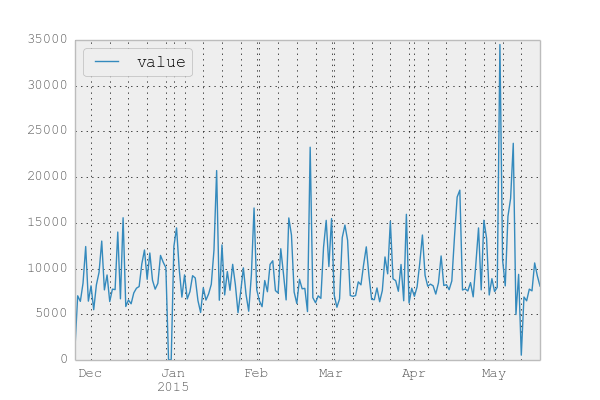
あまり意識はしていないのだが そこそこ歩いているようだ。5月初旬はちょっと出かけていたため 歩数に異常値が出ている。
日付の標準化
日時データの適当な期間での集約は DataFrame.resample でできた。が、時には日時の補正のみを行い、集約はしたくない場合がある。
DatetimeIndex を日付ごとにまとめるのに一番簡単なのは .normalize。
pd.Timestamp('2015-05-01 23:59').normalize() # Timestamp('2015-05-01 00:00:00') pd.Timestamp('2015-05-02 00:00').normalize() # Timestamp('2015-05-02 00:00:00')
他の日時関連のメソッドと同じく、Timestamp, DatetimeIndex 両方で使える。
df.tail().index # DatetimeIndex(['2015-05-18 18:55:00', '2015-05-18 18:56:00', # '2015-05-18 18:57:00', '2015-05-18 18:58:00', # '2015-05-18 18:59:00'], # dtype='datetime64[ns]', freq=None, tz=None) df.tail().index.normalize() # DatetimeIndex(['2015-05-18', '2015-05-18', # '2015-05-18', '2015-05-18', # '2015-05-18'], # dtype='datetime64[ns]', freq=None, tz=None)
当然、集約すれば 日付での .resample と同じ結果になる。
s = df.groupby(df.index.normalize())[['value']].sum() s # 略
オフセット
また、より柔軟に日時補正を行うためにオフセットが提供されている ( 前回記事 )。オフセットを利用してデータを補正/集約する例を書きたい。
オフセットを使わずに .resample で月次平均をとると以下のような結果になる。
s.resample('M', how='mean') # value # 2014-11-30 7012.500000 # 2014-12-31 8435.741935 # 2015-01-31 9134.032258 # 2015-02-28 9323.821429 # 2015-03-31 9326.356129 # 2015-04-30 9938.533333 # 2015-05-31 10973.055439
オフセットを使って同じ処理をするには、日時を月末に補正するオフセット MonthEnd を使う。オフセットの一覧は 公式ドキュメント を参照。
m = offsets.MonthEnd()
m
# <MonthEnd>
オフセットにはいくつか共通のメソッドがあるため、順に記載する。
まず、ある日付が 当該のオフセット上に存在するか調べるには .onOffset。MonthEnd.onOffset の場合は、引数が月末の日付であるとき True になる。
m.onOffset(pd.Timestamp('2015-04-29')) # False m.onOffset(pd.Timestamp('2015-04-30')) # True m.onOffset(pd.Timestamp('2015-05-01')) # False
オフセットを加減算することにより、日時を次の/前のオフセットへと移動できる。また、オフセットの加算と同一の処理として .apply がある。
# 日付を常に 次のオフセットへ移動させる。 pd.Timestamp('2015-04-29') + m # Timestamp('2015-04-30 00:00:00') pd.Timestamp('2015-04-30') + m # Timestamp('2015-05-31 00:00:00') pd.Timestamp('2015-05-01') + m # Timestamp('2015-05-31 00:00:00') # 日付を常に 前のオフセットへ移動させる。 pd.Timestamp('2015-04-29') - m # Timestamp('2015-03-31 00:00:00') pd.Timestamp('2015-04-30') - m # Timestamp('2015-03-31 00:00:00') pd.Timestamp('2015-05-01') - m # Timestamp('2015-04-30 00:00:00') # 日付を常に 次のオフセットへ移動させる。 m.apply(pd.Timestamp('2015-04-29')) # Timestamp('2015-04-30 00:00:00') m.apply(pd.Timestamp('2015-04-30')) # Timestamp('2015-05-31 00:00:00') m.apply(pd.Timestamp('2015-05-01')) # Timestamp('2015-05-31 00:00:00')
オフセットに乗っている日時は動かしたくなければ .rollforward / .rollback というメソッドを使う。
# 日付がオフセットに乗っていない場合 次のオフセットへ移動させる。 m.rollforward(pd.Timestamp('2015-04-29')) # Timestamp('2015-04-30 00:00:00') m.rollforward(pd.Timestamp('2015-04-30')) # Timestamp('2015-04-30 00:00:00') m.rollforward(pd.Timestamp('2015-05-01')) # Timestamp('2015-05-31 00:00:00') # 日付がオフセットに乗っていない場合 前のオフセットへ移動させる。 m.rollback(pd.Timestamp('2015-04-29')) # Timestamp('2015-03-31 00:00:00') m.rollback(pd.Timestamp('2015-04-30')) # Timestamp('2015-04-30 00:00:00') m.rollback(pd.Timestamp('2015-05-01')) # Timestamp('2015-04-30 00:00:00')
よって、月次平均の算出をオフセットのメソッドを使って行う場合は以下のようになる。
# 例示のため一部データをスライス s['2014-11'] # value # 2014-11-25 1396.00 # 2014-11-26 7020.37 # 2014-11-27 6413.63 # 2014-11-28 8396.00 # 2014-11-29 12411.00 # 2014-11-30 6438.00 # 日時を月末に補正 s['2014-11'].index.map(m.rollforward) # array([Timestamp('2014-11-30 00:00:00'), Timestamp('2014-11-30 00:00:00'), # Timestamp('2014-11-30 00:00:00'), Timestamp('2014-11-30 00:00:00'), # Timestamp('2014-11-30 00:00:00'), Timestamp('2014-11-30 00:00:00')], dtype=object) s.groupby(s.index.map(m.rollforward)).mean() # value # 2014-11-30 7012.500000 # 2014-12-31 8435.741935 # 2015-01-31 9134.032258 # 2015-02-28 9323.821429 # 2015-03-31 9326.356129 # 2015-04-30 9938.533333 # 2015-05-31 10973.055439
補足 オフセットは .resample の引数として渡すこともでき、同じ結果になる。
s.resample(m, how='mean') # 略
オフセットを活用した集計
つづけて、カレンダー上の平日 / 休日での差異をみたい。アクセサ ( こちらの記事参照 ) を使って曜日で集計してから処理してもよいが、 BusinessDay オフセットを使えば簡単。BusinessDay.onOffset を使えば平日が True / 休日を False として集計できる。以下の結果をみると、休日の方が歩いている傾向があるようだ。
bday = offsets.BusinessDay() s.groupby(bday.onOffset).mean() # value # False 10860.216800 # True 8716.657583
BusinessDay オフセットは 土日のみを休日として扱うが、任意の祝日も含めて休日として扱いたい場合は CustomBusinessDay オフセットが使える。拙作のパッケージ で 日本の祝日のカレンダーを定義しているので、それを使って、
import japandas as jpd calendar = jpd.JapaneseHolidayCalendar() cday = pd.offsets.CDay(calendar=calendar) s.groupby(cday.onOffset).mean() # value # False 10799.033448 # True 8600.419640
これまでの内容を使って、月ごとに 平日 / 土日 / 祝日の歩数平均をクロス集計したい。クロス集計を行うには pd.pivot_table 。
avg = pd.pivot_table(s, index=s.index.map(m.rollforward), columns=[s.index.map(bday.onOffset), s.index.map(cday.onOffset)], values='value', aggfunc='mean') avg # False True # False False True # 2014-11-30 9424.500000 NaN 5806.500000 # 2014-12-31 10382.275000 11704.0 7579.354545 # 2015-01-31 9534.193333 10162.5 8851.113000 # 2015-02-28 10943.125000 9446.0 8635.578947 # 2015-03-31 11148.877778 NaN 8580.779091 # 2015-04-30 11349.625000 7110.0 9535.666667 # 2015-05-31 12769.000000 11582.7 9572.544211
左から、
- 土日:
BusinessDayCustomBusinessDayともにFalse - 祝日:
BusinessDayはTrue,CustomBusinessDayはFalse - 平日:
BusinessDayCustomBusinessDayともにTrue
となる。ということで上の結果からも 5月の休日は結構歩いたなってことがわかった。
まとめ
日付を index として持つデータに対する リサンプリング/オフセット処理をまとめた。これらは以下のような使い分けをすればよいと思う。
- 比較的単純な集約はリサンプリング
- リサンプリングではできない より細かい補正やクロス集計をしたい場合はオフセット

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る
pandas 0.16.0/0.16.1 の主要な新機能
先日 5/11 に pandas 0.16.1 がリリースされた。前バージョンである 0.16.0 とあわせて、主要な変更点である以下3点の概要をまとめたい。各見出しの括弧内には対応したバージョンを記載した。
- 簡単な列追加 /
DataFrame.assign(0.16.0) - 文字列処理の強化 (0.16.0/0.16.1)
- ランダムサンプリング
DataFrame.sample(0.16.1)
変更点全体はリリースノートを参照。上記3点以外にも便利な変更はあるのだが、 Categorical や Frequency など 元機能の説明がないとわからない箇所なので別途、、。
簡単な列追加 / DataFrame.assign (0.16.0)
DataFrame への列追加をより簡潔に行うためのメソッドとして、DataFrame.assign が追加された。R {dplyr} の mutate 関数に似た書式で複数列の追加ができる。
これまで列追加の際には カラムを指定して代入し、元データを破壊的に変更する必要があった。
import pandas as pd df = pd.DataFrame({'A':[1, 2, 3], 'B':[4, 5, 6]}) df # A B # 0 1 4 # 1 2 5 # 2 3 6 # これまでの列追加 df['C'] = [7, 8, 9] df # A B C # 0 1 4 7 # 1 2 5 8 # 2 3 6 9
assign を使うと、作成したい列名をキーワード引数として以下のように書ける。また、戻り値は元データに列追加したコピーとなり、元データ自体は変更されない。
# D列, E列を追加 df.assign(D=[10, 11, 12], E=[13, 14, 15]) # A B C D E # 0 1 4 7 10 13 # 1 2 5 8 11 14 # 2 3 6 9 12 15 # 元データはそのまま df # A B C # 0 1 4 7 # 1 2 5 8 # 2 3 6 9
キーワード引数の値には通常の列作成時と同じ型が渡せる。各列の値を組み合わせた列 + ダミー列を同時に作りたければ、
df.assign(AB=df['A'] * df['B'], AC=df['A'] + df['C'], dummy=1) # A B C AB AC dummy # 0 1 4 7 4 8 1 # 1 2 5 8 10 10 1 # 2 3 6 9 18 12 1
キーワード引数として渡せない列名、例えば記号や日本語を含む列名は、一度 辞書にしてから渡せばよい。
keys = {'A*B': df['A'] * df['B'], 'A+B':df['A'] + df['B']}
df.assign(**keys)
# A B A*B A+B
# 0 1 4 4 5
# 1 2 5 10 7
# 2 3 6 18 9
補足: メソッド自体は 0.16.0 で追加され、0.16.1 でキーワード引数の処理順序がアルファベット順に固定された。
文字列処理の強化 (0.16.0/0.16.1)
pandas での文字列処理について過去に以下の記事を書いたことがあるのだが、当時はアクセサから利用できるメソッドが限定されていて、少し手間がかかるところがあった。
0.16.0, 0.16.1 で、.str アクセサに以下のメソッド群が追加された。それぞれ、 Python 標準の文字列メソッドと同一の処理を Series 内の値に対して適用するもの。
| Methods | ||||
|---|---|---|---|---|
isalnum |
isalpha |
isdigit |
isspace |
islower |
isupper |
istitle |
isnumeric |
isdecimal |
find |
rfind |
ljust |
rjust |
zfill |
capitalize |
swapcase |
normalize |
partition |
rpartition |
index |
rindex |
translate |
補足 str.normalize は ユニコード正規化 ( unicodedata.normalize ) を値に対して適用するもの。
補足 .str アクセサから利用可能なメソッド全体はこちら。
df = pd.DataFrame({'A': ['xxx', '3', 'yyy'], 'B': [1, 2, 3]})
df
# A B
# 0 xxx 1
# 1 3 2
# 2 yyy 3
# 先頭を大文字に
df['A'].str.capitalize()
# 0 Xxx
# 1 3
# 2 Yyy
# Name: A, dtype: object
# 文字列が数値がどうかを調べる
df['A'].str.isdigit()
# 0 False
# 1 True
# 2 False
# Name: A, dtype: bool
# 5桁分を 0 パディング
df['A'].str.zfill(5)
# 0 00xxx
# 1 00003
# 2 00yyy
# Name: A, dtype: object
やりたいことに応じて適当に組み合わせると、かなり柔軟な処理がかける。例えば 数値の文字列のみを 0 パディングしたければ、
df.loc[df['A'].str.isdigit(), 'A'] = df['A'].str.zfill(5) df # A B # 0 xxx 1 # 1 00003 2 # 2 yyy 3
補足 DataFrame.loc の意味はこちらの記事参照。
また、0.16.1 から .str アクセサを Index からも呼び出せるようになった。これまでは 列名/行名に対する文字列処理は .map を使って関数適用する必要があったが、.str を使えば以下のように書ける。
df # A B # 0 xxx 1 # 1 00003 2 # 2 yyy 3 df.columns.str.lower() # Index([u'a', u'b'], dtype='object') # 列名を小文字に変更 df.columns = df.columns.str.lower() df # a b # 0 xxx 1 # 1 00003 2 # 2 yyy 3
str.split のように複数の値を返しうる処理については、expand オプションを利用して 返り値の型を制御できる。互換性維持のため、既定値はメソッドにより異なる。APIドキュメントを参照。
expand=False: 返り値の次元を増やさない = 返り値はSeriesもしくはIndexexpand=True: 返り値の次元を増やす = 返り値はDataFrameもしくはMultiIndex
補足 これまで同様の制御をおこなっていた return_type オプションは deprecate されており、将来のバージョンで削除される。
s = pd.Series(['a,b', 'a,c', 'b,c']) # 返り値は Series s.str.split(',') # 0 [a, b] # 1 [a, c] # 2 [b, c] # dtype: object # 返り値は DataFrame s.str.split(',', expand=True) # 0 1 # 0 a b # 1 a c # 2 b c idx = pd.Index(['a,b', 'a,c', 'b,c']) # 返り値は 1 レベルの Index idx.str.split(',') # Index([[u'a', u'b'], [u'a', u'c'], [u'b', u'c']], dtype='object') # 返り値は 2 レベルの MultiIndex idx.str.split(',', expand=True) # MultiIndex(levels=[[u'a', u'b'], [u'b', u'c']], # labels=[[0, 0, 1], [0, 1, 1]])
ランダムサンプリング DataFrame.sample (0.16.1)
Series, DataFrame, Panel から適当なデータをサンプリングするためのメソッドとして、.sample が追加された。
ここでは DataFrame.sample を例としてその処理を記載する。まず以下のようなデータを用意した。
df = pd.DataFrame({'A': [1, 2 ,3, 4, 5], 'B': ['a', 'b', 'c', 'd', 'e']})
df
# A B
# 0 1 a
# 1 2 b
# 2 3 c
# 3 4 d
# 4 5 e
DataFrame.sample は 既定では 1 行をランダムにサンプリングする。
n: サンプルサイズを指定する。既定は 1。axis: サンプリングするレコードの方向を指定する。0(既定) で行のサンプリング、1で列のサンプリング。
# 1 行の抽出 df.sample() # A B # 3 4 d # 3 行の抽出 df.sample(3) # A B # 0 1 a # 4 5 e # 2 3 c # 1 列の抽出 df.sample(axis=1) # A # 0 1 # 1 2 # 2 3 # 3 4 # 4 5
また、サンプルサイズでなく抽出比を指定する場合は frac を指定する。
df.sample(frac=0.4) # A B # 3 4 d # 1 2 b
最後に、replace オプションを利用して 抽出方法を変更することができる。
replace=False(既定): 非復元抽出。サンプリングされた各要素の重複を許さない。replace=True: 復元抽出。各要素の重複を許す。
# 復元抽出。2行目、4行目が重複 df.sample(n=4, replace=True) # A B # 1 2 b # 2 3 c # 0 1 a # 2 3 c # 非復元抽出では 元のデータ数以上のサンプルサイズは取れない df.sample(n=6) # ValueError: Cannot take a larger sample than population when 'replace=False' df.sample(n=6, replace=True) # A B # 1 2 b # 4 5 e # 0 1 a # 1 2 b # 2 3 c # 4 5 e
補足 これまでは index の要素をサンプリングして選択する必要があった。以下の記事参照。
まとめ
0.16.0 / 0.16.1 の変更点のうち、以下 3 点の概要をまとめた。
- 簡単な列追加 /
DataFrame.assign(0.16.0) - 文字列処理の強化 (0.16.0/0.16.1)
- ランダムサンプリング
DataFrame.sample(0.16.1)
他にも、バグ修正 / 挙動・仕様の統一など 様々な改善が入っているので使ってみてください。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (9件) を見る
簡単な集約/変換処理を PySpark & pandas の DataFrame で行う
こちらの続き。
準備
サンプルデータは iris 。今回は HDFS に csv を置き、そこから読み取って DataFrame を作成する。
# HDFS にディレクトリを作成しファイルを置く $ hadoop fs -mkdir /data/ $ hadoop fs -put iris.csv /data/ $ hadoop fs -ls / Found 1 items drwxr-xr-x - ec2-user supergroup 0 2015-04-28 20:01 /data # Spark のパスに移動 $ echo $SPARK_HOME /usr/local/spark $ cd $SPARK_HOME $ pwd /usr/local/spark $ bin/pyspark
補足 前回同様に pandas から直接 PySpark の DataFrame を作成した場合、groupBy 時に java.lang.OutOfMemoryError: Java heap space エラーが発生してシェルごと落ちる。
CSV ファイルの読み込み
pandas では前回同様 read_csv。
import numpy as np import pandas as pd # 表示する行数を設定 pd.options.display.max_rows=10 names = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth', 'Species'] # pandas pdf = pd.read_csv('~/iris.csv', header=None, names=names) pdf # 略
PySpark は標準では csv から直接 DataFrame を作成できないため、一度 Row のリストを作成して DataFrame に変換する。
from pyspark.sql import Row lines = sc.textFile("hdfs://127.0.0.1:9000/data/iris.csv") cells = lines.map(lambda l: l.split(",")) rows = cells.map(lambda x: Row(SepalLength=float(x[0]), SepalWidth=float(x[1]), PetalLength=float(x[2]), PetalWidth=float(x[3]), Species=x[4])) sdf = sqlContext.createDataFrame(rows) sdf.show() # 略
グルーピング/集約
ある列の値ごとに集計
pandas, PySpark で多少 文法は異なる。
列の値でグループ分けし、一列の合計を取得する場合:
# pandas pdf.groupby('Species')['SepalLength'].sum() # Species # setosa 250.3 # versicolor 296.8 # virginica 329.4 # Name: SepalLength, dtype: float64 # PySpark sdf.groupBy('Species').sum('SepalLength').show() # Species SUM(SepalLength) # virginica 329.3999999999999 # versicolor 296.8 # setosa 250.29999999999998
指定した複数列の合計を取得する場合:
# pandas pdf.groupby('Species')[['PetalWidth', 'PetalLength']].sum() # PetalWidth PetalLength # Species # setosa 12.2 73.2 # versicolor 66.3 213.0 # virginica 101.3 277.6 # PySpark sdf.groupBy('Species').sum('PetalWidth', 'PetalLength').show() # Species SUM(PetalWidth) SUM(PetalLength) # virginica 101.29999999999998 277.59999999999997 # versicolor 66.30000000000001 213.0 # setosa 12.199999999999996 73.2
全列の合計を取得する場合:
# pandas pdf.groupby('Species').sum() # SepalLength SepalWidth PetalLength PetalWidth # Species # setosa 250.3 170.9 73.2 12.2 # versicolor 296.8 138.5 213.0 66.3 # virginica 329.4 148.7 277.6 101.3 # PySpark sdf.groupBy('Species').sum().show() # Species SUM(PetalLength) SUM(PetalWidth) SUM(SepalLength) SUM(SepalWidth) # virginica 277.59999999999997 101.29999999999998 329.3999999999999 148.7 # versicolor 213.0 66.30000000000001 296.8 138.5 # setosa 73.2 12.199999999999996 250.29999999999998 170.90000000000003
補足 pandas では グループ化したデータも DataFrame と同じようにスライシングできたりする。
一方、PySpark の GroupedData は集約系のAPI しか持っていない。
# pandas pdf.groupby('Species')['PetalWidth'] # <pandas.core.groupby.SeriesGroupBy object at 0x7f62f4218d50> # PySpark (NG!) sdf.groupBy('Species')[['Species']] # TypeError: 'GroupedData' object has no attribute '__getitem__' sdf.groupBy('Species').select('PetalWidth') # AttributeError: 'GroupedData' object has no attribute 'select'
また、pandas では apply で自作の集約関数 (UDAF) を利用することができるが、PySpark 1.3.1 時点 では非対応らしい。PySpark の udf を利用して定義した自作関数を集約時に使うと以下のエラーになる。
# pandas pdf.groupby('Species')[['PetalWidth', 'PetalLength']].apply(np.sum) # PetalWidth PetalLength # Species # setosa 12.2 73.2 # versicolor 66.3 213.0 # virginica 101.3 277.6 # PySpark (NG!) import pyspark.sql.functions np_sum = pyspark.sql.functions.udf(np.sum, pyspark.sql.types.FloatType()) sdf.groupBy('Species').agg(np_sum(sdf.PetalWidth)) # py4j.protocol.Py4JJavaError: An error occurred while calling o334.agg. # : org.apache.spark.sql.AnalysisException: expression 'pythonUDF' is neither present in the group by, nor is it an aggregate function. Add to group by or wrap in first() if you don't care which value you get.;
行持ち / 列持ち変換
複数列持ちの値を行持ちに展開 (unpivot / melt)
pandas では pd.melt。 DataFrame.melt ではないので注意。
# pandas pmelted = pd.melt(pdf, id_vars=['Species'], var_name='variable', value_name='value') pmelted # Species variable value # 0 setosa SepalLength 5.1 # 1 setosa SepalLength 4.9 # 2 setosa SepalLength 4.7 # 3 setosa SepalLength 4.6 # 4 setosa SepalLength 5.0 # .. ... ... ... # 595 virginica PetalWidth 2.3 # 596 virginica PetalWidth 1.9 # 597 virginica PetalWidth 2.0 # 598 virginica PetalWidth 2.3 # 599 virginica PetalWidth 1.8 # # [600 rows x 3 columns]
同様の処理を PySpark でやるには、DataFrame.flatMap。1行の入力に対して複数行 (この例では4行) のデータを返すことができる。fratMap の返り値は RDD インスタンスになるため、必要なら再度 DataFrame 化する。
# PySpark def mapper(row): return [Row(Species=row[4], variable='PetalLength', value=row[0]), Row(Species=row[4], variable='PetalWidth', value=row[1]), Row(Species=row[4], variable='SepalLength', value=row[2]), Row(Species=row[4], variable='SepalWidth', value=row[3])] smelted = sqlContext.createDataFrame(sdf.flatMap(mapper)) smelted.show() # Species value variable # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.1 SepalLength # setosa 3.5 SepalWidth # ... .. ... # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.0 SepalLength # setosa 3.6 SepalWidth smelted.count() # 600L
複数行持ちの値を列持ちに変換 (pivot)
pandas では DataFrame.pivot。pivotするデータは列にする値 (以下では Species ) と行にする値 (以下では variable ) の組がユニークになっている必要がある。そのため、まず pivot 用データを作成 -> その後 pivot する。
# pandas # pivot 用データを作成 punpivot = pmelted.groupby(['Species', 'variable']).sum() punpivot = punpivot.reset_index() punpivot # Species variable value # 0 setosa PetalLength 73.2 # 1 setosa PetalWidth 12.2 # 2 setosa SepalLength 250.3 # 3 setosa SepalWidth 170.9 # 4 versicolor PetalLength 213.0 # .. ... ... ... # 7 versicolor SepalWidth 138.5 # 8 virginica PetalLength 277.6 # 9 virginica PetalWidth 101.3 # 10 virginica SepalLength 329.4 # 11 virginica SepalWidth 148.7 # # [12 rows x 3 columns] # pivot punpivot.pivot(index='variable', columns='Species', values='value') # Species setosa versicolor virginica # variable # PetalLength 73.2 213.0 277.6 # PetalWidth 12.2 66.3 101.3 # SepalLength 250.3 296.8 329.4 # SepalWidth 170.9 138.5 148.7
PySpark の DataFrame のままでは同じ処理はできないようなので、一度 RDD に変換してから、 groupBy -> map
# PySpark # pivot 用データを作成 sunpivot = smelted.groupBy('Species', 'variable').sum() sunpivot.show() # Species variable SUM(value) # versicolor SepalWidth 138.5 # versicolor SepalLength 296.8 # setosa PetalLength 73.2 # virginica PetalWidth 101.29999999999998 # versicolor PetalWidth 66.30000000000001 # setosa SepalWidth 170.90000000000003 # virginica PetalLength 277.59999999999997 # setosa SepalLength 250.29999999999998 # versicolor PetalLength 213.0 # setosa PetalWidth 12.199999999999996 # virginica SepalWidth 148.7 # virginica SepalLength 329.3999999999999 def reducer(obj): # variable : value の辞書を作成 result = {o[1]:o[2] for o in obj[1]} return Row(Species=obj[0], **result) # pivot spivot = sunpivot.rdd.groupBy(lambda x: x[0]).map(reducer) spivot.collect() # [Row(PetalLength=277.59999999999997, PetalWidth=101.29999999999998, SepalLength=329.3999999999999, SepalWidth=148.7, Species=u'virginica'), # Row(PetalLength=73.2, PetalWidth=12.199999999999996, SepalLength=250.29999999999998, SepalWidth=170.90000000000003, Species=u'setosa'), # Row(PetalLength=213.0, PetalWidth=66.30000000000001, SepalLength=296.8, SepalWidth=138.5, Species=u'versicolor')] sqlContext.createDataFrame(spivot).show() # PetalLength PetalWidth SepalLength SepalWidth Species # 277.59999999999997 101.29999999999998 329.3999999999999 148.7 virginica # 73.2 12.199999999999996 250.29999999999998 170.90000000000003 setosa # 213.0 66.30000000000001 296.8 138.5 versicolor
列の分割 / 結合
列の値を複数列に分割
ある列の値を適当に文字列処理して、新しい列を作成したい。pandas には 文字列処理用のアクセサがあるため、 assign と組み合わせて以下のように書ける。
# pandas psplitted = pmelted.assign(Parts=pmelted['variable'].str.slice(0, 5), Scale=pmelted['variable'].str.slice(5)) psplitted # Species variable value Parts Scale # 0 setosa SepalLength 5.1 Sepal Length # 1 setosa SepalLength 4.9 Sepal Length # 2 setosa SepalLength 4.7 Sepal Length # 3 setosa SepalLength 4.6 Sepal Length # 4 setosa SepalLength 5.0 Sepal Length # .. ... ... ... ... ... # 595 virginica PetalWidth 2.3 Petal Width # 596 virginica PetalWidth 1.9 Petal Width # 597 virginica PetalWidth 2.0 Petal Width # 598 virginica PetalWidth 2.3 Petal Width # 599 virginica PetalWidth 1.8 Petal Width # # [600 rows x 5 columns]
PySparkには上記のようなメソッドはないので map で処理する。
# PySpark def splitter(row): parts = row[2][:5] scale = row[2][5:] return Row(Species=row[0], value=row[1], Parts=parts, Scale=scale) ssplitted = sqlContext.createDataFrame(smelted.map(splitter)) ssplitted.show() # Parts Scale Species value # Petal Length setosa 1.4 # Petal Width setosa 0.2 # Sepal Length setosa 5.1 # Sepal Width setosa 3.5 # Petal Length setosa 1.4 # .. .. ... .. # Petal Length setosa 1.4 # Petal Width setosa 0.2 # Sepal Length setosa 5.0 # Sepal Width setosa 3.6
複数列の値を一列に結合
pandas では普通に文字列結合すればよい。
# pandas psplitted['variable2'] = psplitted['Parts'] + psplitted['Scale'] psplitted # Species variable value Parts Scale variable2 # 0 setosa SepalLength 5.1 Sepal Length SepalLength # 1 setosa SepalLength 4.9 Sepal Length SepalLength # 2 setosa SepalLength 4.7 Sepal Length SepalLength # 3 setosa SepalLength 4.6 Sepal Length SepalLength # 4 setosa SepalLength 5.0 Sepal Length SepalLength # .. ... ... ... ... ... ... # 595 virginica PetalWidth 2.3 Petal Width PetalWidth # 596 virginica PetalWidth 1.9 Petal Width PetalWidth # 597 virginica PetalWidth 2.0 Petal Width PetalWidth # 598 virginica PetalWidth 2.3 Petal Width PetalWidth # 599 virginica PetalWidth 1.8 Petal Width PetalWidth # # [600 rows x 6 columns]
PySpark では map。
# PySpark def unite(row): return Row(Species=row[2], value=row[3], variable=row[0] + row[1]) sqlContext.createDataFrame(splitted.map(unite)).show() # Species value variable # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.1 SepalLength # setosa 3.5 SepalWidth # .. .. .. # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.0 SepalLength # setosa 3.6 SepalWidth
補足 withColumn の場合、オペレータは 数値の演算として扱われてしまうようなのでここでは使えない。
# PySpark (NG!) ssplitted.withColumn('variable', splitted.Parts + splitted.Scale).show() # Parts Scale Species value variable # Petal Length setosa 1.4 null # Petal Width setosa 0.2 null # .. .. .. .. ..
まとめ
PySpark と pandas のデータ集約/変形処理を整理した。
データ分析用途で利用したい場合、(ごく当たり前だが) データ量が少なく手元でさっといろいろ試したい場合は pandas、データ量が比較的多く 単純な処理を全体にかけたい場合は Spark がよい。
Spark は map 系の API が充実するとさらに使いやすくなりそうだ。が、小回りの効く文法/機能が充実していくことは考えにくいので 完全に Spark だけでデータ分析をする、、という状態には将来もならないのではないかと思う。小さいデータは pandas 使いましょう。

Learning Spark: Lightning-Fast Big Data Analysis
- 作者: Holden Karau,Andy Konwinski,Patrick Wendell,Matei Zaharia
- 出版社/メーカー: O'Reilly Media
- 発売日: 2015/01/28
- メディア: Kindle版
- この商品を含むブログを見る
Python pandas 関連エントリの目次
このブログ中の pandas 関連のエントリをまとめた目次です。
最近 pandas 開発チーム と PyData グループ の末席に加えていただき、パッケージ自体の改善にもより力を入れたいと思います。使い方についてご質問などありましたら Twitter で @ ください。
目次につけた絵文字は以下のような意味です。
- 🔰: 最初に知っておけば一通りの操作ができそうな感じのもの。
- 🚧: v0.16.0 時点で少し情報が古く、機能の改善を反映する必要があるもの。
- 🚫: 当該の機能が deprecate 扱いとなり、将来的に 代替の方法が必要になるもの。
基本
また、上記に対応した比較エントリ:
R {dplyr}, {tidyr}
PySpark
Julia
機能別
データ選択
- Python pandas データ選択処理をちょっと詳しく <前編>
- Python pandas データ選択処理をちょっと詳しく <中編>
- Python pandas データ選択処理をちょっと詳しく <後編>
グルーピング/集約/集計/データ変形
- Python pandas の算術演算 / 集約関数 / 統計関数まとめ
- Python pandas アクセサ / Grouperで少し高度なグルーピング/集計
- Python pandas データのイテレーションと関数適用、pipe
- Python pandas 図でみる データ連結 / 結合処理
- Python pandas 欠損値/外れ値/離散化の処理
そのほかの前処理
データ操作
文字列、日付など、各データ型に固有の操作。
入出力
可視化
- Python pandas プロット機能を使いこなす
- Python Jupyter + pandas で DataFrame 表示をカスタマイズする
- Python spyre によるデータ分析結果のWebアプリ化
地理情報の可視化
その他
- Python pandas のデータを scikit-learn でうまいこと処理したい
- Python pandas で日本の株価情報取得とローソク足チャート描画
- Python pandas パフォーマンス維持のための 3 つの TIPS
- Dask で 並列 DataFrame 処理
変更点
自作パッケージ
- Python pandas / scikit-learn 向けのちょっとしたパッケージ作った <前編>
- Python pandas / scikit-learn 向けのちょっとしたパッケージ作った <後編>
- Python pandas 日本語環境向けのちょっとしたパッケージ作った
発表資料

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (13件) を見る
簡単なデータ操作を PySpark & pandas の DataFrame で行う
Spark v1.3.0 で追加された DataFrame 、結構いいらしいという話は聞いていたのだが 自分で試すことなく時間が過ぎてしまっていた。ようやく PySpark を少し触れたので pandas との比較をまとめておきたい。内容に誤りや よりよい方法があればご指摘 下さい。
過去に基本的なデータ操作について 以下 ふたつの記事を書いたことがあるので、同じ処理のPySpark 版を加えたい。今回は ひとつめの "簡単なデータ操作〜" に相当する内容。
pandas 版
準備
環境は EC2 に作る。Spark のインストールについてはそのへんに情報あるので省略。サンプルデータは iris を csv でダウンロードしてホームディレクトリにおいた。以降の操作は PySpark のコンソールで行う。
# Spark のパスに移動 $ echo $SPARK_HOME /usr/local/spark $ cd $SPARK_HOME $ pwd /usr/local/spark # 既定だとログの出力が邪魔なので抑制 $ cp conf/log4j.properties.template conf/log4j.properties $ vi conf/log4j.properties # INFO をすべて WARN に変える $ bin/pyspark # Welcome to # ____ __ # / __/__ ___ _____/ /__ # _\ \/ _ \/ _ `/ __/ '_/ # /__ / .__/\_,_/_/ /_/\_\ version 1.3.1 # /_/ # # Using Python version 2.7.9 (default, Apr 1 2015 19:28:03) # SparkContext available as sc, HiveContext available as sqlContext.
ここでは HDFS は使わず、pandas の read_csv で pandas.DataFrame を作成する。列名に "." が入っていると PySpark でうまく動かないようだったため以下のカラム名にした。
import pandas as pd # 表示する行数を設定 pd.options.display.max_rows=10 names = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth', 'Species'] # pandas pdf = pd.read_csv('~/iris.csv', header=None, names=names) pdf # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns]
これを PySpark の DataFrame に変換する。pandas と PySpark の基本的な違いとして、
PySparkにはpandas.IndexにあたるものがないPySparkにはpandas.Seriesにあたるものがない ( 代わり? にRowがあるが、どの程度 柔軟な操作ができるかは未知 )
# PySpark sdf = sqlContext.createDataFrame(pdf) sdf # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double, Species: string] # データの中身を表示するには DataFrame.show() sdf.show() # SepalLength SepalWidth PetalLength PetalWidth Species # 5.1 3.5 1.4 0.2 setosa # 4.9 3.0 1.4 0.2 setosa # 4.7 3.2 1.3 0.2 setosa # 4.6 3.1 1.5 0.2 setosa # .. ... ... ... ... # 5.4 3.9 1.3 0.4 setosa # 5.1 3.5 1.4 0.3 setosa # 5.7 3.8 1.7 0.3 setosa # 5.1 3.8 1.5 0.3 setosa
以降、pdf が pandas、sdf が PySpark の DataFrame をあらわす。
type(pdf) # <class 'pandas.core.frame.DataFrame'> type(sdf) # <class 'pyspark.sql.dataframe.DataFrame'>
基本
まず基本的な操作を。先頭いくつかのデータを確認するには head。
PySpark での返り値は Row インスタンスのリストになる。
# pandas pdf.head(5) # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # PySpark sdf.head(5) # [Row(SepalLength=5.1, SepalWidth=3.5, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.9, SepalWidth=3.0, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.7, SepalWidth=3.2, PetalLength=1.3, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.6, SepalWidth=3.1, PetalLength=1.5, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=5.0, SepalWidth=3.6, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa')] # pandas type(pdf.head(5)) # <class 'pandas.core.frame.DataFrame'> # PySpark type(sdf.head(5)) # <type 'list'>
各カラムのデータ型の確認には dtypes。
# pandas pdf.dtypes # SepalLength float64 # SepalWidth float64 # PetalLength float64 # PetalWidth float64 # Species object # dtype: object # PySpark sdf.dtypes # [('SepalLength', 'double'), ('SepalWidth', 'double'), # ('PetalLength', 'double'), ('PetalWidth', 'double'), # ('Species', 'string')]
要約統計量の確認は describe。
# pandas pdf.describe() # SepalLength SepalWidth PetalLength PetalWidth # count 150.000000 150.000000 150.000000 150.000000 # mean 5.843333 3.054000 3.758667 1.198667 # std 0.828066 0.433594 1.764420 0.763161 # min 4.300000 2.000000 1.000000 0.100000 # 25% 5.100000 2.800000 1.600000 0.300000 # 50% 5.800000 3.000000 4.350000 1.300000 # 75% 6.400000 3.300000 5.100000 1.800000 # max 7.900000 4.400000 6.900000 2.500000 # PySpark sdf.describe().show() # summary SepalLength SepalWidth PetalLength PetalWidth # count 150 150 150 150 # mean 5.843333333333334 3.0540000000000003 3.758666666666666 1.1986666666666668 # stddev 0.8253012917851317 0.4321465800705415 1.7585291834055206 0.760612618588172 # min 4.3 2.0 1.0 0.1 # max 7.9 4.4 6.9 2.5
列操作
列名操作
参照と変更。PySpark ではプロパティへの代入は不可。
# pandas pdf.columns # Index([u'SepalLength', u'SepalWidth', u'PetalLength', u'PetalWidth', u'Species'], dtype='object') # PySpark sdf.columns # [u'SepalLength', u'SepalWidth', u'PetalLength', u'PetalWidth', u'Species'] # pandas pdf.columns = [1, 2, 3, 4, 5] pdf.columns # Int64Index([1, 2, 3, 4, 5], dtype='int64') # PySpark sdf.columns = [1, 2, 3, 4, 5] # AttributeError: can't set attribute
マッピングによって列名を変更するには、pandas では DataFrame.rename。
# pandas pdf.columns # Int64Index([1, 2, 3, 4, 5], dtype='int64') pdf = pdf.rename(columns={1:'SepalLength', 2:'SepalWidth', 3:'PetalLength', 4:'PetalWidth', 5:'Species'}) pdf # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns]
PySpark では DataFrame.withColumnRenamed。
# PySpark sdf.withColumnRenamed('Species', 'xxx') # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double, xxx: string]
補足 非破壊的な処理のため、反映には代入が必要。ここでは列名変更したくないので代入しない。
列名による列選択
pandas.DataFrame の列選択では 当該の列のデータを含む Series が返ってくる。
# pandas pdf.Species # 0 setosa # 1 setosa # 2 setosa # 3 setosa # 4 setosa # ... # 145 virginica # 146 virginica # 147 virginica # 148 virginica # 149 virginica # Name: Species, dtype: object pdf['Species'] # 略
PySpark では、列の属性を表現する Column インスタンスが返ってくる。
# PySpark sdf.Species # Column<Species> sdf['Species'] # Column<Species>
pandas、PySpark いずれも、文字列ではなくリストを渡せば その列を DataFrame としてスライシングする。
# pandas pdf[['PetalWidth']] # PetalWidth # 0 0.2 # 1 0.2 # 2 0.2 # 3 0.2 # 4 0.2 # .. ... # 145 2.3 # 146 1.9 # 147 2.0 # 148 2.3 # 149 1.8 # # [150 rows x 1 columns] # PySpark sdf[['PetalWidth']] # DataFrame[PetalWidth: double] # pandas pdf[['PetalWidth', 'PetalLength']] # PetalWidth PetalLength # 0 0.2 1.4 # 1 0.2 1.4 # 2 0.2 1.3 # 3 0.2 1.5 # 4 0.2 1.4 # .. ... ... # 145 2.3 5.2 # 146 1.9 5.0 # 147 2.0 5.2 # 148 2.3 5.4 # 149 1.8 5.1 # # [150 rows x 2 columns] # PySpark sdf[['PetalWidth', 'PetalLength']] # DataFrame[PetalWidth: double, PetalLength: double]
PySpark では DataFrame.select でもよい。
# PySpark sdf.select(sdf['PetalWidth'], sdf['PetalLength']) # DataFrame[PetalWidth: double, PetalLength: double]
真偽値リストによる列選択
自分はあまり使わないのだが、R {dplyr} との比較という観点で。
indexer = [False, False, True, True, False] # pandas pdf.loc[:, indexer] # PetalLength PetalWidth # 0 1.4 0.2 # 1 1.4 0.2 # 2 1.3 0.2 # 3 1.5 0.2 # 4 1.4 0.2 # .. ... ... # 145 5.2 2.3 # 146 5.0 1.9 # 147 5.2 2.0 # 148 5.4 2.3 # 149 5.1 1.8 # # [150 rows x 2 columns] # PySpark sdf[[c for c, i in zip(sdf.columns, indexer) if i is True]] # DataFrame[PetalLength: double, PetalWidth: double]
列の属性による列選択
列の型が pandas の場合は float、PySpark の場合は double の列のみ取り出す。
# pandas pdf.loc[:, pdf.dtypes == float] # SepalLength SepalWidth PetalLength PetalWidth # 0 5.1 3.5 1.4 0.2 # 1 4.9 3.0 1.4 0.2 # 2 4.7 3.2 1.3 0.2 # 3 4.6 3.1 1.5 0.2 # 4 5.0 3.6 1.4 0.2 # .. ... ... ... ... # 145 6.7 3.0 5.2 2.3 # 146 6.3 2.5 5.0 1.9 # 147 6.5 3.0 5.2 2.0 # 148 6.2 3.4 5.4 2.3 # 149 5.9 3.0 5.1 1.8 # # [150 rows x 4 columns] # PySpark sdf[[c for c, type in sdf.dtypes if type == 'double']] # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double]
行操作
値の条件による行選択
# pandas pdf[pdf['Species'] == 'virginica'] # SepalLength SepalWidth PetalLength PetalWidth Species # 100 6.3 3.3 6.0 2.5 virginica # 101 5.8 2.7 5.1 1.9 virginica # 102 7.1 3.0 5.9 2.1 virginica # 103 6.3 2.9 5.6 1.8 virginica # 104 6.5 3.0 5.8 2.2 virginica # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [50 rows x 5 columns] # PySpark sdf[sdf['Species'] == 'virginica'].show() # SepalLength SepalWidth PetalLength PetalWidth Species # 6.3 3.3 6.0 2.5 virginica # 5.8 2.7 5.1 1.9 virginica # 7.1 3.0 5.9 2.1 virginica # 6.3 2.9 5.6 1.8 virginica # .. ... ... ... ... # 6.5 3.0 5.5 1.8 virginica # 7.7 3.8 6.7 2.2 virginica # 7.7 2.6 6.9 2.3 virginica # 6.0 2.2 5.0 1.5 virginica
PySpark では DataFrame.filter でもよい。
# PySpark sdf.filter(sdf['Species'] == 'virginica').show() # 略
行番号による行選択
PySpark では 冒頭の n 行を抽出したりはできるが、適当な行選択ではできない、、、と思う。PySpark でどうしてもやりたければ index にあたる列を追加 -> 列の値で選択。
4/27追記 DataFrame.collect で Row のリストに変換 -> 再度 DataFrame 化すればスキーマ変更せずにできる、、、が、マスタでデータを処理することになるのでよい方法ではなさそう。
# pandas pdf.loc[[2, 3, 4]] # SepalLength SepalWidth PetalLength PetalWidth Species # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # PySpark [c for i, c in enumerate(sdf.collect()) if i in (2, 3 ,4)] # [Row(SepalLength=4.7, SepalWidth=3.2, PetalLength=1.3, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.6, SepalWidth=3.1, PetalLength=1.5, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=5.0, SepalWidth=3.6, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa')] sqlContext.createDataFrame([c for i, c in enumerate(sdf.collect()) if i in (2, 3 ,4)]).show() # SepalLength SepalWidth PetalLength PetalWidth Species # 4.7 3.2 1.3 0.2 setosa # 4.6 3.1 1.5 0.2 setosa # 5.0 3.6 1.4 0.2 setosa
ランダムサンプリング
pandas ではindex をサンプリングしてスライス。
import random # pandas pdf.loc[random.sample(pdf.index, 5)] # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 64 6.1 2.9 4.7 1.4 versicolor # 17 5.4 3.9 1.3 0.4 setosa # 14 4.3 3.0 1.1 0.1 setosa # 4 4.6 3.1 1.5 0.2 setosa # 146 6.7 3.0 5.2 2.3 virginica
PySpark では DataFrame.sample。この関数、fraction の確率で行ごとに選択しているようで、サンプリングするたびに抽出される行数が変わる ( 行数指定でランダムサンプリング、という処理はできなさそう )。
# PySpark sdf.sample(False, 5.0 / sdf.count()).show() # SepalLength SepalWidth PetalLength PetalWidth Species # 5.4 3.4 1.7 0.2 setosa # 5.7 2.8 4.5 1.3 versicolor # 6.2 2.8 4.8 1.8 virginica # 6.1 3.0 4.9 1.8 virginica
補足 今後、pandas にもサンプリングメソッド DataFrame.sample が実装予定 #9666。
列の追加
pandas では DataFrame に対して 直接 新しい列を追加できるが、PySpark ではできない。
# 破壊的な処理になるのでコピー pdf_temp = pdf.copy() # pandas pdf_temp['PetalMult'] = pdf_temp['PetalWidth'] * pdf_temp['PetalLength'] pdf_temp # SepalLength SepalWidth PetalLength PetalWidth Species PetalMult # 0 5.1 3.5 1.4 0.2 setosa 0.28 # 1 4.9 3.0 1.4 0.2 setosa 0.28 # 2 4.7 3.2 1.3 0.2 setosa 0.26 # 3 4.6 3.1 1.5 0.2 setosa 0.30 # 4 5.0 3.6 1.4 0.2 setosa 0.28 # .. ... ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica 11.96 # 146 6.3 2.5 5.0 1.9 virginica 9.50 # 147 6.5 3.0 5.2 2.0 virginica 10.40 # 148 6.2 3.4 5.4 2.3 virginica 12.42 # 149 5.9 3.0 5.1 1.8 virginica 9.18 # # [150 rows x 6 columns] # PySpark sdf['PetalMult'] = sdf['PetalWidth'] * sdf['PetalLength'] # TypeError: 'DataFrame' object does not support item assignment
PySpark での列追加は DataFrame.withColumn。
# PySpark sdf.withColumn('PetalMult', sdf.PetalWidth * sdf.PetalLength).show() # SepalLength SepalWidth PetalLength PetalWidth Species PetalMult # 5.1 3.5 1.4 0.2 setosa 0.27999999999999997 # 4.9 3.0 1.4 0.2 setosa 0.27999999999999997 # 4.7 3.2 1.3 0.2 setosa 0.26 # 4.6 3.1 1.5 0.2 setosa 0.30000000000000004 # .. .. ... ... ... ... # 5.4 3.9 1.3 0.4 setosa 0.52 # 5.1 3.5 1.4 0.3 setosa 0.42 # 5.7 3.8 1.7 0.3 setosa 0.51 # 5.1 3.8 1.5 0.3 setosa 0.44999999999999996
補足 pandas v0.16.0 以降では 類似のメソッド DataFrame.assign が追加。
# pandas pdf.assign(PetalMult=pdf['PetalWidth'] * pdf['PetalLength']) # 略
まとめ
PySpark と pandas のデータ操作を整理した。
PySpark、上のような基本的な処理は pandas と似たやり方で直感的に使える感じだ。
大規模処理は PySpark、細かい取り回しが必要なものは pandas でうまく併用できるとよさそう。
4/29追記 続きはこちら。

Learning Spark: Lightning-Fast Big Data Analysis
- 作者: Holden Karau,Andy Konwinski,Patrick Wendell,Matei Zaharia
- 出版社/メーカー: O'Reilly Media
- 発売日: 2015/01/28
- メディア: Kindle版
- この商品を含むブログを見る
Python Theano function / scan の挙動まとめ
勉強のため たまに Pylearn2 など Theano を使ったパッケージのソースを眺めたりするのだが、theano.scan の挙動を毎回 忘れてしまう。繰り返し調べるのも無駄なので、一回 整理したい。theano.scan の動作は theano.function が前提となるため、あわせて書く。
準備
import numpy as np import theano import theano.tensor as T
theano.function
まずは Theano における関数にあたる Function インスタンスを作成する theano.function の基本的な挙動について。引数はいろいろあるが、特に重要と思われるのは以下の4つ。
inputs:Functionへの入力 (引数) に対応するシンボル。outputs:Function化される式。updates: 共有変数を更新する式。givens: 引数 (inputs) -> 何かへのマッピングを行う辞書。
引数 ひとつを受け取って 1 インクリメントした値を返す Function は以下のように作る。f1(3) の 3 が 定義中のシンボル a に対応する。inputs には、引数がひとつ もしくは ない場合でもリストを指定すること。
a = T.lscalar('a') f1 = theano.function(inputs=[a], outputs=a + 1) f1 # <theano.compile.function_module.Function at 0x10d00fb90> f1(3) # array(4)
補足 theano は内部的に 式をグラフ構造として処理している。その構造は debugprint などで表示できる。
theano.printing.debugprint(f1) # Elemwise{add,no_inplace} [@A] '' 0 # |TensorConstant{1} [@B] # |a [@C]
補足 また、グラフ構造はダイアグラムとしても描画できる。IPython (Jupyter) 上で描画する場合は、
import IPython.display as display def plot(obj): svg = theano.printing.pydotprint(obj, return_image=True, format='svg') return display.SVG(svg) plot(f1)

引数をふたつ受け取りそれらの和を返す Function は以下のようになる。
b = T.lscalar('b') f2 = theano.function(inputs=[a, b], outputs=(a + b)) f2(3, 4) # array(7)
また、各引数の既定値 や 別名は theano.Param を通すことで指定できる。以下の例では、ふたつめの引数 b が指定されない場合、既定値として 1 を使う。
f3 = theano.function(inputs=[a, theano.Param(b, default=1)], outputs=(a + b)) f3(3) # array(4) f3(3, 2) # array(5)
引数をふたつ受け取り、それらの和と差を返す Function。outputs に複数の式をリストで渡せばよい。
f4 = theano.function(inputs=[a, b], outputs=[a + b, a - b]) f4(3, 4) # [array(7), array(-1)]
引数をひとつ受け取り、別に定義した 共有変数との和を返す Function。共有変数は function 内で直接使ってよい。
s = theano.shared(2) s.get_value() # array(2) f5 = theano.function(inputs=[a], outputs=a + s) f5(3) # array(5)
引数 / 返り値はなしで、別に定義した 共有変数を破壊的に 1 増やす Function。共有変数に対する更新は updates で定義。
inc = theano.function(inputs=[], outputs=None, updates={s: s + 1}) s.get_value() # array(2) inc() # [] s.get_value() # array(3)
引数をひとつ受け取り、共有変数との和を返す Function。同時に、共有変数を破壊的に 1 増やす。共有変数に対する更新は updates で定義。updates の処理は outputs の後に行われているようだ。
s.set_value(0) f6 = theano.function(inputs=[a], outputs=a + s, updates={s: s + 1}) f6(2) # array(2) s.get_value() # array(1) f6(2) # array(3)
また、引数はマッピングさせて使うこともできる。以下のように givens を定義すると、式中のシンボル s が 引数 x でマッピングされる。
x = T.lscalar('x') f7 = theano.function(inputs=[a, x], outputs=a + s, givens=[(s, x)]) f7(2, 5) # array(7)
マッピングは定数でもよい。
f8 = theano.function(inputs=[a], outputs=a + s, givens=[(s, 3)]) f8(2) # array(5)
引数が vector ならブロードキャストされる。
v = T.ivector() f9 = theano.function(inputs=[v], outputs=v * 2) f9([1, 1, 1]) # array([2, 2, 2], dtype=int32)
theano.scan
次に、Theano における 繰り返し処理に対応する theano.scan。scan にはおおきく 以下 2 種類の動きがあり、混同するとわけわからなくなる。それぞれ明確な名前が付いているわけではなさそうだが、便宜上 区別したいので 以下ドキュメントの章題をもとに それぞれ Loop / Iteration と書く。
参考 scan – Looping in Theano — Theano 0.7 documentation
Loop: ある関数
fnを、引数に対してn_steps回 適用する。返り値は 長さn_stepsのベクトルとなり、[fn(x), fn(fn(x))...]のような処理になる。Iteration: ある関数
fnを、シーケンス的な引数に対して適用する。返り値は 引数の各要素と同じ長さのベクトルとなり、[fn(x) for x in ...]のような処理になる。
scan がどちらの処理を行うかは 引数をどのように渡すかによって決まる。
fn: Loop / Iteration において適用される式。sequences: シーケンスとして Iteration 処理される引数。繰り返しのたびにシーケンスの 1, 2, 3... 番目の要素が順にfnへ渡る。outputs_info: Loop 処理の初期値となる値。non_sequences: シーケンスでないfnへの引数。繰り返しのたびに同じ値がfnへ渡る。n_steps: 繰り返し処理を行う回数。
このとき、scan が fn に渡す引数は (最大で) 以下の 3 つになる。それぞれ、対応する引数がない場合は省略される (fn に渡される引数の数自体が変わる)。
- シーケンスの要素 (
sequencesが指定されている場合) - 直前の繰り返し処理の結果 (
outputs_infoが指定されている場合) - シーケンスでない引数 =
non_sequencesそのもの (non_sequencesが指定されている場合)
そのため、scan 処理を書く場合の考え方は以下のようになると思う。
- 処理に対して適切な 引数
sequences,outputs_info,non_sequencesが決まる fnに渡される引数が決まる ->fnの具体的な処理が決まる
まずは 単純な Loop 処理。ひとつ目の引数 5 を 2 倍する処理を 3 回繰り返す。結果は [5*2, 5*2*2, 5*2*2*2] のベクトルとなる。
n = T.iscalar('n') result, updates = theano.scan(fn=lambda prior, nonseq: prior * 2, sequences=None, outputs_info=a, non_sequences=a, n_steps=n) sf1 = theano.function(inputs=[a, n], outputs=result, updates=updates) sf1(5, 3) # array([10, 20, 40])
Loop 処理の最後の結果だけが欲しい場合は、theano.function の outputs でベクトル末尾の要素を指定する。
sf2 = theano.function(inputs=[a, n], outputs=result[-1], updates=updates) sf2(5, 3) # array(40, dtype=int32)
ベクトルに対する Loop 処理。シンボルの型と引数が変わるだけ。
v = T.ivector('v') result, updates = theano.scan(fn=lambda prior, nonseq: prior * 2, sequences=None, outputs_info=v, non_sequences=v, n_steps=n) sf3 = theano.function(inputs=[v, n], outputs=result[-1], updates=updates) sf3([1, 2, 3], 3) # array([ 8, 16, 24], dtype=int32)
fn 内で non_sequences のみを利用したときの結果をみる。non_sequencesには 繰り返し回数にかかわらず同じ値が渡ってくるため、ループ回数 n_steps は結果に影響しなくなる。
result, updates = theano.scan(fn=lambda prior, nonseq: nonseq * 2, sequences=None, outputs_info=v, non_sequences=v, n_steps=n) sf4 = theano.function(inputs=[v, n], outputs=result[-1], updates=updates) sf4([1, 2, 3], 100) # array([2, 4, 6], dtype=int32)
続けて Iteration 処理。以下は 渡された引数 5 と同じ長さのベクトル [0, 1, 2, 3, 4] をつくり、各要素に対して 2 を加算する。
outputs = T.as_tensor_variable(np.asarray(0)) result, updates = theano.scan(fn=lambda seq, prior: seq + 2, sequences=T.arange(a), outputs_info=outputs, non_sequences=None) sf5 = theano.function(inputs=[a], outputs=result, updates=updates) sf5(5) # array([2, 3, 4, 5, 6])
定数ではなく直前の値 (prior) を足すと以下のようになる。直前の結果に各要素が加算されるので、結果は [0, 0+1, 1+2, 3+3, 6+4] のベクトル。
outputs = T.as_tensor_variable(np.asarray(0)) result, updates = theano.scan(fn=lambda seq, prior: seq + prior, sequences=T.arange(a), outputs_info=outputs, non_sequences=None) sf6 = theano.function(inputs=[a], outputs=result, updates=updates) sf6(5) # array([ 0, 1, 3, 6, 10])
n_steps を指定した場合は シーケンスがその長さに達した時点で処理が終わる。
outputs = T.as_tensor_variable(np.asarray(0)) result, updates = theano.scan(fn=lambda seq, prior: seq + prior, sequences=T.arange(a), outputs_info=outputs, non_sequences=None, n_steps=3) sf7 = theano.function(inputs=[a], outputs=result, updates=updates) sf7(5) # array([0, 1, 3])
複数のシーケンスに対して処理をする場合は、sequences に対して各シーケンスに対応するシンボルのリストを渡す。
a = T.ivector('a') b = T.ivector('b') result, updates = theano.scan(fn=lambda seq1, seq2: seq1 + seq2, sequences=[a, b], outputs_info=None, non_sequences=None) sf8 = theano.function(inputs=[a, b], outputs=result, updates=updates) sf8([1, 3, 6], [2, 7, 8]) # array([ 3, 10, 14], dtype=int32)
複数の返り値を持たせる場合は theano.function と同様。
result, updates = theano.scan(fn=lambda seq1, seq2: [seq1 + seq2, seq1 - seq2], sequences=[a, b], outputs_info=None, non_sequences=None) sf9 = theano.function(inputs=[a, b], outputs=result, updates=updates) sf9([1, 3, 6], [2, 7, 8]) # [array([ 3, 10, 14], dtype=int32), array([-1, -4, -2], dtype=int32)]
まとめ
theano.function, theano.scan の挙動を整理した。
scan 処理を書く場合は、
- 処理に対して適切な 引数
sequences,outputs_info,non_sequencesが決まる fnに渡される引数が決まる ->fnの具体的な処理が決まる
scan の処理を読み解く場合は、
- まず引数
sequences,outputs_info,non_sequencesを確認し、Loop / Iteration どちらなのかを見分ける fnに何が渡っているかがわかる ->fnの処理を読み解く
5/3追記 @xiangze さんが、scan の条件付き終了 (while) などについてエントリを書かれているので、こちらもご参照ください。
R {ggplot2} で独自の geom を手軽に作りたい
重要 このエントリは {ggplot2} 1.1.0 以前の情報です。v2.0.0 以降の方法は vignettes "Extending ggplot2" を読んでください。
はじめに
{ggplot2} を使っていると、新しい描画図形 (geom) を作りたいなという場合がたまにある。その方法は {ggplot2} の Wiki に書いてあり、手順は、
- 描画図形の実体を
grid::Grobとして定義する (リンク先の例ではfieldGrob) - 定義した
Grobを 呼び出す描画用クラスをggplot2:::Geomを継承して作る (リンク先の例ではGeomField) - 描画用関数
geom_xxxを作る (リンク先の例ではgeom_field)
手順のうち 新しい Grob を作るのは少し面倒な感じだ。が、作りたい geom が 既存 {ggplot2} 関数へのちょっとした追加処理や 組み合わせで表現できる場合には新しい Grob を作る必要もなく、わりと手軽にできる。ここではその方法を書く。
やりたいこと
ggplot2::geom_ribbon を階段状に描画したい。背景はこちら。
@berobero11 そのステキ関数の存在を知りませんでした。信頼区間の塗りつぶしが一手間必要そうなので、少し時間ください。
— sinhrks (@sinhrks) 2015, 3月 241. 既存の描画用クラス (Geom) を継承して作る
ribbon を階段状に描画するには、geom_ribbon の描画直前に データを階段状に変換する必要がある。他は geom_ribbon 同様に動けばよいので、描画用のクラスは ggplot2:::Geom ではなく ggplot2:::GeomRibbon を継承してつくればよさそうだ。
補足 データを階段状に変更する処理は ggplot2 の geom-path-step.R のものを多少変更して作成。
library(ggplot2) library(proto) # 描画用クラスを定義 GeomConfint <- proto(ggplot2:::GeomRibbon, { objname <- "confint1" required_aes <- c("x", "ymin", "ymax") draw <- function(., data, scales, coordinates, na.rm = FALSE, ...) { if (na.rm) data <- data[complete.cases(data[required_aes]), ] data <- data[order(data$group, data$x), ] # ここでデータを階段状に変換 data <- .$stairstep_confint(data) ggplot2:::GeomRibbon$draw(data, scales, coordinates, na.rm = FALSE, ...) } stairstep_confint <- function (., data) { # データを階段状に変換するメソッド data <- as.data.frame(data)[order(data$x), ] n <- nrow(data) ys <- rep(1:n, each = 2)[-2 * n] xs <- c(1, rep(2:n, each = 2)) data.frame(x = data$x[xs], ymin = data$ymin[ys], ymax = data$ymax[ys], data[xs, setdiff(names(data), c("x", "ymin", "ymax"))]) } }) # 描画用クラスを呼び出す描画関数を定義 geom_confint1 <- function (mapping = NULL, data = NULL, stat = "identity", position = "identity", na.rm = FALSE, ...) { GeomConfint$new(mapping = mapping, data = data, stat = stat, position = position, na.rm = na.rm, ...) }
このとき、GeomConfint$draw の data としては、以下のように描画用に変換された data.frame が渡される。そのため、描画用データに対して処理を行う場合はこの方法が便利。
# x ymin ymax PANEL group colour fill size linetype alpha # 1 1 -1 1 1 1 NA grey20 0.5 1 0.5 # 2 2 -2 2 1 1 NA grey20 0.5 1 0.5 # 3 3 -3 3 1 1 NA grey20 0.5 1 0.5 # 4 4 -4 4 1 1 NA grey20 0.5 1 0.5
描画してみる。上で定義した描画関数 geom_confint1 がそのまま geom として使える。
df <- data.frame(x = c(1, 2, 3, 4), upper = c(1, 2, 3, 4), lower = c(-1, -2, -3, -4)) ggplot(data = df) + geom_confint1(aes(x = x, ymin = lower, ymax = upper), alpha = 0.5)

2. 描画用関数 geom_xxx だけを定義して作る
また、今回の場合 塗りつぶしが必要なければ 複数の geom_step の組み合わせでも描ける。このときは描画関数を以下のように定義すればよい。
geom_confint2 <- function (mapping = NULL, data = NULL, stat = "identity", position = "identity", na.rm = FALSE, ...) { # 上側 / 下側の階段関数 geom_step に渡す mapping を作成 mapping1 <- mapping mapping1['y'] <- mapping['ymax'] mapping2 <- mapping mapping2['y'] <- mapping['ymin'] g1 <- geom_step(mapping = mapping1, data = data, stat = stat, position = position, na.rm = na.rm, ...) g2 <- geom_step(mapping = mapping2, data = data, stat = stat, position = position, na.rm = na.rm, ...) # 複数の geom はリストで返す list(g1, g2) }
描画する。
ggplot(data = df) + geom_confint2(aes(x = x, ymin = lower, ymax = upper))

補足 ただし、上の例では mapping の変換を伴うため、以下の書式では動かない。
ggplot(data = df, mapping = aes(x = x, ymin = lower, ymax = upper)) + geom_confint2() # Error: 引数に異なる列数のデータフレームが含まれています: 7, 0
まとめ
独自の geom は正当な方法以外でもわりと手軽に作れる。
- 既存の描画用クラス (
Geom) を継承して作る - 描画用関数
geom_xxxだけを定義して作る
Python pandas / scikit-learn 向けのちょっとしたパッケージ作った <前編>
こちらの続き。
pandas のデータを scikit-learn でうまく処理するためのパッケージを作ったのでその使い方を書きたい。今回は 適当なデータをファイルから読み込み -> 前処理してクラスタリングする、という例を書く。
このパッケージ 基本的には pandas と使い方は同じなので、以下 追加機能にあたる部分を中心に記載する。
- データは 説明変数 / 目的変数の定義をもち、それぞれプロパティから参照 / 上書きできる。
scikit-learnの各サブパッケージにプロパティからアクセスできる。また、メソッド呼び出し時の引数を省略できる。
インストール
pip install pandas_ml
補足 今後 別パッケージへの拡張を考えているため、scikit-learn への依存は optional にしている。そのため、scikit-learn をインストールしていない方は別途インストールを。
データの読み込み
こういう例を書くとき、 iris 以外のデータがぱっと出てくる人ってかっこいいと思う。
# おまじない import numpy as np import pandas as pd pd.options.display.max_rows = 8 state = np.random.RandomState(1) # csv からデータ読み込み # http://aima.cs.berkeley.edu/data/iris.csv names = ['Sepal.Length', 'Sepal.Width', 'Petal.Length', 'Petal.Width', 'Species'] iris = pd.read_csv('iris.csv', header=None, names=names) iris # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # .. ... ... ... ... ... # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns] type(iris) # <class 'pandas.core.frame.DataFrame'>
上で読み込んだデータを pandas_ml で処理するには、一度 pandas_ml.ModelFrame インスタンスに変換する必要がある。ModelFrame を作成するには、
- 第一引数には、
pd.DataFrameに変換できる何か (DataFrame自体, 辞書など) を渡す targetキーワードには、目的変数として扱いたい 列名の文字列、もしくはpd.Seriesに変換できる何か (Series、もしくはリスト-like )を渡す
詳しくは ドキュメント を。
今回は 第一引数には pd.DataFrame、 target には 目的変数として扱いたい列名である "Species" を指定する。
import pandas_ml as pdml df = pdml.ModelFrame(iris, target='Species') type(df) # <class 'pandas_ml.core.frame.ModelFrame'> # ModelFrame は DataFrame のサブクラス isinstance(df, pd.DataFrame) # True
補足 target キーワードを指定しない場合、目的変数をもたない ModelFrame インスタンスが作成される。
作成した ModelFrame は pd.DataFrame と同じように扱える。一部、 pd.DataFrame にないメソッド / プロパティを持つ。
df # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # .. ... ... ... ... ... # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns] # データが目的変数を持っているかを確認 df.has_target() # True # 説明変数は data プロパティで参照 df.data # Sepal.Length Sepal.Width Petal.Length Petal.Width # 0 5.1 3.5 1.4 0.2 # 1 4.9 3.0 1.4 0.2 # 2 4.7 3.2 1.3 0.2 # 3 4.6 3.1 1.5 0.2 # .. ... ... ... ... # 146 6.3 2.5 5.0 1.9 # 147 6.5 3.0 5.2 2.0 # 148 6.2 3.4 5.4 2.3 # 149 5.9 3.0 5.1 1.8 # # [150 rows x 4 columns] # 目的変数は target プロパティで参照 df.target # 0 setosa # 1 setosa # ... # 148 virginica # 149 virginica # Name: Species, Length: 150, dtype: object # 目的変数の列名を取得 df.target_name # 'Species' # これも一緒 (target の name 属性を見ているので) df.target.name # 'Species'
このとき、ModelFrame.data は 目的変数のない ModelFrame インスタンスに、また ModelFrame.target は pd.Series を継承した ModelSeries インスタンスになっている。
type(df.data) # <class 'pandas_ml.core.frame.ModelFrame'> df.data.has_target() # False type(df.target) # <class 'pandas_ml.core.series.ModelSeries'> isinstance(df.target, pd.Series) # True
データの前処理
いくつか前処理を行う。まず、target についているラベル ( "setosa", "versicolor", "virginica" ) を数値 (0, 1, 2) に変換する。この処理は pandas の map がカンタン。
df.target.map({'setosa': 0, 'versicolor': 1, 'virginica': 2}) # 0 0 # 1 0 # ... # 148 2 # 149 2 # Name: Species, Length: 150, dtype: int64 # これをそのまま target に代入すればよい df.target = df.target.map({'setosa': 0, 'versicolor': 1, 'virginica': 2}) df # Species Sepal.Length Sepal.Width Petal.Length Petal.Width # 0 0 5.1 3.5 1.4 0.2 # 1 0 4.9 3.0 1.4 0.2 # 2 0 4.7 3.2 1.3 0.2 # 3 0 4.6 3.1 1.5 0.2 # .. ... ... ... ... ... # 146 2 6.3 2.5 5.0 1.9 # 147 2 6.5 3.0 5.2 2.0 # 148 2 6.2 3.4 5.4 2.3 # 149 2 5.9 3.0 5.1 1.8 # # [150 rows x 5 columns]
次に、(特に理由はないが) "Sepal.Length" と "Sepal.Width" のみを正規化する。これは pandas にはない処理なので、 sklearn.preprocessing.normalize を使う。
ModelFrame は scikit-learn の各サブパッケージに対応したプロパティ (アクセサ) を持っており、サブパッケージをロードしなくても 対応する関数をメソッドとして利用できる。このとき、呼び出し元のデータが自動的に引数として渡されるため データの引数指定は不要。
サブパッケージとプロパティの対応一覧はドキュメント を。
df[['Sepal.Length', 'Sepal.Width']].preprocessing.normalize() # Sepal.Length Sepal.Width # 0 0.824513 0.565842 # 1 0.852851 0.522154 # 2 0.826599 0.562791 # 3 0.829266 0.558853 # .. ... ... # 146 0.929491 0.368846 # 147 0.907959 0.419058 # 148 0.876812 0.480833 # 149 0.891385 0.453247 # # [150 rows x 2 columns] # これもそのまま代入すればよい df[['Sepal.Length', 'Sepal.Width']] = df[['Sepal.Length', 'Sepal.Width']].preprocessing.normalize() df # Species Sepal.Length Sepal.Width Petal.Length Petal.Width # 0 0 0.824513 0.565842 1.4 0.2 # 1 0 0.852851 0.522154 1.4 0.2 # 2 0 0.826599 0.562791 1.3 0.2 # 3 0 0.829266 0.558853 1.5 0.2 # .. ... ... ... ... ... # 146 2 0.929491 0.368846 5.0 1.9 # 147 2 0.907959 0.419058 5.2 2.0 # 148 2 0.876812 0.480833 5.4 2.3 # 149 2 0.891385 0.453247 5.1 1.8 # # [150 rows x 5 columns]
クラスタリング
ModelFrame.cluster.k_means で、sklearn.cluster.k_means を呼び出せる。このメソッドの返り値は centroid, label, inertia の 3つになる。
centroid, label, inertia = df.cluster.k_means(n_clusters=3, random_state=state) centroid # array([[ 0.82602947, 0.56251635, 1.464 , 0.244 ], # [ 0.91055414, 0.41171559, 5.59583333, 2.0375 ], # [ 0.90545279, 0.42259059, 4.26923077, 1.34230769]]) label # 0 0 # 1 0 # ... # 148 1 # 149 1 # Length: 150, dtype: int32 inertia # 31.598367459843072
もしくは、ModelFrame.cluster.KMeans から sklearn.cluster.KMeans インスタンスを作成して fit -> predict する。メソッドの呼び出しを ModelFrame 側から行うことで引数を省略できる。
kmeans = df.cluster.KMeans(n_clusters=3, random_state=state) df.fit(kmeans) # KMeans(copy_x=True, init='k-means++', max_iter=300, n_clusters=3, n_init=10, # n_jobs=1, precompute_distances=True, # random_state=<mtrand.RandomState object at 0x104053410>, tol=0.0001, # verbose=0) df.predict(kmeans) # 0 1 # 1 1 # ... # 148 0 # 149 0 # Length: 150, dtype: int32
直前に利用された estimator は ModelFrame.estimator から参照できる。また、直近のクラスタリング結果は ModelFrame.predicted にキャッシュされている。
df.estimator # KMeans(copy_x=True, init='k-means++', max_iter=300, n_clusters=3, n_init=10, # n_jobs=1, precompute_distances=True, # random_state=<mtrand.RandomState object at 0x104053410>, tol=0.0001, # verbose=0) df.predicted # 0 1 # 1 1 # ... # 148 0 # 149 0 # Length: 150, dtype: int32
ModelFrame.metrics はsklearn.metrics の各メソッドを引数省略して呼び出せる。このとき、内部的には上のキャッシュが利用されている。
df.metrics.completeness_score()
# 0.8643954288752761
引数省略時の内部処理について
scikit-learn の各メソッドと 内部的に渡すデータの対応関係一覧は以下ドキュメントに記載している。何かおかしなことをやっていたら GitHub からご指摘ください。
まとめ
pandas_ml を利用して データの読み込み / 前処理 / クラスタリングを行う方法を記載した。
次は、Pipeline, Cross Validation, Grid Search あたりを。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (10件) を見る
9/12追記 パッケージ名変更。