R {ggplot2} で 少し複雑なサブプロットが描きたい
この記事は R Advent Calendar 2015 4 日目の記事です。
{ggplot2} でのサブプロット
{ggplot2} でサブプロットを描画したいことがある。同じ種類のプロットを水準別に描画するなど、単純なものであれば facet で描ける。例えば 適当な散布図を Species 別に描きたい場合、
library(dplyr) library(ggplot2) ggplot(iris, aes(Sepal.Width, Sepal.Length)) + geom_point() + facet_wrap(~ Species)

facet では異なる種類のプロットをサブプロットとすることはできない。例えば散布図の縦軸/横軸をそれぞれ変えたい、といった場合には gridExtra::grid.arrange を使う。
library(gridExtra) p1 <- ggplot(iris, aes(Sepal.Width, Sepal.Length)) + geom_point() p2 <- ggplot(iris, aes(Petal.Width, Petal.Length)) + geom_point() p3 <- ggplot(iris, aes(Sepal.Length, Petal.Length)) + geom_point() p4 <- ggplot(iris, aes(Sepal.Width, Petal.Width)) + geom_point() grid.arrange(p1, p2, p3, p4)

が、grid.arrange は引数を ドット ... で受け取ること、また呼び出すと直ちに描画を行うことから、以下のような場合は少し面倒だ。
- 描画が直ちに行われるため、サブプロットの描画設定をまとめて変更できない。単一のテーマを使う場合でも、個々のプロットそれぞれについてあらかじめ処理を行っておく必要がある。
- リストが渡せない。サブプロットの数を動的に変更したい場合は、リストに入れて
do.callが必要である。
というわけで、自作パッケージ {ggfortify} でこの辺の操作を少し便利にできるようにした。サブプロット周りの処理は ggmultiplot クラスで行う。
コンストラクタの引数にはリストが渡せるため、サブプロット数が変わる場合でも楽だと思う。また、インスタンスに対して + 演算子を用いることで ggplot と同じ処理を各プロットに対して適用できる。
# install.packages('ggfortify') library(ggfortify) p <- new('ggmultiplot', plots = list(p1, p2, p3, p4)) p # 略。上記 grid.arrange と同じ出力 # theme_bw を全サブプロットに適用 p + theme_bw()

サブプロットの要素には リストと同じようにアクセスできる。[ でのアクセスは ggmultiplot インスタンスに、[[ での アクセスは ggplot インスタンスになる。代入もできるため、一部のプロットにだけ処理を適用したい場合は以下のように書ける。
p[2:3] <- p[2:3] + stat_smooth(method = 'lm') p
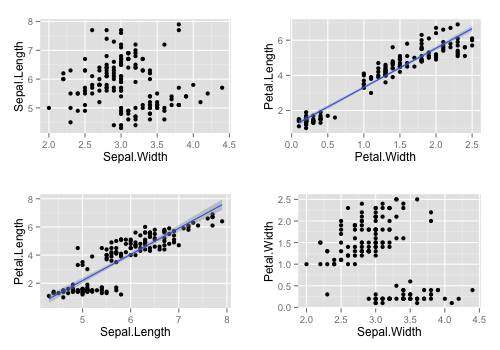
+ 演算子の右項に ggplot インスタンスを指定するとサブプロットが追加できる。
p + (ggplot(iris, aes(Sepal.Width, fill = Species)) + geom_density())
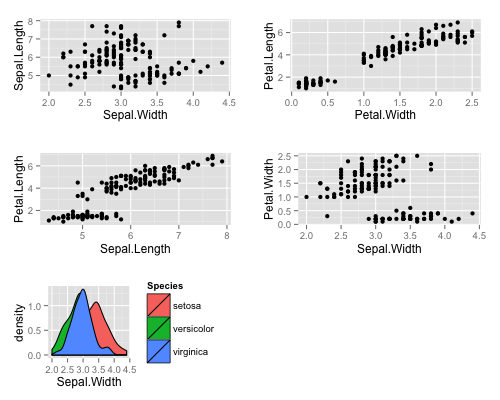
ggmultiplot 作成時に ncol もしくは nrow キーワードを指定することで、サブプロットの列数 / 行数が指定できる。
p <- new('ggmultiplot', plots = list(p1, p2, p3, p4), ncol = 3) p

autoplot との組み合わせ
{ggfortify} を使うと主要なクラスを ggplot2::autoplot 関数で描画できるようになる。autoplot にリストを渡した場合は、リストの各要素が autoplot 可能であればそれらをサブプロットとして描画する。
サンプルとして、{broom} パッケージの vignettes にある例をやってみたい。ここでは、適当なデータに対して クラスタ数を変えながら kmeans クラスタリングを行った結果をプロットしている。
これは autoplot を使って以下のように書ける。
kclusts <- data.frame(k=1:9) %>% dplyr::group_by(k) %>% dplyr::do(kclust = kmeans(iris[-5], .$k)) autoplot(kclusts$kclust, data = iris[-5], ncol = 3) + theme(legend.position = "none")
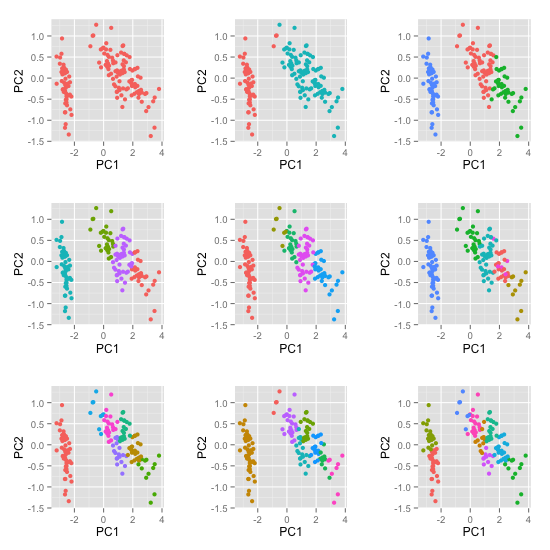
一行目は今なら {purrr} を使ったほうが簡潔でわかりやすくなると思う。
library(purrr) kclusts <- purrr::map(1:9, ~ kmeans(iris[-5], .)) autoplot(kclusts, data = iris[-5], ncol = 3) + theme(legend.position = "none") # 略
まとめ
{ggfortify} では ggmultiplot クラス、ならびに autoplot を利用してサブプロットを簡単に扱うことができる。

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
R パッケージを CRAN で公開する
少し前に 自作パッケージを CRAN で公開したのだが ブログに書くのを忘れていた。CRAN 公開時の注意点に関して、日本語の説明があまりない / 情報が古いので簡単にまとめたい。
パッケージの作成
この資料を読みましょう。
www.slideshare.net
継続的インテグレーション (CI)
Travis CI は R をサポート (community supportだが) しているため、.travis.yml に2行記載するだけで利用できる。CI 上でパッケージのチェック (R CMD check) も走るので利用したほうが楽。
複数の環境でテストを実行したい場合、Travis CI では Build Matrix という仕組みがある。R では実行したいテストの数だけ env: タグ中に環境変数を記載するのがよいと思う。自分のパッケージでは {ggplot2} の公式版 / GitHub 上の開発版で 2 つのテストを実行している。
そのほかの CI ツール
ほか、R から CI を利用するツールとして、以下のようなものもある。
- r-appveyor: Windows 用の CI 環境である AppVeyor が利用できる。
- r-builder: Travis CI を R から利用する。R-devel が利用できる。
vignettes の作成
11/30追記 {knitr} で作るのがカンタン。vignettes 化したい {knitr}ドキュメント中に以下のようなタグを含めればよい。VignetteIndexEntry は vignettes へのリンクテキストとして利用される。ASCII文字以外を含める場合、DESCRIPTION ファイル中で Encoding: UTF-8 を指定する。
<!--
%\VignetteEngine{knitr::knitr}
%\VignetteIndexEntry{Introduction to ggfortify package}
-->
参考 Package vignettes: How to build package vignettes with knitr | knitr
パッケージのチェック
開発版 (R-devel) の日次ビルド を利用して、R CMD Check --as-cran package_name.tar.gz で行う。開発版のほうが警告が厳しく出るようだ。
自分は 開発版環境を AWS 上に構築している。 Amazon Linux であれば以下のスクリプトでインストールできると思う。
R CMD check では ERROR, WARNING はもちろん、NOTE も "checking CRAN incoming feasibility" 1 つを除いて全て表示されないようにするのが望ましい。
特に、{dplyr} 等で NSE を使っている場合は xxx: no visible binding for global variable 'xxx' といった NOTE が表示されるが、これらについても CRAN 管理者から指摘される場合がある。 SE を使うか、関数中に (ダミーとして) 同名の変数を作るなどで回避しておくのがよい。
パッケージのサブミット
FTP で直接あげてはダメ。Submit package to CRAN に必要事項を記載して Upload package をする。その後、確認のメールが来たら Confirmation する。
公開待ちのパッケージは 以下の FTP サーバにて確認できる。
パッケージによっては 拡張子の後に以下のようなステータス情報が付けられる場合がある。これは CRAN 公式の情報ではなく、いくつかのフォーラム文書をみて自分が推測したものなので間違っているかもしれない。
.save:UploadされたがConfirmationされていないもの。.pending: CRAN 管理者にて何らかの理由でペンディングされているもの (放っておくしかない)。.noemail:Confirmationはされているが確認時のメールが CRAN に飛んでいないもの。
参考 What are the steps in submitting an R-package to CRAN and how long does each step take? - Stack Overflow (ほぼ情報ない)
その後の対応
CRAN 管理者から指摘がきたら適切に対応してください。
パッケージの公開
11/30追記 CRAN 上でのテストとレビューに問題がなければ CRAN 公開後にメールがくる。その後 1日程度すると Windows 用のバイナリのビルドが自動的に行われ、改めて通知がくる。
R {ggplot2} の散布図に凸包 / 確率楕円を描きたい
小ネタ。{ggplot2} でグループ別の散布図を描くときに、ちょっと飾り付けをしてグループをわかりやすくしたい。
凸包 (Convex)
最初にベースとなる散布図を描く。
library(dplyr) library(ggplot2) df <- iris p <- ggplot(df, aes(x = Petal.Width, y = Petal.Length)) + geom_point() p

まずは 散布図全体について凸包をとる。ある点の集合の凸包は、 grDevices::chull で計算できる。chull は凸な点の index を返すので、この返り値に含まれるデータのみをフィルタして geom_polygon に渡せばよい。
chull(df[c('Petal.Width', 'Petal.Length')]) # [1] 44 17 23 14 33 25 135 123 119 110 145 115 hulls <- df[chull(df[c('Petal.Width', 'Petal.Length')]), ] hulls # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 44 5.0 3.5 1.6 0.6 setosa # 17 5.4 3.9 1.3 0.4 setosa # 23 4.6 3.6 1.0 0.2 setosa # 14 4.3 3.0 1.1 0.1 setosa # 33 5.2 4.1 1.5 0.1 setosa # 25 4.8 3.4 1.9 0.2 setosa # 135 6.1 2.6 5.6 1.4 virginica # 123 7.7 2.8 6.7 2.0 virginica # 119 7.7 2.6 6.9 2.3 virginica # 110 7.2 3.6 6.1 2.5 virginica # 145 6.7 3.3 5.7 2.5 virginica # 115 5.8 2.8 5.1 2.4 virginica p + geom_polygon(data = hulls, alpha = 0.2)

凸包をグループ別に描画したい場合は、{dplyr} で各グループへ chull を適用する。
p <- ggplot(df, aes(x = Petal.Width, y = Petal.Length, colour = Species)) + geom_point() hulls <- df %>% dplyr::group_by(Species) %>% dplyr::do(.[chull(.[c('Petal.Width', 'Petal.Length')]), ]) hulls # Source: local data frame [22 x 5] # Groups: Species # # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 1 5.4 3.9 1.3 0.4 setosa # 2 4.6 3.6 1.0 0.2 setosa # 3 4.3 3.0 1.1 0.1 setosa # 4 5.2 4.1 1.5 0.1 setosa # 5 4.8 3.4 1.9 0.2 setosa # 6 5.1 3.8 1.9 0.4 setosa # 7 5.0 3.5 1.6 0.6 setosa # 8 5.1 2.5 3.0 1.1 versicolor # 9 4.9 2.4 3.3 1.0 versicolor # 10 5.8 2.7 4.1 1.0 versicolor # 11 6.1 2.8 4.7 1.2 versicolor # 12 6.0 2.7 5.1 1.6 versicolor # 13 6.7 3.0 5.0 1.7 versicolor # 14 5.9 3.2 4.8 1.8 versicolor # 15 5.8 2.8 5.1 2.4 virginica # 16 4.9 2.5 4.5 1.7 virginica # 17 6.0 2.2 5.0 1.5 virginica # 18 6.1 2.6 5.6 1.4 virginica # 19 7.7 2.8 6.7 2.0 virginica # 20 7.7 2.6 6.9 2.3 virginica # 21 7.2 3.6 6.1 2.5 virginica # 22 6.7 3.3 5.7 2.5 virginica p + geom_polygon(data = hulls, alpha = 0.2)
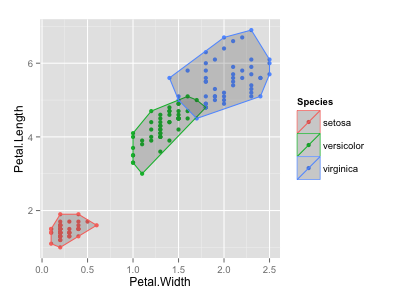
参考 以下リンクにある plyr::ddply を使うバージョンを参考にした。
確率楕円 (Probability Ellipse)
こっちは ggplot2 1.0.0 以降なら stat_ellipse 一発なので簡単。
p + stat_ellipse()

クラスタリング結果への凸包 / 確率楕円の描画
もともとやりたかったのは {ggplot2} + {ggfortify} でクラスタリング結果を autoplot するときに凸包 / 確率楕円を描画すること。これを autoplot 時の frame オプションで指定できるようにした。
ついでに {cluster} パッケージの非階層クラスタリング法 cluster::clara, cluster::fanny, cluster::pam も autoplot できるようにした。
# library(devtools) # install_github('sinhrks/ggfortify') library(ggplot2) library(ggfortify) df <- iris[-5] autoplot(kmeans(df, 3), original = iris, frame = TRUE)
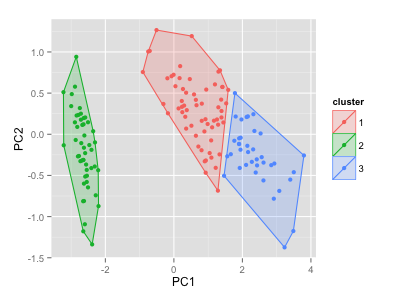
library(cluster) autoplot(clara(df, 3), frame = TRUE, frame.type = 'norm')
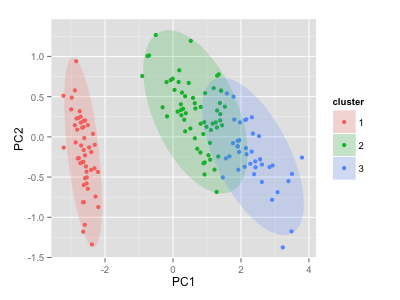
ほか、細かい使い方はこちら。
クラスタリング以外の autoplot についてはこちら。

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
R ggplot2 で線形回帰/一般化線形モデルの残差プロット
ggplot2 で出力している他のグラフとデザインを揃えたかったので作ってみた。
インストール
library(devtools) install_github('sinhrks/ggfortify')
サンプル
デフォルトの plot と (ほぼ) 同じ見た目にしたつもり。
library(ggplot2) library(ggfortify) m <- lm(Petal.Width ~ Petal.Length, data = iris) autoplot(m, which = 1:6)
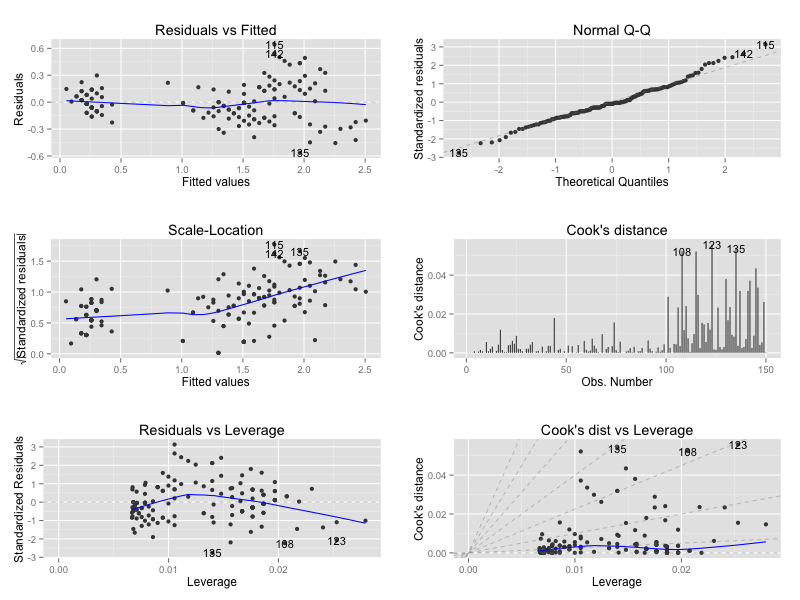
通常の残差プロット
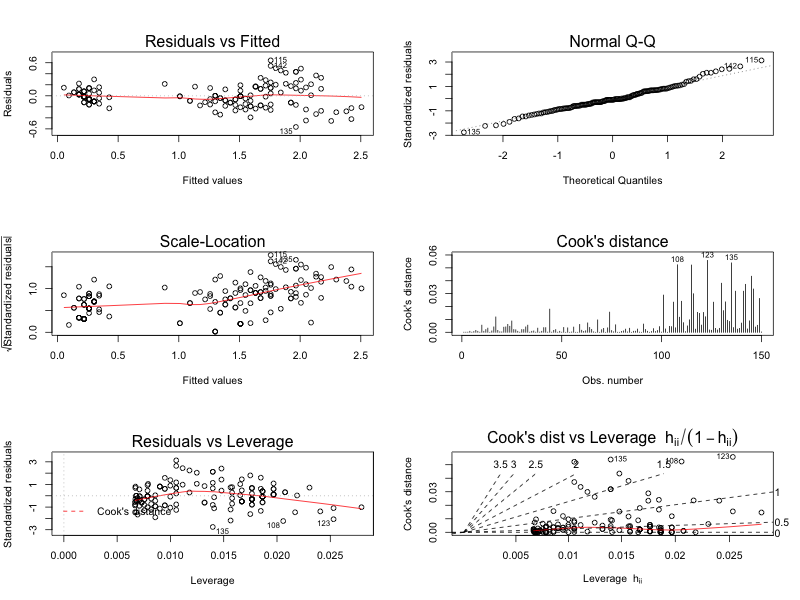
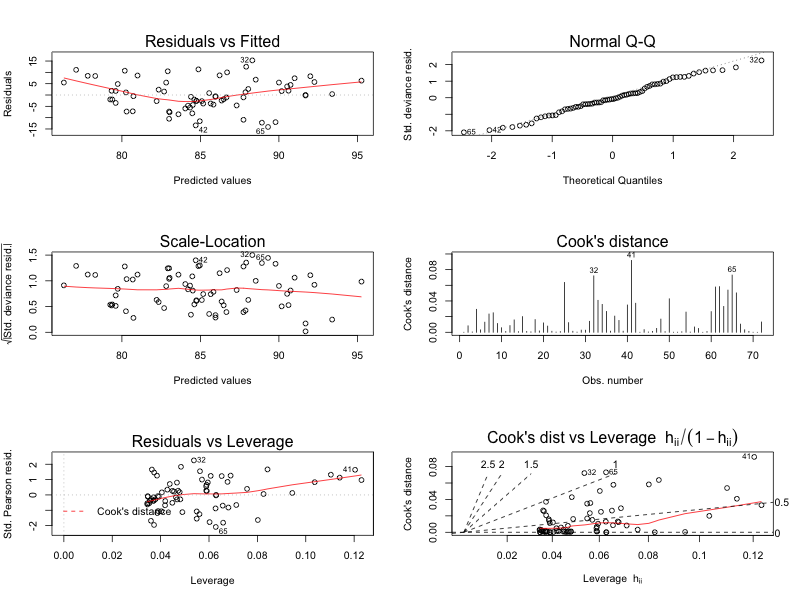
一般化線形モデル
data(anorexia, package = "MASS") m <- glm(Postwt ~ Prewt + Treat + offset(Prewt), family = gaussian, data = anorexia) autoplot(m, which = 1:6)
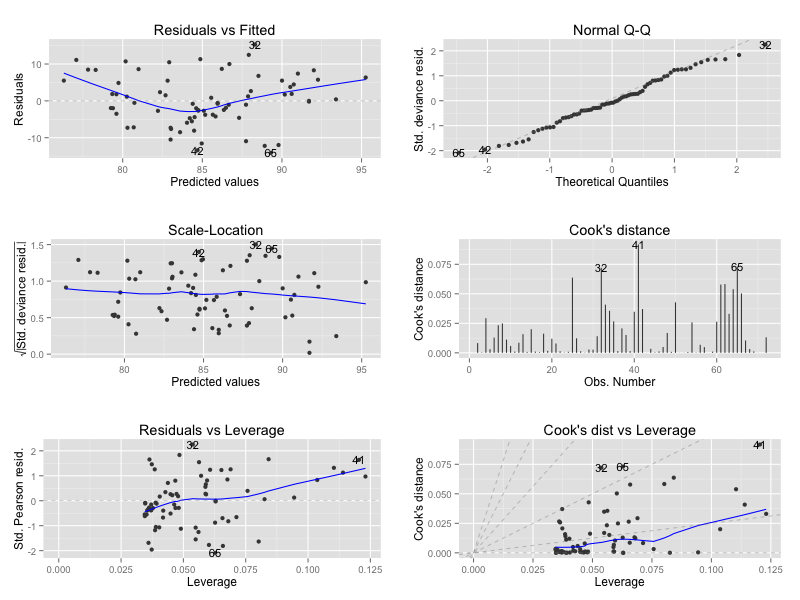
通常の残差プロット
設定できるオプション
which: 描画対象のグラフ (1〜6まで、通常のplotと同じ)colour: データの色label.colour: ラベルの色label.size: ラベルの文字サイズlabel.n: ラベルを表示する数smooth.colour: 平滑線の色smooth.linetype: 平滑線のスタイルad.colour: 補助線の色ad.linetype: 補助線のスタイルad.size: 補助線のサイズnrow: 複数プロットする際の行数ncol: 複数プロットする際の列数

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
R ggplot2 で monthplot
Rで時系列データの周期特性をちゃっと確認したいとき、monthplotという関数を使うとデータを月次に分割してグラフ作成してくれる。
monthplot(AirPassengers)
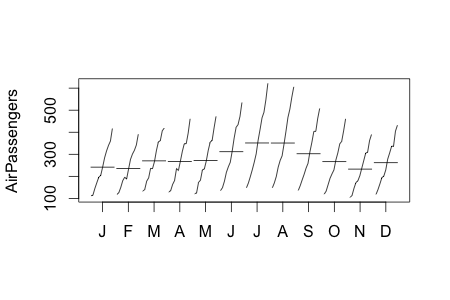
が、出力がちょっとアレなのと 月次以外の周期性をみたい場合があるので、 ggplot2 で作ってみた。
library(devtools) install_github('sinhrks/ggfortify') library(ggfortify) ggfreqplot(AirPassengers)
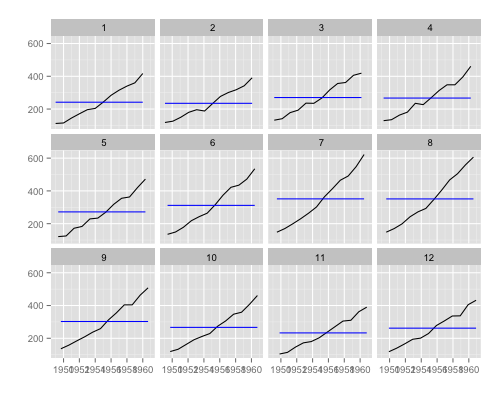
6ヶ月ごとの周期をみたいとき。
ggfreqplot(AirPassengers, freq = 6)
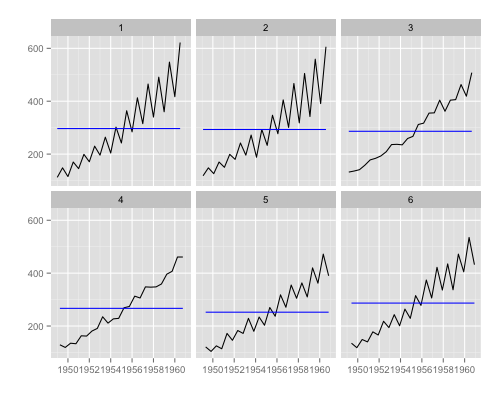
RPubs - Plotting Time Series Statistics with ggplot2 and ggfortify

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
ggplot2でよく使うパッケージを autoplot する
パッケージを書いた。
つかいかた
- RPubs - Plotting Time Series with ggplot2 and ggfortify
- RPubs - Plotting Time Series Statistics with ggplot2 and ggfortify
- RPubs - Plotting PCA/clustering results using ggplot2 and ggfortify
- RPubs - Plotting Survival Curves using ggplot2 and ggfortify
- RPubs - Plotting Probability Distributions with ggplot2 and ggfortify

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
ggplot2でsurvival::survfitさせた生存曲線を描画する
こちらの survival 版: ggplot2でforecastインスタンスを描画する - StatsFragments
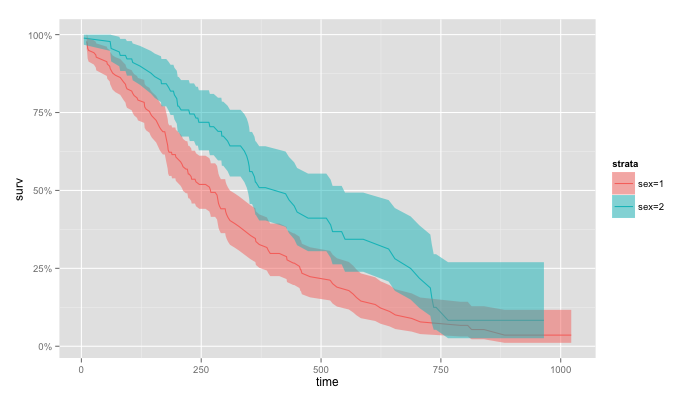
同じように survfit 用の fortify を定義すればよい。lung(肺ガンデータ)を使って、男女別のKaplan-Meier曲線を描いてみる。
library(survival)
library(ggplot2)
library(scales)
d.survfit <- survival::survfit(survival::Surv(time, status) ~ sex,
data = lung)
fortify.survfit <- function(survfit.data) {
data.frame(time = survfit.data$time,
n.risk = survfit.data$n.risk,
n.event = survfit.data$n.event,
n.censor = survfit.data$n.censor,
surv = survfit.data$surv,
std.err = survfit.data$std.err,
upper = survfit.data$upper,
lower = survfit.data$lower,
strata = rep(names(survfit.data$strata), survfit.data$strata))
}
head(ggplot2::fortify(d.survfit))
# time n.risk n.event n.censor surv std.err upper lower strata
# 1 11 138 3 0 0.9782609 0.01268978 1.0000000 0.9542301 sex=1
# 2 12 135 1 0 0.9710145 0.01470747 0.9994124 0.9434235 sex=1
# 3 13 134 2 0 0.9565217 0.01814885 0.9911586 0.9230952 sex=1
# 4 15 132 1 0 0.9492754 0.01967768 0.9866017 0.9133612 sex=1
# 5 26 131 1 0 0.9420290 0.02111708 0.9818365 0.9038355 sex=1
# 6 30 130 1 0 0.9347826 0.02248469 0.9768989 0.8944820 sex=1
y軸はパーセント表示したいので、scale_y_continuous で設定する。
ggplot(data = d.survfit) + geom_line(aes_string(x = 'time', y = 'surv', colour = 'strata')) + geom_ribbon(aes_string(x = 'time', ymin = 'lower', ymax = 'upper', fill = 'strata'), alpha = 0.5) + scale_y_continuous(labels = scales::percent)
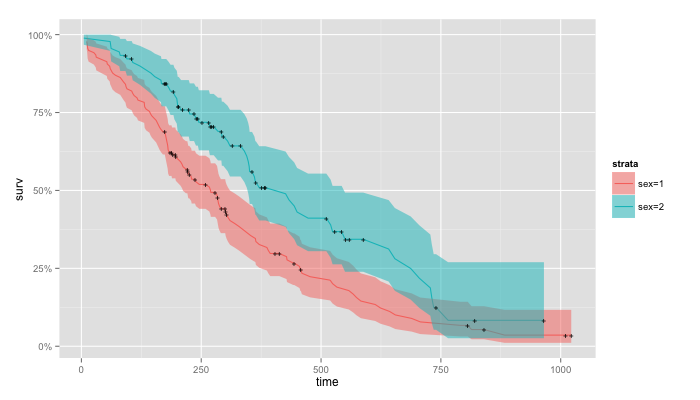
censorを描画したい場合は、
fortified <- ggplot2::fortify(d.survfit)
ggplot(data = fortified) +
geom_line(aes_string(x = 'time', y = 'surv', colour = 'strata')) +
geom_ribbon(aes_string(x = 'time', ymin = 'lower', ymax = 'upper', fill = 'strata'), alpha = 0.5) +
geom_point(data = fortified[fortified$n.censor > 0, ],
aes_string(x = 'time', y = 'surv'), shape = '+', size = 3) +
scale_y_continuous(labels = scales::percent)
スクリプト: Allow ggplot2 to handle survival::survfit result · GitHub
※survfit用の描画関数を直接定義するやり方もあるが、けっこう大変。
Creating good looking survival curves – the 'ggsurv' function | R-statistics blog
→ 10/20 追記: パッケージ化してみた。

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
ggplot2でforecastインスタンスを描画する
最近 時系列データを forecast パッケージを使って処理している。便利!
library(forecast) d <- AirPassengers d.arima <- forecast::auto.arima(d) d.forecast <- forecast(d.arima, level = c(95), h = 50) plot(d.forecast)
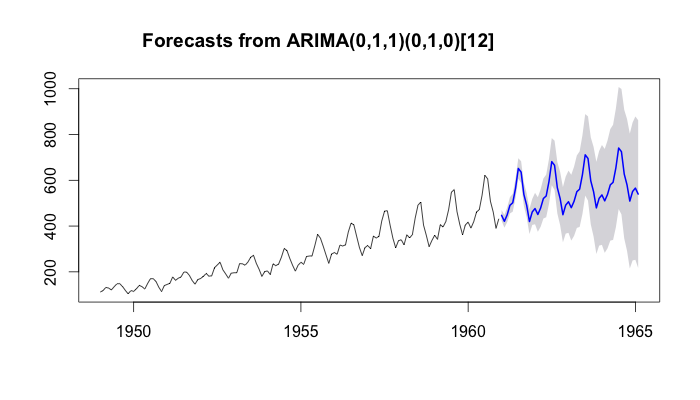
この結果を、総称関数 plot と同じように ggplot で描画したいが、そのまま ggplot に渡すとエラーになる。
library(ggplot2) ggplot(data = d.forecast) + geom_line(aes(x = x, y = fitted)) # エラー: ggplot2 doesn't know how to deal with data of class forecast
ggplot では内部で fortify という関数を使い、入力データを data.frame に変換している。ので forecast 用にも fortify を定義してやればよい。
forecastには forecast オブジェクト用の as.data.frame があるが、これは予測値/信頼区間のみしか data.frame 化しない。こちらに原系列を追加してやる必要がある。
fortify.forecast <- function(forecast.data) {
require(dplyr)
forecasted <- as.data.frame(forecast.data)
forecasted$Time <- as.Date(time(forecast.data$mean))
fitted <- data.frame(Time = as.Date(time(forecast.data$fitted)),
Original = forecast.data$x,
Fitted = forecast.data$fitted)
rownames(fitted) <- NULL
rownames(forecasted) <- NULL
dplyr::rbind_list(fitted, forecasted)
}
head(ggplot2::fortify(d.forecast))
# Time Original Fitted Point Forecast Lo 95 Hi 95
# 1 1949-01-01 112 111.9353 NA NA NA
# 2 1949-02-01 118 117.9664 NA NA NA
# 3 1949-03-01 132 131.9662 NA NA NA
# 4 1949-04-01 129 128.9774 NA NA NA
# 5 1949-05-01 121 120.9892 NA NA NA
# 6 1949-06-01 135 134.9782 NA NA NA
日時, 原系列, 推定値を Time, Original, Fitted カラムとして追加するようにした。これで ggplot から通常の data.frame と同じように扱える。スペースを含む列名はバッククオートでエスケープする。
ggplot(data = d.forecast) + geom_line(mapping = aes_string(x = 'Time', y = 'Original')) + geom_line(mapping = aes_string(x = 'Time', y = '`Point Forecast`'), colour='blue') + geom_ribbon(mapping = aes_string(x = 'Time', ymin = '`Lo 95`', ymax = '`Hi 95`'), alpha = 0.5)
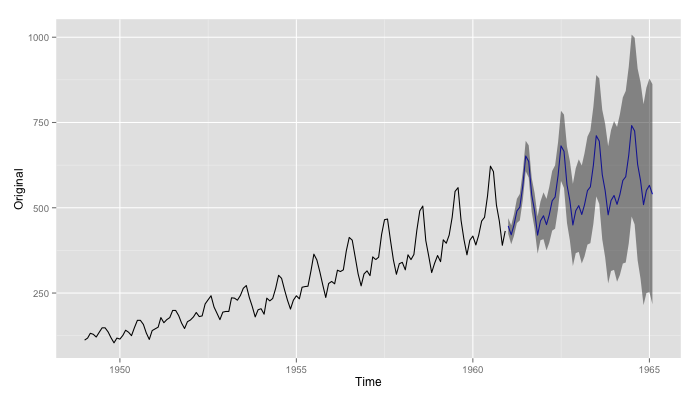
スクリプト: Allow ggplot2 to handle forecast result · GitHub
→ 10/20 追記: パッケージ化してみた。

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る