pandas 0.17.0 の主要な変更点
先日 10/9 に pandas 0.17.0 がリリースされた。直近のバージョンアップの中では かなり機能追加が多いリリースとなった。
重要な変更は リリースノート にハイライトとして列挙しているのだが、これらはある程度 pandas を使いこなしている方向けの記載となっている。
そのため、ここでは よりライトなユーザ向けに重要と思われる変更を書く。特に、ユーザ側のプログラムに影響がある以下の3点について記載する。
- ソート API の統合 (
sort_values/sort_index) - 重複削除 API の改善 (
drop_duplicates/duplicated) .plotアクセサの追加
準備
import numpy as np import pandas as pd np.__version__ # '1.10.1' pd.__version__ # u'0.17.0'
1. ソート API の統合 ( sort_values / sort_index )
これまで一貫性がなかったソート API が以下のように統合された。以前のメソッド / 引数は deprecate されており、将来のバージョン ( 通例では 0.19.0 以降 ) で削除される。
値によるソート ( sort_values として統合):
| 0.16.2以前 | 0.17.0以降 |
|---|---|
Series.order() |
Series.sort_values() |
Series.sort() |
Series.sort_values(inplace=True) |
DataFrame.sort(columns=...) |
DataFrame.sort_values(by=...) |
ラベル ( Index ) によるソート ( sort_index として統合):
| 0.16.2以前 | 0.17.0以降 |
|---|---|
Series.sort_index() |
Series.sort_index() |
Series.sortlevel(level=...) |
Series.sort_index(level=...) |
DataFrame.sort_index() |
DataFrame.sort_index() |
DataFrame.sortlevel(level=...) |
DataFrame.sort_index(level=...) |
DataFrame.sort() |
DataFrame.sort_index() |
それぞれ、引数として以下のオプションが利用できる。
inplace: 破壊的にソートするかどうかを指定する。既定はFalse(非破壊)axis: ソートの方向(軸)を指定するascending: ソート順序 (昇順/降順) を指定する。既定はTrue(昇順)
s = pd.Series(list('afcedb'), index=[1, 3, 2, 4, 0, 5]) s # 1 a # 3 f # 2 c # 4 e # 0 d # 5 b # dtype: object # 値によるソート s.sort_values() # 1 a # 5 b # 2 c # 0 d # 4 e # 3 f # dtype: object # ラベル (index) によるソート s.sort_index() # 0 d # 1 a # 2 c # 3 f # 4 e # 5 b # dtype: object
補足 もともとの仕様は NumPy (というか Python) の .sort メソッドの挙動 ( 破壊的なソート) にあわせたものだった。今回の変更により NumPy とは差異が発生する。
a = np.array([3, 2, 1]) a # array([3, 2, 1]) a.sort() a # array([1, 2, 3])
2. 重複削除 API の改善 ( drop_duplicates / duplicated )
重複した値の削除を行う drop_duplicates で、重複値の全削除ができるようになった。
| 0.16.2以前 | 0.17.0以降 |
|---|---|
.drop_duplicates() |
.drop_duplicates() |
.drop_duplicates(take_last=True) |
.drop_duplicates(keep='last') |
| - | .drop_duplicates(keep=False) |
削除方法の指定は これまでの take_last から keep オプション に変更された。
s = pd.Series(list('abebdcd')) s # 0 a # 1 b # 2 e # 3 b # 4 d # 5 c # 6 d # dtype: object # 重複がある場合、最初に出現した値を残す s.drop_duplicates() # 0 a # 1 b # 2 e # 4 d # 5 c # dtype: object # 重複がある場合、最後に出現した値を残す s.drop_duplicates(keep='last') # 0 a # 2 e # 3 b # 5 c # 6 d # dtype: object # 重複した値を残さない s.drop_duplicates(keep=False) # 0 a # 2 e # 5 c # dtype: object
また、値が重複しているかどうかを調べる .duplicated でも keep オプションが利用できる。
# 重複がある場合、最初に出現した値以外を True s.duplicated() # 0 False # 1 False # 2 False # 3 True # 4 False # 5 False # 6 True # dtype: bool # 重複がある場合、最後に出現した値以外を True s.duplicated(keep='last') # 0 False # 1 True # 2 False # 3 False # 4 True # 5 False # 6 False # dtype: bool # 重複 全てを True s.duplicated(keep=False) # 0 False # 1 True # 2 False # 3 True # 4 True # 5 False # 6 True # dtype: bool
Index の値から重複を全削除する場合は Index.duplicated を以下のように利用すればよい。
s = pd.Series(np.arange(6), index=list('abcbda')) s # a 0 # b 1 # c 2 # b 3 # d 4 # a 5 # dtype: int64 s[~s.index.duplicated(keep=False)] # c 2 # d 4 # dtype: int64
3. .plot アクセサの追加
DataFrame や Series をプロットする際のグラフの種類が、 .plot.bar() や .plot.hist() のように .plot をアクセサとして指定できるようになった。
これまでのメソッド呼び出し ( .plot(kind='bar') ) は引き続き利用できる。
df = pd.DataFrame(np.random.randn(5, 10)) # df.plot(kind='box') と同じ df.plot.box()

その他
また、上記ほどではないが重要な内容を列挙する。気になるものがあれば詳細はリンク先で。
- GIL 解放 (
Dask利用時のパフォーマンス向上) (詳細) pandas.io.dataを deprecate し、別パッケージpandas-datareaderとして分離 (詳細)pd.to_datetimeのエラー処理の変更、日時パース時の挙動統一 (詳細)Index同士の比較演算時の一部挙動の変更 (詳細)- ターミナル上での日本語データ表示時の位置補正オプションの追加 (詳細)
まとめ
pandas 0.17.0 での特に重要な変更点 3 点を記載した。
- ソート API の統合 (
sort_values/sort_index) - 重複削除 API の改善 (
drop_duplicates/duplicated) .plotアクセサの追加
これら以外の変更点については リリースノート を一読お願いします。
10/18 編集 コメントご指摘により誤記を修正

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (11件) を見る
PyConJP 2015: pandas/Daskについてお話させていただきました
10日、11日と PyCon JP に参加させていただきました。ご参加いただいた皆様、スタッフの皆様ありがとうございました。資料はこちらになります。
pandas internals
パフォーマンス向上のための pandas 内部実装の説明といくつかの TIPS について。そのうち翻訳するかもしれません。
Dask: 軽量並列分散フレームワーク (LT)
元ネタ
以下のエントリをベースに、それぞれ新しい内容を追加しています。
Python XGBoost + pandas 連携の改善
一部 こちらの続き。その後 いくつかプルリクを送り、XGBoost と pandas を連携させて使えるようになってきたため、その内容を書きたい。
できるようになったことは 以下 3 点。
DMatrixでのラベルと型の指定pd.DataFrameからのDMatrixの作成xgb.cvの結果をpd.DataFrameとして取得
補足 XGBoost では PyPI の更新をスクリプトで不定期にやっているようで、同一バージョンに見えても枝番が振られていたりして見分けにくい。記載は本日時点のこのコミットの情報。
%matplotlib inline import numpy as np import xgboost as xgb from sklearn import datasets import matplotlib.pyplot as plt plt.style.use('ggplot') xgb.__version__ # '0.4'
1. DMatrix でのラベルと型の指定
これは pandas 関係ない。DMatrix に以下 2 つのプロパティが追加され、任意のラベル/型が指定できるようにした (指定しない場合はこれまでと同じ挙動)。
feature_names: 変数のラベルを指定。ラベルは英数字のみ。feature_types: 変数の型を指定。指定できるのは、q: 量的変数 (既定)i: ダミー変数int: 整数型float: 浮動小数点型。
まずは feature_names のみを指定。
iris = datasets.load_iris() features = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth'] dm = xgb.DMatrix(iris.data, label=iris.target, feature_names=features) dm.feature_names # ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth']
DMatrix で指定した feature_names は xgb.Booster へも引き継がれ、.get_dump 時 やプロットに利用できる。
params={'objective': 'multi:softprob',
'eval_metric': 'mlogloss',
'eta': 0.3,
'num_class': 3}
np.random.seed(1)
bst = xgb.train(params, dm, num_boost_round=18)
xgb.plot_importance(bst)

次に feature_types。これは内部的な学習には全く関係がなく、.get_dump や xgb.plot_tree をする際の ラベルのフォーマットを決めるだけ。例えば以下のようなデータがあったとする。
data = np.array([[1, 0.1, 0], [2, 0.2, 1], [2, 0.2, 0]]) data # array([[ 1. , 0.1, 0. ], # [ 2. , 0.2, 1. ], # [ 2. , 0.2, 0. ]])
このとき、1 列目を int, 2 列目を float, 3 列目をダミー変数 i として扱いたければ以下のように指定すると、 plot_tree したときの見た目が若干変わる。全変数 既定 (q: 量的変数) でもとくに不自由ないので、強いこだわりのある方以外は指定する必要はない。
dm = xgb.DMatrix(data, feature_names=['A', 'B', 'C'], feature_types=['int', 'float', 'i']) dm.feature_types # ['int', 'float', 'i']
補足 これまで、変数のラベル/型の指定は "featmap.txt" のような設定ファイルから別途指定する必要があった。この指定が Python で直接できるようになった。
2 pd.DataFrame からの DMatrix の作成
pandas.DataFrame から DMatrix が作成できるようにした。このとき、feature_names と feature_types は DataFrame の定義から適当に設定される。
補足 DMatrix の label には pd.Series が渡せる (前からできた)。
import pandas as pd # 表示行数を指定 pd.set_option('display.max_rows', 8) # 一行に表示する文字数を指定 pd.set_option('display.width', 120) # pd.DataFrame を作成 train = pd.DataFrame(iris.data, columns=['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth']) train # SepalLength SepalWidth PetalLength PetalWidth # 0 5.1 3.5 1.4 0.2 # 1 4.9 3.0 1.4 0.2 # 2 4.7 3.2 1.3 0.2 # 3 4.6 3.1 1.5 0.2 # .. ... ... ... ... # 146 6.3 2.5 5.0 1.9 # 147 6.5 3.0 5.2 2.0 # 148 6.2 3.4 5.4 2.3 # 149 5.9 3.0 5.1 1.8 # # [150 rows x 4 columns] # DMatrix を作成 dm = xgb.DMatrix(train, label=pd.Series(iris.target)) dm.feature_names # ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth'] dm.feature_types # ['q', 'q', 'q', 'q']
DMatrix に変換できるのは、列が int64, float64, bool の DataFrame のみ。他の型を自動でダミー変数に変換したりはしない。
df = pd.DataFrame([[1, 0.1, False], [2, 0.2, True], [2, 0.2, False]], columns=['A', 'B', 'C']) df # A B C # 0 1 0.1 False # 1 2 0.2 True # 2 2 0.2 False df.dtypes # A int64 # B float64 # C bool # dtype: object dm = xgb.DMatrix(df) dm.feature_names # ['A', 'B', 'C'] dm.feature_types # ['int', 'q', 'i'] # object 型を含むためエラー df = pd.DataFrame([[1, 0.1, 'x'], [2, 0.2, 'y'], [2, 0.2, 'z']], columns=['A', 'B', 'C']) df # A B C # 0 1 0.1 x # 1 2 0.2 y # 2 2 0.2 z df.dtypes # A int64 # B float64 # C object # dtype: object xgb.DMatrix(df) # ValueError: DataFrame.dtypes must be int, float or bool
3. xgb.cv の結果を pd.DataFrame として取得
クロスバリデーションを行う xgb.cv はこれまで実行結果を文字列で返していたため、その結果をパースもしくは目視で確認する必要があった。この返り値を pd.DataFrame もしくは np.ndarray とし、プログラムで処理がしやすいようにした。
train = pd.DataFrame(iris.data, columns=['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth']) dm = xgb.DMatrix(train, label=pd.Series(iris.target)) cv = xgb.cv(params, dm, num_boost_round=50, nfold=10) cv # test-mlogloss-mean test-mlogloss-std train-mlogloss-mean train-mlogloss-std # 0 0.753459 0.027033 0.737631 0.003818 # 1 0.552303 0.048738 0.526929 0.005102 # 2 0.423481 0.066469 0.390115 0.005873 # 3 0.339942 0.082163 0.295637 0.006148 # .. ... ... ... ... # 46 0.208001 0.259161 0.018070 0.001759 # 47 0.208355 0.261166 0.017898 0.001724 # 48 0.208468 0.261520 0.017755 0.001703 # 49 0.208566 0.260967 0.017617 0.001686 # # [50 rows x 4 columns] cv.sort(columns='test-mlogloss-mean') # test-mlogloss-mean test-mlogloss-std train-mlogloss-mean train-mlogloss-std # 11 0.175506 0.177823 0.058496 0.004715 # 13 0.175514 0.194641 0.045222 0.004117 # 14 0.176150 0.203265 0.040493 0.004050 # 12 0.176277 0.186902 0.051074 0.004423 # .. ... ... ... ... # 3 0.339942 0.082163 0.295637 0.006148 # 2 0.423481 0.066469 0.390115 0.005873 # 1 0.552303 0.048738 0.526929 0.005102 # 0 0.753459 0.027033 0.737631 0.003818 # # [50 rows x 4 columns]
pandas がインストールされていない、もしくは as_pandas=False を指定した場合、返り値は np.ndarray となる。
cv = xgb.cv(params, dm, num_boost_round=50, nfold=10, as_pandas=False) cv # array([[ 0.7534586 , 0.02703308, 0.7376308 , 0.00381775], # ... # [ 0.2085664 , 0.26096721, 0.0176169 , 0.00168606]]) np.argmin(cv, axis=0) # array([11, 0, 49, 49])
まとめ
XGBoost と pandas をより便利に使うため、以下 3 点の修正を行った。
DMatrixでのラベルと型の指定pd.DataFrameからのDMatrixの作成xgb.cvの結果をpd.DataFrameとして取得
これ使って Kaggle で大勝利したい...と思ったのですが、他の日本勢に使っていただいたほうが勝率高いはずなのでシェアします。
Python Dask で 並列 DataFrame 処理
はじめに
先日のエントリで少し記載した Dask について、その使い方を書く。Dask を使うと、NumPy や pandas の API を利用して並列計算/分散処理を行うことができる。また、Dask は Out-Of-Core (データ量が多くメモリに乗らない場合) の処理も考慮した実装になっている。
上にも書いたが、Daskは NumPy や pandas を置き換えるものではない。数値計算のためのバックエンドとして NumPy や pandas を利用するため、むしろこれらのパッケージが必須である。
Dask は NumPy や pandas の API を完全にはサポートしていないため、並列 / Out-Of-Core 処理が必要な場面では Dask を、他では NumPy / pandas を使うのがよいと思う。pandasとDask のデータはそれぞれ簡単に相互変換できる。
補足 とはいえ都度の変換は手間なので、pandas の処理実行時に Dask を利用するオプションをつける という検討はされている。
インストール
pip もしくは conda で。
pip install dask
準備
まずは必要なパッケージを import する。
import numpy as np import pandas as pd import dask.dataframe as dd np.__version__ # '1.9.3' pd.__version__ # '0.16.2' # バージョン表示のためにインポート。 import dask dask.__version__ # '0.7.1'
pandas から Dask への変換
サンプルデータは すでにメモリ上にある pd.DataFrame とする。
df = pd.DataFrame({'X': np.arange(10),
'Y': np.arange(10, 20),
'Z': np.arange(20, 30)},
index=list('abcdefghij'))
df
# X Y Z
# a 0 10 20
# b 1 11 21
# c 2 12 22
# d 3 13 23
# e 4 14 24
# f 5 15 25
# g 6 16 26
# h 7 17 27
# i 8 18 28
# j 9 19 29
pandas のデータ構造から Dask に変換するには dd.from_pandas。2つめの引数で データをいくつのパーティションに分割するかを指定している。結果は dask.dataframe.DataFrame ( dd.DataFrame ) となる。
divisions はデータがどこで分割されたかを示す。表示から、1つ目のパーティションには .index が "a" 〜 "e" までのデータが、2つ目のには "f" 〜 "j" までのデータが含まれていることがわかる。
重要 dd.DataFrame の処理全般について、行の順序は保証されない。各パーティションには divisions で示される .index を持つ行が含まれるが、パーティション内が常にソートされているとは限らない。
ddf = dd.from_pandas(df, 2) ddf # dd.DataFrame<from_pandas-b08addf72f0693a9fa1bb6c21d91f3d4, divisions=('a', 'f', 'j')> # DataFrame の列名 ddf.columns # ('X', 'Y', 'Z') # DataFrame の index ddf.index # dd.Index<from_pandas-b08addf72f0693a9fa1bb6c21d91f3d4-index, divisions=('a', 'f', 'j')> # DataFrame の divisions (パーティションの分割箇所) ddf.divisions # ('a', 'f', 'j') # DataFrame のパーティション数 ddf.npartitions # 2
dd.DataFrame からデータを取得する (計算処理を実行する) には .compute()。結果、元の pd.DataFrame が得られる。
ddf.compute() # X Y Z # a 0 10 20 # b 1 11 21 # c 2 12 22 # d 3 13 23 # e 4 14 24 # f 5 15 25 # g 6 16 26 # h 7 17 27 # i 8 18 28 # j 9 19 29
内部処理
ここから、dd.DataFrame でどういった処理ができるのか、内部動作とあわせて記載する。といっても難しいことは全くやっていない。
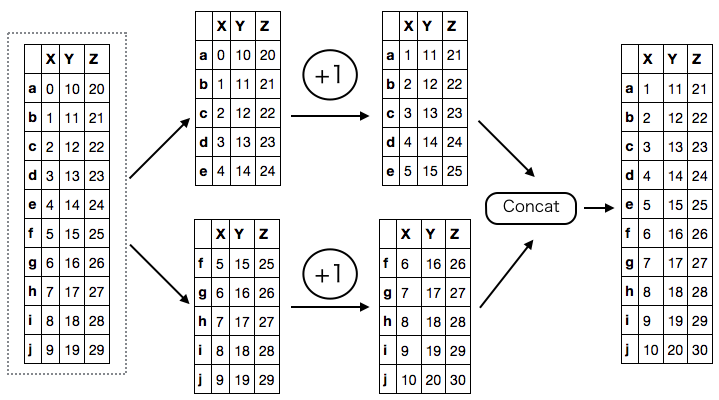
まず、データ全体について 1 加算する処理を考える。これは 各パーティションごとに 1 加算して連結するのと同じ。
ddf + 1 # dd.DataFrame<elemwise-5b9ae0407254158903343113fac41421, divisions=('a', 'f', 'j')> (ddf + 1).compute() # 略
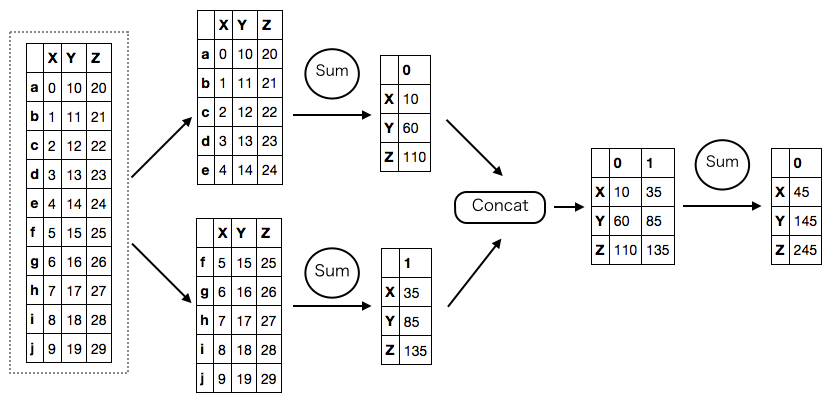
次に、列ごとの合計をとる処理。これは、各パーティションごとに列の合計をとって連結し、もう一度 合計をとる処理と同じ。
列の合計をとるため、結果は Series になる。ddf.sum() の時点では .compute() が呼ばれていないため実際の計算は行われていないが、Dask が結果の型 や divisions を推測し正しく表示している。
ddf.sum() # dd.Series<dataframe-sum--second-7ba12c9d58c17f61406b84b6c30d7a26, divisions=(None, None)> ddf.sum().compute() # X 45 # Y 145 # Z 245 # dtype: int64
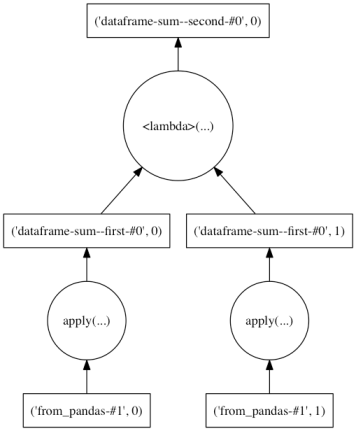
Dask ではこのような形で、計算処理をパーティションごとに並列 / Out-Of-Core 実行できる形に読み替えている。これらの処理は内部的には Computational Graph ( Dask Graph ) として表現され、.compute() によって実行される。
各処理の Dask Graph は、.visualize() メソッドを利用して確認できる。Graph 上で縦につながっていない処理同士は並列で実行できる。
ddf.sum().visualize()
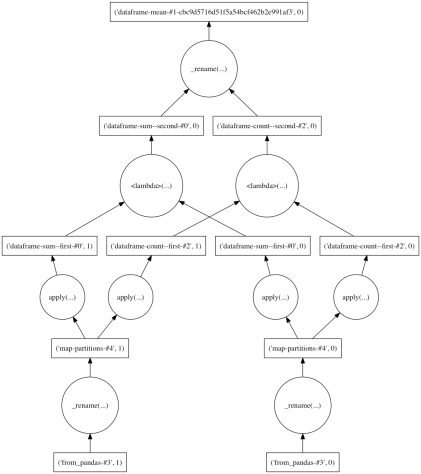
各列の平均をとる場合、内部的には各列の .sum()と 各列の .count() をそれぞれ計算して除算。
ddf.mean().compute() # X 4.5 # Y 14.5 # Z 24.5 # dtype: float64 ddf.mean().visualize()
DataFrame 同士の演算や、演算をチェインすることもできる。互いのパーティションが異なる場合はそれらが一致するよう調整が行われる。
((ddf - (ddf * 2)) == - ddf).visualize()
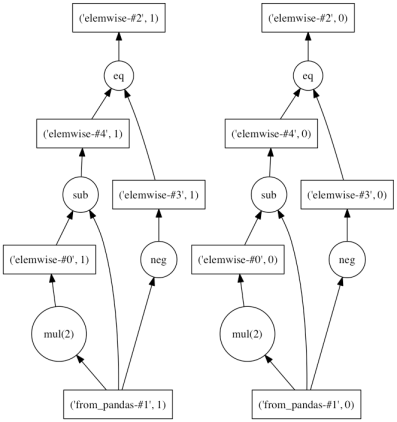
また、累積関数 ( cumxxx ) や ウィンドウ関数 ( rolling_xxx ) なども利用できる。
ddf.cumsum().compute() # X Y Z # a 0 10 20 # b 1 21 41 # c 3 33 63 # d 6 46 86 # e 10 60 110 # f 15 75 135 # g 21 91 161 # h 28 108 188 # i 36 126 216 # j 45 145 245 ddf.cumsum().visualize()

concat, join などの 連結 / 結合もできる。通常の演算と同じく、dd.DataFrame 同士のパーティションは適当に調整される。
df2 = pd.DataFrame({'A': np.arange(5),
'B': np.arange(10, 15)},
index=list('adefg'))
df2
# A B
# a 0 10
# d 1 11
# e 2 12
# f 3 13
# g 4 14
ddf2 = dd.from_pandas(df2, 2)
ddf2
# dd.DataFrame<from_pandas-667963fc37e22688843f02da80df5963, divisions=('a', 'f', 'g')>
ddf.join(ddf2).compute()
# X Y Z A B
# a 0 10 20 0 10
# b 1 11 21 NaN NaN
# c 2 12 22 NaN NaN
# d 3 13 23 1 11
# e 4 14 24 2 12
# f 5 15 25 3 13
# g 6 16 26 4 14
# h 7 17 27 NaN NaN
# i 8 18 28 NaN NaN
# j 9 19 29 NaN NaN
ddf.join(ddf2).visualize()
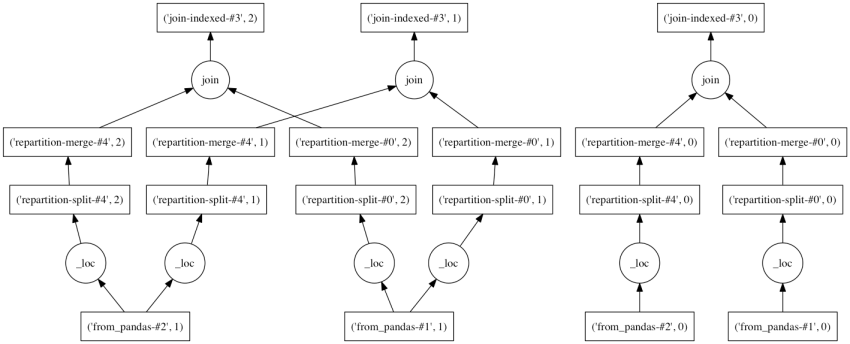
サポートされている処理の一覧は以下のAPIドキュメントを。一部利用できない引数が明記されていないが、次バージョンにて改訂。
9/26 追記 処理結果については、行の順序以外は pandas の処理と一致するはず。例外は quantile のような percentile をとる処理。これらは Out-Of-Core 処理のための近似アルゴリズムを使っており、正確な値とずれることがある。
実データでの利用例
こちらが良エントリ (英語)。
- Analyzing Reddit Comments with Dask and Castra
- Out-of-Core Dataframes in Python: Dask and OpenStreetMap
まとめ
Dask を利用して DataFrame を並列処理する方法を記載した。手順は、
dd.from_pandasを利用してpd.DataFrameをdd.DataFrameへ変換。- 実行したいメソッド / 演算を
dd.DataFrameに対して適用。 .compute()で計算を実行し、結果を取得する。計算処理はDaskにて自動的に並列化される。
最後、pandas 0.16.2 時点では並列処理による速度向上は大きくはない。これは Python の GIL (Global Interpreter Lock ) により並列実行できる処理が限定されているため。今月中にリリース予定の pandas 0.17.0 では いくつかの処理で Cython から明示的に GIL 解放するよう実装を変更しており、並列化による速度向上は大きくなる。
Python 次世代の多次元配列パッケージ群
このところ、たびたび NumPy 後継が...とか 並列処理が...という話を聞くので、この秋 注目の多次元配列パッケージをまとめたい。
バックエンド系
NumPy のように数値計算処理を自前で実装しているパッケージ。
DyND
Blaze プロジェクトのひとつ。C++ 実装 + Python バインディング。GitHub にいくつか Example があがっているが、複合型やカテゴリカル型、GroupBy 操作がサポートされていて熱い。ラベルデータも NumPy より簡単に実装できそうだ。
並列分散系
自身では直接 数値計算処理を行わず、バックエンド ( 主に NumPy )を利用して並列/分散処理を行うパッケージ。1 物理PC/複数コアでの並列計算を主用途とし、NumPy, pandas では少し苦しいが PySpark などを使うほどじゃない...という場合に利用するもの。
Dask
Blaze プロジェクトのひとつ。Pure-Python 実装。主用途は単一物理PCでの複数コア計算だが、複数PCに処理を分散させることもできる。
NumPy の API で並列計算を行う dask.array のほか、toolz 相当の操作を行う dask.bag、pandas 相当の処理を行う dask.dataframe など一連のデータ構造が揃っている。
開発者はベースパッケージである NumPy、toolz、pandas のコミッタとの兼任が多い。自分もコミット権限をいただいており、dask.dataframe への API 追加を行なっている。
DistArray
NumPy の API で並列計算を行う DistArray のみをサポート、ほかのデータ構造はない。Pure-Python 実装。見た感じ、並列処理の基本的な考え方は Dask と同じようだ。Enthought が好きな方はこちらを使えばよいかと。
bolt
NumPy の API でローカル計算/分散処理を行う。Pure-Python実装。ローカル計算は NumPy をそのまま使い (並列処理しない)、分散処理は Spark で行う。海外の方が期待の新星っぽい扱いをしていたので気になったのだが、現時点で実装されている API は多くはない。
ほかにもいくつかプロジェクトがあるが、自分としては Dask を流行らせたいので、その記事を書きます。
9/24 追記 書きました。
NumPy でつくる俺々データ構造
はじめに
Python での数値計算の基盤をなす NumPy 、直感的なスライスやブロードキャスト、関数のベクトル適用など大変便利だ。
import numpy as np np.__version__ # '1.9.2' np.array([1, 2, 3]) # array([1, 2, 3]) np.array([1, 2, 3])[:2] # array([1, 2]) np.array([1, 2, 3]) + 1 # array([2, 3, 4])
が、用途によっては NumPy 標準ではその機能を実現できない場合がある。例えば、
- 配列とメタデータをひとつのクラスで扱いたい
- 配列への入力や型を制約/検証したい
- 自作クラスを NumPy の Universal Functions (ufunc) に対応させたい
- 新しい型 (
dtype) を作りたい
こういったとき、NumPy の動作を継承した自作のクラスが作れるとうれしい。方法として、大きく二つがある。
- NumPy
ndarrayを継承する - NumPy Array Interface を実装する
どちらを使うべきかは目的による。サブクラス化しないほうが柔軟な処理ができるが、互換をとるための実装は多くなる。pandas の主要データ構造では 柔軟性を重視して ndarray の継承をやめ、現在は 2 の方法を取っている。
ndarray のサブクラスを作成する方法は以下のドキュメントに記載されている。きちんとやりたい場合はこちらを読むのがよい。
このエントリでは 2. Array Interface を使って最低限の動作をさせる方法を書く。
クラスの定義
サンプルとして、名前をもつ配列クラス NamedArray を考える。__init__での初期化時に、名前をあらわす name と配列の値 values を渡すことにする。
class NamedArray(object): def __init__(self, name, values): self.name = name self.values = np.array(values) def __repr__(self): return 'NamedArray: {0}: {1}'.format(self.name, self.values) def __len__(self): return len(self.values) n = NamedArray('x', [1, 2, 3]) n # NamedArray: x: [1 2 3] len(n) # 3
このクラスに ndarray と互換の挙動をさせたい。まず、NamedArray インスタンスから ndarray が直接作成できるとうれしい。が、NamedArray を np.array に渡すと shape のない ndarray (0-dimmentional array) が作成されてしまう。
np.array(n) # array(NamedArray: x: [1 2 3], dtype=object) np.array(n).shape # ()
理由は np.array が NamedArray をスカラーとして扱うため。期待の動作をさせるためには、np.array に NamedArray クラスが配列であることを教える必要がある。
具体的には NamedArray に Array Interface ( __array__ ) を定義すればよい。以下のように __array__ メソッドを追加したクラス NPNamedArray を作成すると、期待通り values の値から ndarray が作成されていることがわかる。
class NPNamedArray(NamedArray): def __array__(self): return self.values n = NPNamedArray('x', [1, 2, 3]) np.array(n) # array([1, 2, 3]) np.array(n).shape # (3,)
ufunc 処理のオーバーライド
また、Array Interface をもつインスタンスには ufunc が適用できるようになる。引数は ufunc 内で ndarray に変換されるため、返り値は ndarray になる。
np.sin(n)
# array([ 0.84147098, 0.90929743, 0.14112001])
このときの内部の動きをみてみる。ufunc 適用後の処理は __array_wrap__ メソッドによって制御することができる。
__array_wrap__ メソッドには out_arr と context 2 つの引数を定義する必要がある。これらに何が渡されているのか、print して確認する。
class NPNamedArray(NamedArray): def __array__(self): return self.values def __array_wrap__(self, out_arr, context=None): print('out_arr: ', out_arr) print('context: ', context) return out_arr n = NPNamedArray('x', [1, 2, 3]) np.sin(n) # ('out_arr: ', array([ 0.84147098, 0.90929743, 0.14112001])) # ('context: ', (<ufunc 'sin'>, (NamedArray: x: [1 2 3],), 0)) # array([ 0.84147098, 0.90929743, 0.14112001])
out_arr には ufunc が適用された結果の ndarray が、 context には ufunc を呼び出した際のシグネチャ ( ufunc 自体, ufunc の引数, ufunc のドメイン ) が tuple で渡ってきている。__array_wrap__ の返り値を NPNamedArray に書き換えてやれば、期待する動作ができそうだ。
補足 np.isreal や np.ones_like など、Universal Functions に記載されていても __array_wrap__ を通らないものもある。
補足 ufunc のドメインは NumPy の場合 (おそらく) 全部 0。
class NPNamedArray(NamedArray): def __array__(self): return self.values def __array_wrap__(self, out_arr, context=None): return NPNamedArray(self.name, out_arr) n = NPNamedArray('x', [1, 2, 3]) np.sin(n) # NamedArray: x: [ 0.84147098 0.90929743 0.14112001]
ここまでの時点で、NPNamedArray には __add__ メソッドを定義していないため、通常の加算はエラーになる。
n + 1 # TypeError: unsupported operand type(s) for +: 'NPNamedArray' and 'int'
が、ufunc である np.add や ndarray を含む演算は利用できるようになる。これは先日のエントリに記載した通り ndarray 側に Array Interface をもつクラスとの演算の定義があるため。
np.add(n, 1) # NamedArray: x: [2 3 4] n + np.array([5, 5, 5]) # NamedArray: x: [6 7 8]
また、__array_wrap__ の context 引数の値によって、特定の ufunc の処理をオーバーライドすることができる。例えば np.sin を適用したときに任意の値を返すこともできる。
class NPNamedArray(NamedArray): def __array__(self): return self.values def __array_wrap__(self, out_arr, context=None): if context[0] is np.sin: return u'返したい値' return NPNamedArray(self.name, out_arr) n = NPNamedArray('x', [1, 2, 3]) np.sin(n) # u'ひまわり'
とはいえ、上のように個々の関数について条件分岐を作成するのは手間だ。こんなとき、 ufunc 自体のプロパティを参照することで より簡単に書ける場合がある。
例えば、特定の型への処理をサポートする ufunc のみを利用する場合は .types プロパティを参照すればよい。このプロパティは ufunc がどのような引数/返り値の型を取るかを示す Array-protocol Type String を返す。
参考
np.sin.types # ['e->e', 'f->f', 'd->d', 'g->g', 'F->F', 'D->D', 'G->G', 'O->O'] np.cos.types # ['e->e', 'f->f', 'd->d', 'g->g', 'F->F', 'D->D', 'G->G', 'O->O'] np.add.types # ['??->?', 'bb->b', 'BB->B', 'hh->h', 'HH->H', 'ii->i', 'II->I', 'll->l', 'LL->L', # 'qq->q', 'QQ->Q', 'ee->e', 'ff->f', 'dd->d', 'gg->g', 'FF->F', 'DD->D', 'GG->G', # 'Mm->M', 'mm->m', 'mM->M', 'OO->O']
まとめ
NumPy 互換の動作を実現する方法のうち、Array Interface を利用した方法を記載した。自分で一から実装するよりは簡単だと思う。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (12件) を見る
Python でパイプ演算子を使いたい
ネタ記事です。/ This is a joke post which makes no practical sense.
はじめに
Python pandas では主要な操作を以下のようにメソッドチェインの形で書くことができる。
# Python (pandas) df.assign(x=df['y'] + df['z']).groupby('x').sum()
pandas v0.16.2 で DataFrame と Series に .pipe というメソッドが追加され、このチェインを外部の関数/メソッドに対して連結できるようになった。利用例は以下のリンクを。
補足 matplotlib でも v1.5.0 で ラベルデータ対応 が追加され、各関数が .pipe から利用できるようになる予定。
このメソッドチェインによる "処理の連結" を、R ( {magrittr} ) のようにパイプ演算子 %>% を使って統一的に書きたい。
# R (magrittr + dplyr) df %>% mutate(x = y + z) %>% group_by(x) %>% summarize_each(funs(sum))
R と異なり、 Python では自作の演算子を定義することはできない。Python の演算子の中では 右方向ビットシフト演算子 >> がパイプ演算子っぽく見えなくもない。これを使って R のような動作をさせたい。
演算子のオーバーライド
まずはサンプルデータを作成する。
import numpy as np import pandas as pd pd.__version__ # '0.16.2' df = pd.DataFrame({'X': [1, 2, 3, 4, 5], 'Y': ['a', 'b', 'a', 'b', 'a']}) df # X Y # 0 1 a # 1 2 b # 2 3 a # 3 4 b # 4 5 a
普通に .pipe を使うとこのような感じになる。
func = lambda x: x df.pipe(func) # X Y # 0 1 a # 1 2 b # 2 3 a # 3 4 b # 4 5 a
この処理を >> 演算子を使って書きたい。そのままでは、演算方法が未定義のため TypeError となる。
df >> func
# TypeError: unsupported operand type(s) for >>: 'DataFrame' and 'function'
Python では各演算子に対応する処理はマジックメソッドとして実装する。そのため、>> に対応する __rshift__ に .pipe と同じ処理をさせればよい。また、R のように 右辺には関数を渡したいので、pandas のメソッドを適当にラップする関数 pipify を定義する。
pd.DataFrame.__rshift__ = pd.DataFrame.pipe def pipify(f): def wrapped(*args, **kwargs): return lambda self: f(self, *args, **kwargs) return wrapped head = pipify(pd.DataFrame.head)
できた。
df >> head(n=2) # X Y # 0 1 a # 1 2 b
groupby も同じように書ける。集約関数は DataFrame や DataFrameGroupBy 双方から使うため以下のように定義する。
groupby = pipify(pd.DataFrame.groupby) def sum(*args, **kwargs): return lambda self: self.sum(*args, **kwargs)
また、groupby した結果からも >> で演算を連結したい。そのためにDataFrameGroupBy.__rshift__ を定義する。
def _pipe(self, func): return func(self) pd.core.groupby.DataFrameGroupBy.__rshift__ = _pipe
できた。
df >> groupby('Y') >> sum() # X # Y # a 9 # b 6 df >> sum() # X 15 # Y ababa # dtype: object
右辺側クラスによるオーバーライド
DataFrame が左辺にある場合、パイプ演算は上記のように定義できた。加えて、DataFrame が右辺にある場合も同じようなことがしたい。
以下のような記法で、左辺の辞書から DataFrame を作成したい。これも (当然) そのままではできない。
data = {'X': [1, 2, 3, 4, 5],
'Y': ['a', 'b', 'a', 'b', 'a']}
data >> pd.DataFrame()
# TypeError: unsupported operand type(s) for >>: 'dict' and 'DataFrame'
Python では、左辺側に 右辺を処理できるマジックメソッドがない場合は、右辺側のマジックメソッド ( DataFarme.__rrshift__ ) が利用される。これを適当に定義すればよい。
def _get(a, b): return pd.DataFrame(b) pd.DataFrame.__rrshift__ = _get
できた。
data = {'X': [1, 2, 3, 4, 5],
'Y': ['a', 'b', 'a', 'b', 'a']}
data >> pd.DataFrame()
# X Y
# 0 1 a
# 1 2 b
# 2 3 a
# 3 4 b
# 4 5 a
NumPy に対するオーバーライド
DataFrame は np.ndarray からも作成することができる。が、>> ではうまく動かない。
data = np.array([[1, 2], [3, 4]]) pd.DataFrame(data) # 0 1 # 0 1 2 # 1 3 4 data >> pd.DataFrame() # TypeError: ufunc 'right_shift' not supported for the input types, and the inputs could not be safely coerced to any supported types according to the casting rule ''safe''
ndarray のマジックメソッドは スカラーや ndarray ライクな引数を汎用的に処理できるように定義されている。具体的には、引数が ndarray のサブクラス、もしくは Array Interface ( __array__ ) をもつとき、ndarray によって処理が行われる。
DataFrame は ndarray のサブクラスではないが Array Interface をもつ。右辺が不正なため通常の例外が送出されてしまい、右辺側のマジックメソッドに処理が移らない。
参考 Subclassing ndarray — NumPy v1.11 Manual
issubclass(pd.DataFrame, np.ndarray) # False df.__array__() # array([[1, 'a'], # [2, 'b'], # [3, 'a'], # [4, 'b'], # [5, 'a']], dtype=object)
ここで DataFrame 側のマジックメソッドを利用したい場合、上記リンクに記載されている __array_priority__ プロパティを設定すればよい。DataFrame 側の優先度を上げると期待した処理が行われることがわかる。
pd.DataFrame.__array_priority__ = 1000 data >> pd.DataFrame() # 0 1 # 0 1 2 # 1 3 4
まとめ
R 使ったほうがいい。Use R!
補足 こんなパッケージもあります。
2016/10/18 追記
続き(?)です。
Python XGBoost の変数重要度プロット / 可視化の実装
Gradient Boosting Decision Tree の C++ 実装 & 各言語のバインディングである XGBoost、かなり強いらしいという話は伺っていたのだが自分で使ったことはなかった。こちらの記事で Python 版の使い方が記載されていたので試してみた。
その際、Python でのプロット / 可視化の実装がなかったためプルリクを出した。無事 マージ & リリースされたのでその使い方を書きたい。まずはデータを準備し学習を行う。
import numpy as np import xgboost as xgb from sklearn import datasets import matplotlib.pyplot as plt plt.style.use('ggplot') xgb.__version__ # '0.4' iris = datasets.load_iris() dm = xgb.DMatrix(iris.data, label=iris.target) np.random.seed(1) params={'objective': 'multi:softprob', 'eval_metric': 'mlogloss', 'eta': 0.3, 'num_class': 3} bst = xgb.train(params, dm, num_boost_round=18)
1. 変数重要度のプロット
Python 側には R のように importance matrix を返す関数がない。GitHub 上でも F score を見ろ、という回答がされていたので F score をそのままプロットするようにした。
xgb.plot_importance(bst)

棒グラフの色、タイトル/軸のラベルは以下のように変更できる。
xgb.plot_importance(bst, color='red', title='title', xlabel='x', ylabel='y')

color にリストを渡せば棒ごとに色が変わる。色の順序は matplotlib の barh と同じく下からになる。また、ラベルを消したい場合は None を渡す。
xgb.plot_importance(bst, color=['r', 'r', 'b', 'b'], title=None, xlabel=None, ylabel=None)

XGBoost は内部的に変数名を保持していない。変数名でプロットしたい場合は 一度 F score を含む辞書を取得して、キーを差し替えてからプロットする。
bst.get_fscore() # {'f0': 17, 'f1': 16, 'f2': 95, 'f3': 59} iris.feature_names # ['sepal length (cm)', # 'sepal width (cm)', # 'petal length (cm)', # 'petal width (cm)'] mapper = {'f{0}'.format(i): v for i, v in enumerate(iris.feature_names)} mapped = {mapper[k]: v for k, v in bst.get_fscore().items()} mapped # {'petal length (cm)': 95, # 'petal width (cm)': 59, # 'sepal length (cm)': 17, # 'sepal width (cm)': 16} xgb.plot_importance(mapped)

2. 決定木のプロット
以下二つの関数を追加した。graphviz が必要なためインストールしておくこと。
to_graphviz: 任意の決定木をgraphvizインスタンスに変換する。IPython上であればそのまま描画できる。plot_tree:to_graphvizで取得したgraphvizインスタンスをmatplotlibのAxes上に描画する。
IPython から実行する。num_trees で指定した番号に対応する木が描画される。
xgb.to_graphviz(bst, num_trees=1)

エッジの色分けが不要なら明示的に黒を指定する。
xgb.to_graphviz(bst, num_trees=2, yes_color='#000000', no_color='#000000')

IPython を使っていない場合や、サブプロットにしたい場合には plot_tree を利用する。
_, axes = plt.subplots(1, 2) xgb.plot_tree(bst, num_trees=2, ax=axes[0]) xgb.plot_tree(bst, num_trees=3, ax=axes[1])

何かおかしいことをやっていたら 本体の方で issue お願いします。
10/3追記 その後の修正を以下にしました。変数名の指定などが簡単になっています。
Python xray で 多次元データを pandas ライクに扱う
はじめに
pandas では 2 次元、表形式のデータ ( DataFrame ) を主な対象としているが、ときには 3 次元以上のデータを扱いたい場合がある。そういった場合 以下のような方法がある。
MultiIndexを使い、2 次元のデータにマッピングする。- 3 次元データ構造である
Panel、4 次元のPanel4D、もしくは任意の次元のデータ構造 (PanelND) をファクトリ関数 で定義して使う。 numpy.ndarrayのまま扱う。
自分は MultiIndex を使うことが多いが、データを 2 次元にマップしなければならないため 種類によっては直感的に扱いにくい。Panel や PanelND は DataFrame と比べると開発が活発でなく、特に Panel4D、PanelND は 現時点で Experimental 扱いである。また、今後の扱いをどうするかも議論がある。numpy.ndarray では データのラベル付けができない。
xray とは
ラベル付きの多次元データを多次元のまま 直感的に扱えるパッケージとして xray がある。作者は pandas 開発チーム仲間の shoyer だ。そのため、API は pandas にかなり近いものになっている。
xray は大きく以下ふたつのデータ構造を持つ。これらを使うと多次元データをより直感的に操作することができる。
xray.DataArray:numpyの多次元配列にラベルでのアクセスを追加したもの。データは任意の次元を持つことができる。xray.Dataset: 複数のxray.DataArrayをまとめるセット。
インストール
pip で。
$ pip install xray
データの準備
まず、必要なパッケージをインポートする。
import numpy as np import pandas as pd pd.__version__ # '0.16.2' import xray xray.__version__ # '0.5.2'
サンプルデータとして、気象庁から 2015年7月20日〜25日の東京、八王子、大島の最高気温のデータを使いたい。以下のサイトからダウンロードした。データは 日付、場所 2 次元の配列となる。
- 出典:気象庁ホームページ (URL: http://www.data.jma.go.jp/gmd/risk/obsdl/index.php)

data2 = np.array([[34.2, 30.2, 33.5], [36.0, 29.3, 34.9], [35.3, 29.7, 32.8], [30.1, 27.6, 30.4], [33.6, 30.1, 33.9], [34.1, 28.0, 33.1]])
この numpy.ndarray から xray.DataArray インスタンスを作成する。データへアクセスするためのラベル ( pandas でいう index のようなもの ) は coords キーワードで指定する。また、dims キーワードを使って各次元それぞれにも名前をつけることができる。ここでは 以下のような DataArray を作成している。
- データは 2 つの次元
date,locationを持つ。 date次元はdatesで指定される 6 つの日付のラベルを持つ。location次元はlocsで指定される 3 つの場所のラベルを持つ。
2 次元のため、pandas.DataFrame でいうと date が indexに、 location が columns に対応しているイメージ。
locs = [u'八王子', u'大島', u'東京'] dates = pd.date_range('2015-07-20', periods=6, freq='D') da2 = xray.DataArray(data2, coords=[dates, locs], dims=['date', 'location']) da2 # <xray.DataArray (date: 6, location: 3)> # array([[ 34.2, 30.2, 33.5], # [ 36. , 29.3, 34.9], # [ 35.3, 29.7, 32.8], # [ 30.1, 27.6, 30.4], # [ 33.6, 30.1, 33.9], # [ 34.1, 28. , 33.1]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 各次元の名前 da2.dims # ('date', 'location') # 各次元の詳細 da2.coords # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # date 次元のラベル da2.coords['date'] # <xray.DataArray 'date' (date: 6)> # array(['2015-07-20T09:00:00.000000000+0900', # '2015-07-21T09:00:00.000000000+0900', # '2015-07-22T09:00:00.000000000+0900', # '2015-07-23T09:00:00.000000000+0900', # '2015-07-24T09:00:00.000000000+0900', # '2015-07-25T09:00:00.000000000+0900'], dtype='datetime64[ns]') # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ...
データの選択
xray でのデータ選択は pandas と類似の方法で行える。pandas でのデータ選択についてはこちらを。
ある次元から、特定のラベルをもつデータを選択したい場合、pandas と同じく .loc が使える。
# 7/25 のデータを選択 da2.loc[pd.Timestamp('2015-07-25')] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
また、対象の次元が datetime64 型の場合は、pandas と同じく日時文字列でも指定が可能 (部分文字列によるスライシング、詳細以下)。
# 7/25 のデータを選択 da2.loc['2015-07-25'] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
また、ラベルではなく位置 ( n番目など ) によって選択する場合は __getitem__ を使う。pandas では .iloc に対応。
# 末尾 = 最新の日付のデータを取得 da2[-1] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
2 次元目以降をラベル / 位置によって指定する場合は、引数をカンマで区切り、選択に利用する次元の位置に対応する値を渡す。
# date は全選択、location が東京のデータを選択 da2.loc[:, u'東京'] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # date は全選択、location が 3 番目 = 東京のデータを選択 da2[:, 2] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac'
データ選択に利用する次元自体もラベルで指定したい場合は .sel を使う。.sel では次元の順序に関係なく値を指定できるため、高次元になった場合もシンプルだ。
# 東京のデータを選択 da2.sel(location=u'東京') # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # 7/22, 7/23 の 東京のデータを選択 da2.sel(location=u'東京', date=pd.DatetimeIndex(['2015-07-22', '2015-07-23'])) # <xray.DataArray (date: 2)> # array([ 32.8, 30.4]) # Coordinates: # * date (date) datetime64[ns] 2015-07-22 2015-07-23 # location <U3 u'\u6771\u4eac'
もしくは .loc, __getitem__ に以下のような辞書を渡してもよい。
# 東京のデータを選択 da2.loc[dict(location=u'東京')] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac'
公式ドキュメント では、xray.DataArray からのデータ選択を以下のような表で整理している。
| 次元の指定 | 値の指定 | DataArray のメソッド |
|---|---|---|
| 位置 | 位置 | da2[:, 0] |
| 位置 | ラベル | da2.loc[:, u'東京'] |
| ラベル | 位置 | da2.isel(location=0), da2[dict(space=0)] |
| ラベル | ラベル | da2.sel(location=u'東京'), da2.loc[dict(location=u'東京')] |
次元の追加
ここまでは 2 次元のデータを使っていたが、最低気温と平均気温のデータを追加して 3 次元のデータとする。
# 最低気温 data3 = np.array([[24.7, 25.1, 25.8], [23.4, 24.3, 25.4], [22.7, 23.8, 25.4], [24.5, 24.4, 24.7], [24.9, 24.6, 25.0], [23.7, 24.8, 24.8]]) # 平均気温 data4 = np.array([[27.8, 26.5, 28.8], [29.7, 26.3, 29.4], [29.5, 26.5, 28.9], [26.9, 25.3, 27.0], [27.7, 26.4, 28.1], [29.0, 26.1, 28.6]]) data = np.dstack([data2, data3, data4]) # 日時、場所、データの種類 の 3 次元 data.shape # (6, 3, 3) da3 = xray.DataArray(data, coords=[dates, locs, [u'最高', u'最低', u'平均']], dims=['date', 'location', 'type']) da3 # <xray.DataArray (date: 6, location: 3, type: 3)> # array([[[ 34.2, 24.7, 27.8], # [ 30.2, 25.1, 26.5], # [ 33.5, 25.8, 28.8]], # # ... # # [[ 34.1, 23.7, 29. ], # [ 28. , 24.8, 26.1], # [ 33.1, 24.8, 28.6]]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
3 次元以上の場合もデータ選択のルールは一緒なのでわかりやすい。
# 7/24 のデータを選択 da3.loc['2015-07-24'] # <xray.DataArray (location: 3, type: 3)> # array([[ 33.6, 24.9, 27.7], # [ 30.1, 24.6, 26.4], # [ 33.9, 25. , 28.1]]) # Coordinates: # date datetime64[ns] 2015-07-24 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # 7/24 の 最高気温のデータを選択 da3.loc['2015-07-24', :, u'最高'] # <xray.DataArray (location: 3)> # array([ 33.6, 30.1, 33.9]) # Coordinates: # date datetime64[ns] 2015-07-24 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # type <U2 u'\u6700\u9ad8' # 東京 の 最高気温のデータを選択 da3.sel(location=u'東京', type=u'最高') # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # type <U2 u'\u6700\u9ad8'
算術演算
pandas と同じく、xray.DataArray 同士での算術演算が可能。各日の最高気温と最低気温の差を求めると、
da3.sel(type=u'最高') - da3.sel(type=u'最低') # <xray.DataArray (date: 6, location: 3)> # array([[ 9.5, 5.1, 7.7], # [ 12.6, 5. , 9.5], # [ 12.6, 5.9, 7.4], # [ 5.6, 3.2, 5.7], # [ 8.7, 5.5, 8.9], # [ 10.4, 3.2, 8.3]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 結果から 八王子 のデータだけを選択 (da3.sel(type=u'最高') - da3.sel(type=u'最低')).sel(location=u'八王子') # <xray.DataArray (date: 6)> # array([ 9.5, 12.6, 12.6, 5.6, 8.7, 10.4]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u516b\u738b\u5b50'
データのグループ化 / 集約
グループ化 / 集約も pandas とほぼ同じ形式でできる。location によってグループ化し、期間中の最高気温を出してみる。
da3.groupby('location') # <xray.core.groupby.DataArrayGroupBy at 0x109a56f90> da3.groupby('location').max() # <xray.DataArray (location: 3)> # array([ 36. , 30.2, 34.9]) # Coordinates: # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
グループ化した結果は、イテレーションによって順番に処理することもできる。
for name, g in da3.groupby('location'): print(name) print(g) # 八王子 # <xray.DataArray (date: 6, type: 3)> # array([[ 34.2, 24.7, 27.8], # [ 36. , 23.4, 29.7], # [ 35.3, 22.7, 29.5], # [ 30.1, 24.5, 26.9], # [ 33.6, 24.9, 27.7], # [ 34.1, 23.7, 29. ]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # location <U4 u'\u516b\u738b\u5b50' # # 以降略
データの結合 / 連結
上で作成した DataArray に、別のデータを追加したい。適当なデータを探したところ、ある成人男性のお住まいの気温データ を見つけた。これを日別で集計して連結したい。
# GitHub からデータを取得 df = pd.read_csv('https://raw.githubusercontent.com/dichika/mydata/master/room.csv') df['time'] = pd.to_datetime(df['time']) # light は光量、temperatur は気温 df.head() # time light temperature # 0 2015-02-12 01:45:04 463.0 26.784 # 1 2015-02-12 02:00:03 473.0 26.630 # 2 2015-02-12 02:15:04 467.9 25.983 # 3 2015-02-12 02:30:04 0.0 25.453 # 4 2015-02-12 02:45:04 0.0 23.650 # 期間中にフィルタ df = df[df['time'] >= pd.Timestamp('2015-07-20')] # 日時でグループ化 / 集約 agg = df.groupby(pd.Grouper(key='time', freq='D'))['temperature'].agg(['max', 'min', 'mean']) agg # max min mean # time # 2015-07-20 28.374 26.450 27.484604 # 2015-07-21 33.790 26.800 30.132792 # 2015-07-22 34.180 27.070 30.134375 # 2015-07-23 28.779 27.412 28.290918
xray でデータを連結するためには、連結する次元 (ここでは location) 以外のデータの要素数を一致させる必要がある。上記のデータは数日遅れで公開されているため、直近を NaN でパディングして xray.DataArray を作成する。
agg.loc[pd.Timestamp('2015-07-24'), :] = np.nan agg.loc[pd.Timestamp('2015-07-25'), :] = np.nan agg # max min mean # time # 2015-07-20 28.374 26.450 27.484604 # 2015-07-21 33.790 26.800 30.132792 # 2015-07-22 34.180 27.070 30.134375 # 2015-07-23 28.779 27.412 28.290918 # 2015-07-24 NaN NaN NaN # 2015-07-25 NaN NaN NaN d = xray.DataArray(agg.values.reshape(6, 1, 3), coords=[agg.index, [u'誰かの家'], [u'最高', u'最低', u'平均']], dims=['date', 'location', 'type']) d # <xray.DataArray (date: 6, location: 1, type: 3)> # array([[[ 28.374 , 26.45 , 27.48460417]], # [[ 33.79 , 26.8 , 30.13279167]], # [[ 34.18 , 27.07 , 30.134375 ]], # [[ 28.779 , 27.412 , 28.29091765]], # [[ nan, nan, nan]], # [[ nan, nan, nan]]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U4 u'\u8ab0\u304b\u306e\u5bb6' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
データの連結は xray.concat で可能。新しい場所のデータを追加 (連結) したいので、対象の次元として location を指定する。
da3 = xray.concat([da3, d], dim='location') da3.sel(location=u'誰かの家') # <xray.DataArray (date: 6, type: 3)> # array([[ 28.374 , 26.45 , 27.48460417], # [ 33.79 , 26.8 , 30.13279167], # [ 34.18 , 27.07 , 30.134375 ], # [ 28.779 , 27.412 , 28.29091765], # [ nan, nan, nan], # [ nan, nan, nan]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # location <U4 u'\u8ab0\u304b\u306e\u5bb6'
ほか、merge で結合もできる。
Dataset の利用
xray では、DataArray クラスを複数まとめて Dataset クラスとして扱うことができる。元となる DataArray は同じ次元でなくてもよい。
Dataset を作成するため、降水量、湿度 それぞれ 2 次元の DataArray を用意する。
# 降水量 precip = np.array([[0, np.nan, 0], [0, np.nan, np.nan], [0, 0, np.nan], [6.5, 1, 4.5], [30.0, 0, 7.0], [0, np.nan, np.nan]]) precip = xray.DataArray(precip, coords=[dates, locs], dims=['date', 'location']) precip # <xray.DataArray (date: 6, location: 3)> # array([[ 0. , nan, 0. ], # [ 0. , nan, nan], # [ 0. , 0. , nan], # [ 6.5, 1. , 4.5], # [ 30. , 0. , 7. ], # [ 0. , nan, nan]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 湿度 humid = np.array([[np.nan, 88, 75], [np.nan, 85, 65], [np.nan, 87, 61], [np.nan, 91, 80], [np.nan, 86, 83], [np.nan, 88, 80]]) humid = xray.DataArray(humid, coords=[dates, locs], dims=['date', 'location']) humid # <xray.DataArray (date: 6, location: 3)> # array([[ nan, 88., 75.], # [ nan, 85., 65.], # [ nan, 87., 61.], # [ nan, 91., 80.], # [ nan, 86., 83.], # [ nan, 88., 80.]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
これらと気温データをあわせて xray.Dataset を作成する。Dataset が持つ次元は Dimenstions, Coordinates に表示される。
Dataset に含まれる DataArray は Data variables 中に表示され、それぞれどの次元を含んでいるかがわかる。元データと同じく、気温は 3 次元、ほかは 2 次元のデータとなっている。
ds = xray.Dataset({'temperature': da3,
'precipitation': precip,
'humidity': humid})
ds
# <xray.Dataset>
# Dimensions: (date: 6, location: 4, type: 3)
# Coordinates:
# * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ...
# * location (location) object u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
# * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
# Data variables:
# precipitation (date, location) float64 0.0 nan 0.0 nan 0.0 nan nan nan ...
# temperature (date, location, type) float64 34.2 24.7 27.8 30.2 25.1 ...
# humidity (date, location) float64 nan 88.0 75.0 nan nan 85.0 65.0 ...
ds.data_vars
# Data variables:
# precipitation (date, location) float64 0.0 nan 0.0 nan 0.0 nan nan nan ...
# temperature (date, location, type) float64 34.2 24.7 27.8 30.2 25.1 ...
# humidity (date, location) float64 nan 88.0 75.0 nan nan 85.0 65.0 ...
Dataset からのデータ選択は、Dataset に含まれるすべての DataArray に対して行われる。DataArray とは異なり、次元は必ずラベルで指定する必要がある。
| 次元の指定 | 値の指定 | Dataset のメソッド |
|---|---|---|
| 位置 | 位置 | なし |
| 位置 | ラベル | なし |
| ラベル | 位置 | ds.isel(location=2), ds[dict(location=2)] |
| ラベル | ラベル | ds.sel(location=u'東京'), ds.loc[dict(location=u'東京')] |
# 東京 のデータを選択 ds.sel(location=u'東京') # <xray.Dataset> # Dimensions: (date: 6, type: 3) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u6771\u4eac' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # Data variables: # precipitation (date) float64 0.0 nan nan 4.5 7.0 nan # temperature (date, type) float64 33.5 25.8 28.8 34.9 25.4 29.4 32.8 ... # humidity (date) float64 75.0 65.0 61.0 80.0 83.0 80.0 # 東京 の 平均気温 を選択 ds.sel(location=u'東京', type=u'平均')['temperature'] # <xray.DataArray 'temperature' (date: 6)> # array([ 28.8, 29.4, 28.9, 27. , 28.1, 28.6]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # type <U2 u'\u5e73\u5747' # location object u'\u6771\u4eac' # 東京 の 湿度 を選択 ds.sel(location=u'東京')['humidity'] # <xray.DataArray 'humidity' (date: 6)> # array([ 75., 65., 61., 80., 83., 80.]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u6771\u4eac' # 対応する DataArray がない場合は NaN となる ds.sel(location=u'誰かの家') # <xray.Dataset> # Dimensions: (date: 6, type: 3) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u8ab0\u304b\u306e\u5bb6' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # Data variables: # precipitation (date) float64 nan nan nan nan nan nan # temperature (date, type) float64 28.37 26.45 27.48 33.79 26.8 30.13 ... # humidity (date) float64 nan nan nan nan nan nan
そのほかの操作も DataArray と同じようにできる。
# location ごとに期間中の最大値を計算 ds.groupby('location').max() # <xray.Dataset> # Dimensions: (location: 3) # Coordinates: # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # Data variables: # precipitation (location) float64 30.0 1.0 7.0 # temperature (location) float64 36.0 30.2 34.9 # humidity (location) float64 nan 91.0 83.0
pandas のデータ形式への変換
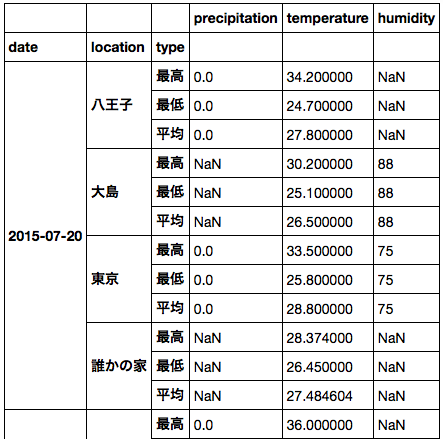
DataArray, Dataset はそれぞれ pandas のデータに変換できる。元データが 3 次元以上の場合、変換後の pandas のデータは MultiIndex を持つことになる。
また、上の例では 次元が異なる DataArray から Dataset を作成した。このとき、存在しない次元 ( ここでは type ) のデータはすべて同じ値でパディングされる。
ds.to_dataframe()
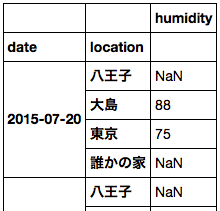
パディングしたくない場合は、個々の DataArray ごとに DataFrame に変換すればよい。
ds['humidity'].to_dataframe()
まとめ
xray を使えば 多次元のラベル付きデータを多次元のまま、pandas に近い方法で扱うことができる。
{rbokeh} で Bokeh を R から使いたい
はじめに
Bokeh は Python 以外にも R, Scala, Julia 用のパッケージを提供している。パッケージといっても Python の Bokeh と連携するものではなく、Bokeh がブラウザでのレンダリングに使っている Bokeh.js を各言語で扱えるようにするもののようだ。そのため、各パッケージはそれぞれ単体で利用できる。
うち、R 用のパッケージである {rbokeh} を使ってみたい。R には {htmlwidgets} を使った JavaScript 利用の可視化パッケージが多数提供されているが、数が多くて使い方が覚えられない。できれば {rbokeh} だけを使いたい。また、Pythonと見た目が揃えられると嬉しい。
インストール
現時点で CRAN には公開されていないため、GitHub からインストールする。現時点のバージョンは 0.2.3.2。
install.packages('htmlwidgets') install.packages('devtools') library(devtools) devtools::install_github('bokeh/rbokeh')
データの準備
サンプルとして {ggplot2} に含まれる mpg データを利用する。単に {rbokeh} を利用するだけなら {ggplot2} は不要。
library(ggplot2) library(rbokeh) dim(mpg) # [1] 234 11 head(mpg) # manufacturer model displ year cyl trans drv cty hwy fl class # 1 audi a4 1.8 1999 4 auto(l5) f 18 29 p compact # 2 audi a4 1.8 1999 4 manual(m5) f 21 29 p compact # 3 audi a4 2.0 2008 4 manual(m6) f 20 31 p compact # 4 audi a4 2.0 2008 4 auto(av) f 21 30 p compact # 5 audi a4 2.8 1999 6 auto(l5) f 16 26 p compact # 6 audi a4 2.8 1999 6 manual(m5) f 18 26 p compact
基本的な操作
Python の Bokeh は matplotlib を連想させる API を持っているが、 {rbokeh} は別の API ( {ggplot2} や {ggvis} に近い ) を持つ。そのため、API は言語ごとに覚える必要がある。
mpg データの displ 列と hwy 列を利用して散布図を描画する。
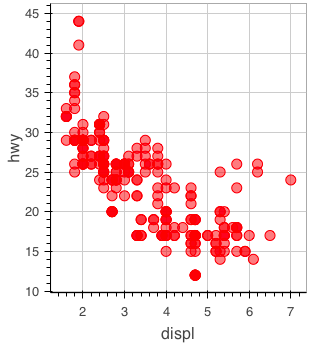
figure() %>% ly_points(x = displ, y = hwy, data = mpg)

加えて、 class 列で色分けする。サイズの変更など、利用できるオプションは help(ly_points) で確認できる。
figure() %>% ly_points(x = displ, y = hwy, color = class, data = mpg)

色名は文字列で指定することもできる。
figure() %>% ly_points(x = displ, y = hwy, color = 'red', data = mpg)
NSE と SE
{rbokeh} では、{ggplot2} のように NSE ( Non-standard evaluation )と SE ( Standard evaluation ) 用の関数を明示的に分けていない。そのため、一部のオプションは以下のようにSEでも動作する。
# scatter figure() %>% ly_points(x = 'displ', y = 'hwy', data = mpg) # 出力省略
が、 SE では動作しないものもあるため、現時点では NSE を使ったほうがよさそうだ。
# scatter figure() %>% ly_points(x = 'displ', y = 'hwy', color = 'class', data = mpg) # 以下にエラー nchar(opts[[fld]]) : 'nchar()' は文字ベクトルを要求します
平滑化 / 回帰直線の描画
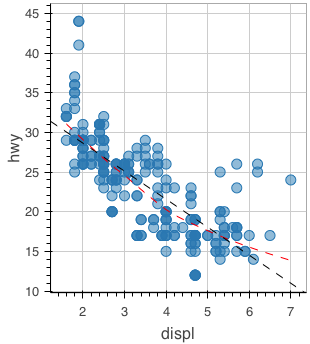
R のモデルを関数に渡すことで描画できる。lowess に対しては ly_lines を、lm に対しては ly_abline を用いる。この使い分けはちょっと覚えにくい。
figure() %>% ly_points(x = displ, y = hwy, data = mpg) %>% ly_lines(lowess(mpg$displ, mpg$hwy), color = "red", type = 2) %>% ly_abline(lm(hwy ~ displ, data = mpg), type = 2)
サブプロット ( facet ) の描画
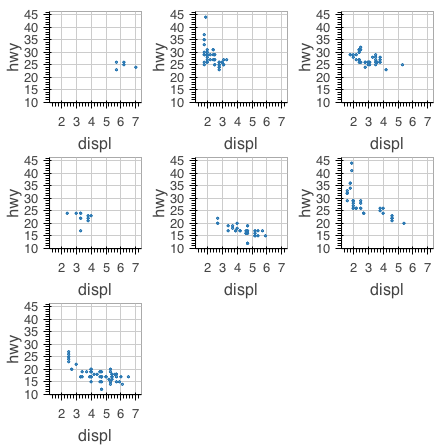
{htmlwidgets} ブログ中の {rbokeh} 紹介記事 では、サブプロットを lapply + {pipeR} を使って描画する例が記載されている。自分は {dplyr} 好きなので {dplyr} でやりたい。
library(dplyr) mpg %>% dplyr::group_by(class) %>% dplyr::do(dummy = ly_points(figure(width = 200, height = 200), x = displ, y = hwy, data = ., size = 2)) %>% { as.list(.[['dummy']]) } %>% grid_plot(nrow = 3, ncol = 3, same_axes = TRUE )
様々なプロット
以降、{rbokeh} で描画できるプロットを例示する。
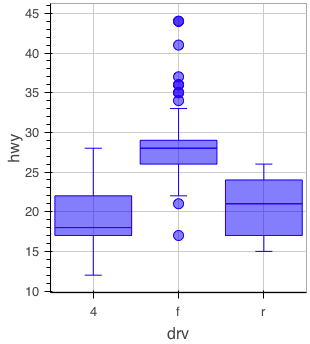
箱ひげ図
figure() %>% ly_boxplot(x = drv, y = hwy, data = mpg)
ヒストグラム
figure() %>% ly_hist(x = hwy, data = mpg)
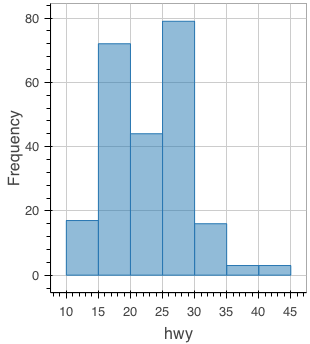
ヒストグラムをグループ別に描画するには少し手間が必要。グループ別に塗り分けるためのカラーパレットを用意し、Reduce で各グループ別のヒストグラムを追加する。
library(scales) colors <- scales::hue_pal()(length(levels(mpg$class))) colors <- setNames(colors, levels(mpg$class)) colors # 2seater compact midsize minivan pickup subcompact suv # "#F8766D" "#C49A00" "#53B400" "#00C094" "#00B6EB" "#A58AFF" "#FB61D7" ghist <- function(fig, group) { ly_hist(fig, x = hwy, data = dplyr::filter(mpg, class == group), breaks = seq(10, 45, 5), color = colors[[group]]) } Reduce(ghist, levels(mpg$class), figure())
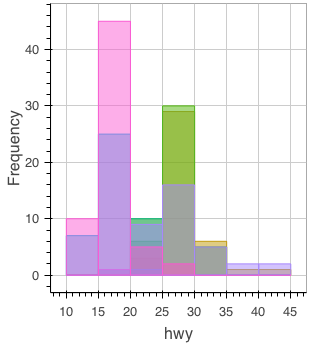
カーネル密度推定のプロット
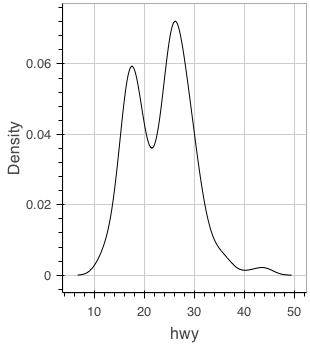
グループ分けしたい場合はヒストグラム同様の処理が必要。
figure() %>% ly_density(x = hwy, data = mpg)
折れ線グラフ
df <- data.frame(x = c(1, 2, 3), y = c(4, 2, 5)) figure() %>% ly_lines(x = x, y = y, data = df)

地図
地図は {maps} パッケージのデータを利用してプロットができる。
library(maps) figure() %>% ly_map("state", color = "gray")

また、 Google Maps を利用することもできる。
gmap(lat = 35.684, lng = 139.753, zoom = 15, map_type = 'terrain')
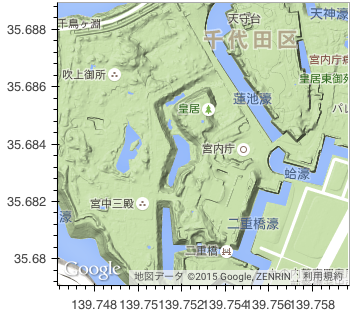
現在 (簡単には) できないもの
R でよく使う種類のプロットを優先しているせいか、以下のような基本的なプロットにはまだ対応していないようだ。
| 種類 | GitHub Issue | 備考 |
|---|---|---|
| 棒グラフ | #8 | 頑張れば ly_hist で描ける。 |
| エリアプロット | #10 | |
| ヒートマップ | #86 | ly_hexbin はある。ly_image で近いことはできるが、軸のラベル付けが手間。 |
| 円/ドーナツ |
まとめ
{rbokeh}、上に挙げた基本的なプロットが使えるようになれば実用できそうな感じだ。Bokeh.js 側には主要な可視化が揃っているため、他のパッケージと比べると利用範囲が広いこと、見た目を複数言語で揃えられるのがメリットかなと思う。