Python でパイプ演算子を使いたい <2>
ネタ記事です。/ This is a joke post which makes no practical sense.
過去にこんなエントリを書いた。
R では パイプ演算子 %>% を使って連続した処理を記述できる。式に含まれる
x, y, z は非標準評価 (NSE) によって data.frame の列として解決される。
# R (magrittr + dplyr) df %>% mutate(x = y + z) %>% group_by(x) %>% summarize_each(funs(sum))
Python (pandas) ではほぼ同じ処理をメソッドチェインを使って書ける。チェインとパイプ演算子でどちらが読みやすいかは好みの問題だと思うものの、式の中に 何度も df が出てくるのはちょっとすっきりしない。
# Python (pandas) df.assign(x=df['y'] + df['z']).groupby('x').sum()
上の式をこんな風に書けるとうれしい。
df >> assign(x=y+z) >> groupby(x) >> sum()
前のエントリでは マジックメソッドと関数定義で近いことをやったが、以下のような課題がある。
- パイプ演算子で処理したいメソッドを、それぞれ関数として別に定義しなければならない
- R の非標準評価のようなことはできない (ある変数を別の環境で評価することができない)
前者は手間をかければなんとかなるが、後者については NameError が発生してしまうためどうしようもない。
df.assign(x=y+z)
# NameError: name 'y' is not defined
上のような表現を式に含めるには、 lambda を使って無名関数にすればよい。lambda を使うと、下のように定義されていない関数 / 変数を式に含めることができる。
lambda: df >> assign(x=y+z) # <function <lambda> at 0x102310620>
当然、この無名関数をそのまま評価すると NameError が発生する。定義した無名関数の中身を調べて、妥当な処理に置き換えてやればよい。
Python 標準では ast モジュールを使って 抽象構文木 (AST) の探索や置換ができる。が、今回はそこまで複雑なことをやるわけではないので、バイトコードを直接書き換えられるとうれしい。
バイトコードの置換について簡単な方法がないか調べたところ CodeTransformer というパッケージを見つけた。今回はこれを使いたい。
CodeTransformerとは
バイトコードに対するパターンマッチ⇨置換が簡単にできる。詳しい使い方はドキュメントを。
まずは関数がバイトコードとしてどのように表現されているのか確かめたい。これは標準の dis モジュールを使うのが簡単。
import dis dis.dis(lambda x: x + 2) # 1 0 LOAD_FAST 0 (x) # 3 LOAD_CONST 1 (2) # 6 BINARY_ADD # 7 RETURN_VALUE
CodeTransformer を使うと適当なパターンにマッチするバイトコードを置換することができる。ドキュメントの例にある通り、バイトコード中の加算 (BINARY_ADD) を乗算 (BINARY_MULTIPLY) に置き換える CodeTransformer は以下のようになる。
import codetransformer codetransformer.__version__ # '0.6.0' from codetransformer import CodeTransformer, pattern from codetransformer.instructions import BINARY_ADD, BINARY_MULTIPLY class Multiply(CodeTransformer): @pattern(BINARY_ADD) def _add2mul(self, add_instr): yield BINARY_MULTIPLY().steal(add_instr)
pattern デコレータで指定したバイトコードにマッチした時、対応したメソッドが呼び出される。呼び出されるメソッドから置換後のバイトコードを返せばよい。
作成した CodeTransformer を使って関数を置換する。
mul = Multiply()(lambda x: x + 2) mul(5) # 10
Multiply によって置き換えられた後のバイトコードを確認すると、BINARY_ADD が BINARY_MULTIPLY に置換されていることが確かめられる。
dis.dis(mul) # 1 0 LOAD_FAST 0 (x) # 3 LOAD_CONST 0 (2) # 6 BINARY_MULTIPLY # 7 RETURN_VALUE
パイプ演算子を使いたい
ここから、pandas と組み合わせて使ってみる。まずはデータを準備する。
import pandas as pd df = pd.DataFrame({'A': [1, 2, 3, 4, 5, 6], 'B': list('ABABAB'), 'C': [1, 2, 3, 1, 2, 3]}) df # A B C # 0 1 A 1 # 1 2 B 2 # 2 3 A 3 # 3 4 B 1 # 4 5 A 2 # 5 6 B 3
パイプ演算子を処理するには、lambda: df >> head() のバイトコードを lambda: df.head() のバイトコードに置換すればよい。置換前、置換後のバイトコードをそれぞれ確認すると、
# 置換前 dis.dis(lambda: df >> head()) # 1 0 LOAD_GLOBAL 0 (df) # 3 LOAD_GLOBAL 1 (head) # 6 CALL_FUNCTION 0 (0 positional, 0 keyword pair) # 9 BINARY_RSHIFT # 10 RETURN_VALUE # 置換後 dis.dis(lambda: df.head()) # 1 0 LOAD_GLOBAL 0 (df) # 3 LOAD_ATTR 1 (head) # 6 CALL_FUNCTION 0 (0 positional, 0 keyword pair) # 9 RETURN_VALUE
比べてみると、以下のような変換を行えばよさそうだ。
- 置換前のパターン:
LOAD_GLOBAL → CALL_FUNCTION → BINARY_RSHIFT - 置換後のパターン:
LOAD_ATTR → CALL_FUNCTION
連続したパターンとマッチさせるには、pattern デコレータと対応するメソッドに複数の引数を指定すればよい。
from codetransformer.instructions import BINARY_RSHIFT, LOAD_GLOBAL, LOAD_ATTR, CALL_FUNCTION class Pipify(CodeTransformer): @pattern(LOAD_GLOBAL, CALL_FUNCTION, BINARY_RSHIFT) def _pipe(self, load, call, rshift): # LOAD_GLOBAL を LOAD_ATTR に置換 yield LOAD_ATTR(load.arg) yield call # BINARY_SHIFT は無視
これにパイプ演算を含む無名関数を渡すと、期待通り動いているように見える。
Pipify()(lambda: df >> head())() # A B C # 0 1 A 1 # 1 2 B 2 # 2 3 A 3 # 3 4 B 1 # 4 5 A 2 dis.dis(Pipify()(lambda: df >> head())) # 1 0 LOAD_GLOBAL 0 (df) # 3 LOAD_ATTR 1 (head) # 6 CALL_FUNCTION 0 (0 positional, 0 keyword pair) # 9 RETURN_VALUE
もっとも、上のクラスは非常に単純な条件でしか動かない。たとえば、パイプで接続される関数(メソッド)に引数がある場合、patternの定義とマッチしなくなるため置換が行われない。
# NG Pipify()(lambda: df >> head(2))() # NameError: name 'head' is not defined dis.dis(lambda: df >> head(2)) # 1 0 LOAD_GLOBAL 0 (df) # 3 LOAD_GLOBAL 1 (head) # 6 LOAD_CONST 1 (2) # 9 CALL_FUNCTION 1 (1 positional, 0 keyword pair) # 12 BINARY_RSHIFT # 13 RETURN_VALUE
もう少しマシな CodeTransformer の定義を考える。パイプ演算子の記法から、 LOAD_GLOBAL から次の BINARY_RSHIFT までを 1 単位とした pattern 定義でマッチすればよいような気がする。
CodeTransformer の定義は以下のようになる。(~BINARY_RSHIFT)[plus] は BINARY_SHIFT 以外の任意のコードの繰り返しとマッチする。
from codetransformer import plus class Pipify(CodeTransformer): @pattern(LOAD_GLOBAL, (~BINARY_RSHIFT)[plus], BINARY_RSHIFT) def _pipe(self, load, *args): # メソッド呼び出しの処理 if hasattr(self, 'first_load'): yield LOAD_ATTR(load.arg) else: # 最初の LOAD_GLOBAL は置き換えない self.first_load = load yield load # メソッドへの引数の処理 for arg in args[:-1]: if isinstance(arg, LOAD_GLOBAL): # 関数の一番最初の LOAD_GLOBAL から解決 # この定義では、パイプ演算中に更新されたデータやグローバル変数には # アクセスできない yield self.first_load yield LOAD_ATTR(arg.arg) else: yield arg # 末尾は BINARY_RSHIFT なので無視
コメントで "メソッドへの引数の処理" とある部分は、 (記載のとおり限定的だが) R の非標準評価に近い処理になる。
この定義を使うと、上で失敗したメソッド呼び出しも処理できる。
Pipify()(lambda: df >> head(2))() # A B C # 0 1 A 1 # 1 2 B 2 dis.dis(Pipify()(lambda: df >> head(2))) # 1 0 LOAD_GLOBAL 0 (df) # 3 LOAD_GLOBAL 0 (df) # 6 LOAD_ATTR 1 (head) # 9 LOAD_CONST 0 (2) # 12 CALL_FUNCTION 1 (1 positional, 0 keyword pair) # 15 RETURN_VALUE
いちいち Pipify()(lambda: df >> ...)() と書くのは面倒なので、パイプ演算を開始する関数 p を定義する。この関数を使うとパイプ演算は p(lambda: df >> ...) のように書ける。
def p(expr): return Pipify()(expr)()
以下、いくつかのパターンを試す。それぞれ、冒頭にコメントとして記載した処理と同じ結果になる。
# df.head() p(lambda: df >> head()) # A B C # 0 1 A 1 # 1 2 B 2 # 2 3 A 3 # 3 4 B 1 # 4 5 A 2 # df.head(2) p(lambda: df >> head(2)) # A B C # 0 1 A 1 # 1 2 B 2
パイプ演算中の B は NameError とはならず df.B として解決される (非標準評価もどき)。
# df.groupby(df.B).sum() p(lambda: df >> groupby(B) >> sum()) # A C # B # A 9 6 # B 12 6
もう少し複雑な処理もできる。.assign で df.A + df.C の結果からなる X 列を新規で作成し、B 列の値ごとに集約して合計を取る。
# df.assign(X=df.A+df.C).groupby(df.B).sum() p(lambda: df >> assign(X=A+C) >> groupby(B) >> sum()) # A C X # B # A 9 6 15 # B 12 6 18
改行を含む場合は括弧で囲む。
p(lambda: (df >> assign(X=A+C) >> groupby(B) >> sum())) # 略
まとめ
CodeTransformerでバイトコードを置換することで、パイプ演算のような処理が書ける。また、非標準評価に近いこともできる。- R を使ったほうがいい。
PyConJP 2016: pandasでの時系列処理についてお話させていただきました
21日、22日と PyCon JP に参加させていただきました。ご参加いただいた皆様、スタッフの皆様ありがとうございました。資料はこちらになります。
pandas による時系列データ処理
pandas を使った時系列データの前処理と、statsmodels での時系列モデリングの触りをご紹介しました。
時系列モデルの考え方については全く説明していないので、以下書籍などをご参照ください。

経済・ファイナンスデータの計量時系列分析 (統計ライブラリー)
- 作者: 沖本竜義
- 出版社/メーカー: 朝倉書店
- 発売日: 2010/02/01
- メディア: 単行本
- 購入: 4人 クリック: 101回
- この商品を含むブログ (6件) を見る
元ネタ
以下のエントリをベースに新しい内容を追加しています。
時系列モデルを含む Python パッケージ
トーク中では ARIMA などの時系列モデルを含むパッケージとして statsmodels についてご説明、PyFlux をご紹介しました。一方 変化点検知や異常検知では、広く使われている Python パッケージはありません。
というわけで、作りました。現状、以下の2手法が実装されています。適当に手法追加しつつ、そのうちblogも書きます。
- 累積和法による変化点検知: R の
{changepoint}の実装と同一 - 成分分解 + Generalized ESD test による異常検知: R の
{AnomalyDetection}の実装に近いもの (同じではない)
補足 statsmodels v0.9ではマルコフ転換モデルが実装予定。また、skyline という異常検知アプリはあります。
Python pandas 欠損値/外れ値/離散化の処理
データの前処理にはいくつかの工程がある。書籍「データ分析プロセス」には 欠損など 前処理に必要なデータ特性の考慮とその対処方法が詳しく記載されている。
が、書籍のサンプルは R なので、Python でどうやればよいかよく分からない。同じことを pandas でやりたい。

- 作者: 福島真太朗,金明哲
- 出版社/メーカー: 共立出版
- 発売日: 2015/06/25
- メディア: 単行本
- この商品を含むブログ (2件) を見る
とはいえ、pandas 自身は統計的 / 機械学習的な前処理手法は持っていない。また Python には R と比べると統計的な前処理手法のパッケージは少なく、自分で実装しないと使えない方法も多い。ここではそういった方法は省略し、pandas でできる前処理 / 可視化を中心に書く。
また、方法自体の説明は記載しないので、詳細を知りたい方は 「データ分析プロセス」を読んでください。
データの要約
import numpy as np import pandas as pd pd.__version__ # u'0.17.1' import matplotlib.pyplot as plt import seaborn as sns
データの特徴をつかむため、要約統計量や相関係数が見たい。ここでは 「データ分析プロセス」と同じく Iris データ (scikit-learn に含まれているもの / 書籍とは少し値が違う) を例として使う。
import sklearn.datasets as datasets iris_data = datasets.load_iris() iris = pd.DataFrame(iris_data.data, columns=iris_data.feature_names) iris['species'] = iris_data.target iris.head()

変数の要約 (要約統計量)
pandas での要約統計量の表示は DataFrame.describe。5 列目
"species" も数値型だが、カテゴリ変数のため除外する。
iris.iloc[:, :4].describe()

"species" についてはラベルごとの頻度が見たいので Series.value_counts で集計する。
iris['species'].value_counts() # 2 50 # 1 50 # 0 50 # Name: species, dtype: int64
2 変数の関係 (相関係数と散布図行列)
また、相関係数の表示は DataFrame.corr。
iris.iloc[:, :4].corr()

散布図行列を描くには seaborn.pairplot。"species" に応じて色分けして描画する。
sns.pairplot(iris, hue='species');

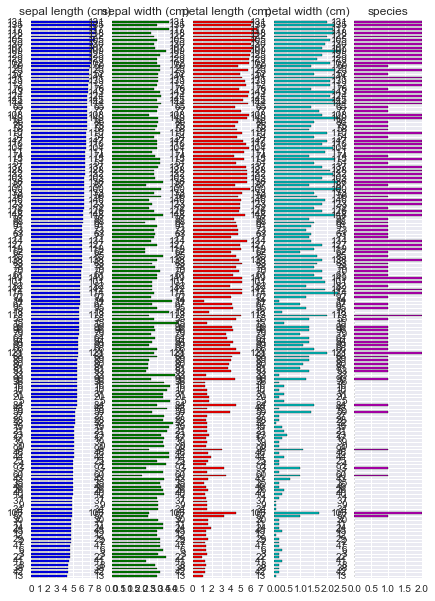
また、R には {tabplot} という data.frame 可視化のためのパッケージがある。これに近い出力は pandas でもかんたんに得られる。
(iris.sort_values('sepal length (cm)'). plot.barh(subplots=True, layout=(1, 5), sharex=False, legend=False));
欠損値
「データ分析プロセス」で使われているサンプルデータ employee_IQ_JP.csv を使う。ファイルは出版社のサポートページ からダウンロードできる。
データは知能指数 "IQ" と業務成果 "JobPerformance" 2 つの変数の関係を示している。3 列目以降は "JobPerformance" が以下いずれかのパターンで欠損した場合の例を示している。
| 欠損発生のパターン | 概要 |
|---|---|
| MCAR | ランダムに欠損している ( 欠損は "IQ" や "JobPerformance" の値に関係しない ) |
| MAR | 他の変数の値と関係して欠損している ( "IQ" が低いと "JobPerformance" の欠損が多い ) |
| MNAR | 欠損が発生しているデータ自身と関係して欠損している ( "JobPerformance" の真の値が低いと "JobPerformance" の欠損が多い ) |
df = pd.read_csv('employee_IQ_JP.csv')
df.head()

欠損パターンの可視化
欠損がそれぞれのパターンで発生した場合に、真の値 "JobPerformance" のうち欠損値となった箇所を 赤三角 "▲" で描く。
fig, axes = plt.subplots(1, 3) fig.tight_layout(w_pad=2.0) for col, ax in zip(['MCAR', 'MAR', 'MNAR'], axes): indexer = df[col].isnull() df[indexer].plot.scatter(x='IQ', y='JobPerformance', marker='^', color='red', label='missing', ax=ax) df[~indexer].plot.scatter(x='IQ', y='JobPerformance', ax=ax) ax.set_title(col)

次に、上よりもカラム数が多いサンプルデータを使って欠損のパターンを可視化する例を示す。R の {mice} パッケージから、nhanes データセットを CSV に出力し、pandas で読み込む。
nhances = pd.read_csv('nhanes.csv', index_col=0) nhances.head()

上の通り複数の変数で欠損が発生している。欠損がどのように発生しているかを調べるには以下のように集計すればよい。
missing = nhances.copy() # 欠損している場合に True とする missing = missing.apply(pd.isnull, axis=0) missing['count'] = 1 missing.groupby(['age', 'bmi', 'hyp', 'chl']).sum()

この結果から以下のことがわかる。
- 欠損がない (全て
False) レコードは 13件 ) - "chl" のみ欠損している ( "chl"のみ
True) レコードは 3 件 - 以下略
また、変数別に欠損しているレコード数を調べるには sum を取ればよい。
missing[['age', 'bmi', 'hyp', 'chl']].sum() # age 0 # bmi 9 # hyp 8 # chl 10 # dtype: int64
また、ある 2 つの変数 "bmi", "hyp" を選んで、欠損がどのように発生しているかを調べたい。DataFrame.pivot_table で集計する。
missing.pivot_table(index='hyp', columns='bmi', values='count', aggfunc='sum')

この結果から、
- 2 変数とも欠損なし: 16 件
- "bmi" のみ欠損: 1 件
- 以下略
上で調べた欠損値の発生状況をプロットすると以下のようになる。
- 左側: 各変数が欠損しているレコード数
- 右側: 欠損している変数の組み合わせごとのレコード数
fig, axes = plt.subplots(1, 3) missing[['age', 'bmi', 'hyp', 'chl']].sum().plot.bar(ax=axes[0]) missing.groupby(['age', 'bmi', 'hyp', 'chl']).sum().plot.barh(ax=axes[2]) axes[1].set_visible(False);

また、変数 "age" について、自身以外の変数 "bmi", "hyp", "chl" がそれぞれが欠損した / しなかった場合の分布を箱ヒゲ図で描く。 "age" の値が 他の変数の欠損とどのような関係にあるかがわかる。
missing['age'] = nhances['age'] fig, axes = plt.subplots(1, 4) fig.tight_layout(w_pad=3.0) sns.boxplot(data=missing, y='age', ax=axes[0]) sns.boxplot(data=missing, y='age', x='bmi', ax=axes[1]) sns.boxplot(data=missing, y='age', x='hyp', ax=axes[2]) sns.boxplot(data=missing, y='age', x='chl', ax=axes[3]);

欠損に対する処理
欠損値に対する対応にはいくつかの方法がある。うち、pandas, scikit-learn でできる方法を記載する。
| 方法 | 概要 |
|---|---|
| リストワイズ法 | 欠損レコードを除去 |
| ペアワイズ法 | 相関係数など 2 変数を用いて計算を行う際に、対象の変数が 欠損している場合に計算対象から除外 |
| 平均代入法 | 欠損を持つ変数の平均値を補完 |
| 回帰代入法 | 欠損を持つ変数の値を 回帰式をもとに補完 |
完全情報最尤推定、多重代入は Python にはなさそうなので、使うなら R のパッケージを呼び出すしかないと思う。
リストワイズ法
リストワイズ法では欠損を除去すれば良いため DataFrame.dropna でできる。
nhances.shape # (25, 4) nhances.dropna(subset=['bmi']).shape # (16, 4) nhances.dropna(subset=['bmi', 'chl'], how='any').shape # (13, 4)
ペアワイズ法
少し手間がかかるが、対象となる 2 変数について欠損しているレコードを除去 -> 計算を繰り返せばできる。
平均代入法
平均代入のように代表値で埋める場合は DataFrame.fillna。
nhances['bmi'] # 1 NaN # 2 22.7 # 3 NaN # 4 NaN # ... # 24 24.9 # 25 27.4 # Name: bmi, dtype: float64 nhances['bmi'].fillna(nhances['bmi'].mean()) # 1 26.5625 # 2 22.7000 # 3 26.5625 # 4 26.5625 # ... # 24 24.9000 # 25 27.4000 # Name: bmi, dtype: float64
回帰代入法
回帰代入では欠損が発生している変数と 欠損の発生に影響している変数とで回帰式を作り、作られた回帰式を使って欠損を補完する。欠損は MAR で発生していないとダメ。サンプルデータとしては 再び employee_IQ_JP.csv を使う。
回帰には scikit-learn を使う。当たり前だが補間された値は回帰直線 (灰色破線) 上に乗る。
import sklearn.linear_model as lm reg = lm.LinearRegression() indexer = df['MAR'].isnull() reg.fit(df.loc[~indexer, ['IQ']], df.loc[~indexer, 'MAR']) predicted = reg.predict(df.loc[indexer, ['IQ']]) df.loc[indexer, 'MAR'] = predicted # プロット ax = df[indexer].plot.scatter(x='IQ', y='MAR', marker='^', color='red', label='missing'); ax = df[~indexer].plot.scatter(x='IQ', y='MAR', ax=ax); x = np.linspace(*ax.get_xlim()) ax.plot(x, reg.coef_[0] * x + reg.intercept_, color='gray', linestyle='dashed')

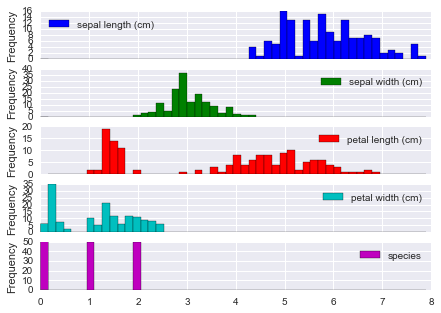
外れ値
外れ値をみるにはまずデータの分布 / 箱ヒゲ図を描くのがかんたん。
iris.plot(kind='hist', bins=50, subplots=True);
四分位範囲での検出
seaborn.boxplot では 外れ値はダイヤ "♦︎" で描画される。外れ値とみなす閾値は whis オプションを利用して指定できる。既定は 1.5 で、四分位範囲 (IQR) = 第3四分位 - 第1四分位 の 1.5 倍を超えるレコードが外れ値となる。
ここでは "species" のラベルごとに、各変数の箱ヒゲ図を描く。
fig, axes = plt.subplots(3, 1) for i, (n, g) in enumerate(iris.groupby('species')): sns.boxplot(data=g.iloc[:, :4], ax=axes[i]) axes[i].set_ylabel(n)

「データ分析プロセス」に記載されているその他の方法のうち、LOF (Local Outlier Factor) には Python のパッケージがあるが、メンテされているか謎だ。
また、scikit-learn の 1 クラス SVM や ガウス過程 を使う方法もある。これらは 機械学習プロフェッショナルシリーズ「状態変化と異常検知」に記載がある。

- 作者: 井手剛,杉山将
- 出版社/メーカー: 講談社
- 発売日: 2015/08/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (1件) を見る
離散化
以下 2 つの方法については pandas でできる。
| 方法 | 概要 |
|---|---|
| 等間隔区間 (EWD) | 対象のカラムの値を等間隔の区分で分割する |
| 等頻度区間 (EFD) | 分割した区分に同程度の数のレコードが含まれるように分割する |
等間隔区間による離散化
等間隔区間による離散化は pd.cut。どのように分割されたかは categories として表示される。
pd.cut(iris['sepal length (cm)'], 5) # 0 (5.02, 5.74] # 1 (4.296, 5.02] # ... # 148 (5.74, 6.46] # 149 (5.74, 6.46] # Name: sepal length (cm), dtype: category # Categories (5, object): [(4.296, 5.02] < (5.02, 5.74] < (5.74, 6.46] < (6.46, 7.18] < (7.18, 7.9]]
区分ごとのレコード数を数えるには Series.value_counts。結果を Series.sort_index して区分の順番に並べている。
pd.cut(iris['sepal length (cm)'], 5).value_counts().sort_index() # (4.296, 5.02] 32 # (5.02, 5.74] 41 # (5.74, 6.46] 42 # (6.46, 7.18] 24 # (7.18, 7.9] 11 # dtype: int64
等頻度区間による離散化
等頻度区間による離散化は pd.qcut。
pd.qcut(iris['sepal length (cm)'], 5) # 0 (5, 5.6] # 1 [4.3, 5] # ... # 148 (6.1, 6.52] # 149 (5.6, 6.1] # Name: sepal length (cm), dtype: category # Categories (5, object): [[4.3, 5] < (5, 5.6] < (5.6, 6.1] < (6.1, 6.52] < (6.52, 7.9]] pd.qcut(iris['sepal length (cm)'], 5).value_counts().sort_index() # [4.3, 5] 32 # (5, 5.6] 33 # (5.6, 6.1] 30 # (6.1, 6.52] 25 # (6.52, 7.9] 30 # dtype: int64
「データ分析プロセス」に記載されているその他の方法のうち、最小記述長原理 (MDLP) での離散化は scikit-learn に PR が上がっている。
| 方法 | Python パッケージ / リンク |
|---|---|
| 最小記述長原理 (MDLP) | Discretization using Fayyad's MDLP stop criterion by hlin117 · Pull Request #4801 · scikit-learn/scikit-learn · GitHub |
まとめ
書籍「データ分析プロセス」の流れに沿って、欠損値/外れ値/離散化の処理を、pandas で行う方法を記載した。
上で記載した方法 = Python でできる方法は書籍の内容のうち比較的 簡単な方法だけだ。より網羅的に知りたい方は書籍を読んでいただくのがいい。R ユーザに限らずおすすめ。

- 作者: 福島真太朗,金明哲
- 出版社/メーカー: 共立出版
- 発売日: 2015/06/25
- メディア: 単行本
- この商品を含むブログ (2件) を見る
Python pandas で e-Stat のデータを取得したい
e-Stat とは
"「政府統計の総合窓口(e-Stat)」は、各府省が公表する統計データを一つにまとめ、統計データの検索をはじめとした、さまざまな機能を備えた政府統計のポータルサイト" だそうだ。このデータを pandas で読めるとうれしい...ということで対応した。
インストール
$ pip install japandas
パッケージのインポート
import numpy as np np.__version__ # '1.10.2' import pandas as pd pd.__version__ # u'0.17.1' import japandas as jpd jpd.__version__ # '0.2.0'
アプリケーション ID の取得
e-Stat を利用するには 利用登録とアプリケーション ID の取得が必要。利用ガイドに沿って登録する。
データの取得
japandas を利用してデータを取得する。データの取得は以下 2 ステップで行う
- "政府統計コード" を利用して、統計調査に含まれる統計表 ( 実データ ) の一覧とその ID ( 統計表 ID ) を取得する。
- 取得した統計表 ID を利用して、実データを取得する
1. 統計表一覧の取得
e-Stat 提供データ一覧 に含まれる統計調査のうち、今回は 00200564 全国消費実態調査 を利用する。
jpd.DataReader で ID "00200564" のデータを取得すると、以下のような DataFrame が返ってくる。各カラムの詳細は e-Stat API 仕様 に記載されている。
key = "Your application ID" dlist = jpd.DataReader("00200564", 'estat', appid=key) dlist

一つの "統計表題名及び表番号" は "調査年月" が異なる複数のデータを持つことがある。値をユニークにした方が中身を確認しやすい。
tables = dlist[u'統計表題名及び表番号'].value_counts().to_frame()
tables

ここでは "平成26年全国消費実態調査 > 全国 > 品目及び購入先・購入地域に関する結果 > 単身世帯" の "男女,年齢階級,購入形態,品目別1世帯当たり1か月間の支出" のデータを取得したい。
この時点では 正確な "統計表題名及び表番号" がわからないため、まずはそれらしい文字列でレコードを抽出する。
indexer = tables.index.str.contains(u'男女,年齢階級,購入形態,品目別1世帯当たり1か月間の支出') indexer # array([False, False, False, ..., False, False, False], dtype=bool) tables[indexer]

上の結果から 正確な "統計表題名及び表番号" が得られるため、元データから対象のレコードが抽出できる。
table = tables[indexer].index[0] table # [単身世帯]フロー編第149表 男女,年齢階級,購入形態,品目別1世帯当たり1か月間の支出 target = dlist[dlist[u'統計表題名及び表番号'] == table] target

2. 実データの取得
- で調べた "統計表 ID" (
"0003109612") をjpd.DataReaderに渡せばよい。
df = jpd.DataReader("0003109612", 'estat', appid=key) # 略
が、いちいち文字列を抽出したり再入力するのは面倒だ。そんな時は、上の結果 ( 取得対象の "統計表 ID" を含む DataFrame) をそのまま渡してもよい。複数のレコードがある場合は全データを連結して返す。
df = jpd.DataReader(target, 'estat', appid=key)
df

出典:「平成26年全国消費実態調査調査結果」(総務省統計局)
取得したデータを集計してみる。各属性に対応する値は "value" カラムに含まれている。まず、カラム名を簡潔なものに変更する。
df.columns =[u'value', u'世帯区分', u'品目分類表', u'地域', u'表章項目', u'男女', u'年齢階級', u'購入形態']
また、dtypes を見ると全ての列が object 型になっている。通常、"value" には数値が入るため、jpd.DataReader は "value" が数値に変換できる場合は自動で変換するのだが、このデータでは何らかの理由で失敗しているようだ。
df.dtypes # value object # 世帯区分 object # 品目分類表 object # 地域 object # 表章項目 object # 男女 object # 年齢階級 object # 購入形態 object # dtype: object
pd.to_numeric で数値に変換しようとすると ValueError が発生する。データを見ると "value" にハイフン "-" がいくつか使われているようだ (やめてくれ...)。
文字列処理してもよいが、今回は特に何もしなくても以降の処理で解決されるため、とりあえずそのままにしておく。
1/9 補足 v0.2.1 では "-" を欠損として扱うように修正済み。
pd.to_numeric(df['value']) # ValueError: Unable to parse string df['value'].str.isnumeric() # 時間軸(年次) # 2014-01-01 True # 2014-01-01 True # 2014-01-01 True # 2014-01-01 False # ... # 2014-01-01 False # 2014-01-01 False # 2014-01-01 False # 2014-01-01 False # Name: value, dtype: bool
"品目分類表" の値をみるとかなり細かい項目に分けられていることがわかる。
統計調査に利用された調査票 を参照すると、調査形式は家計簿のようなフォーマットに任意の品名を書き込むもののようだ。品目分類を被調査者が記入する欄はなく、集計時に家計簿を品目分類表に従って分類しているのだろう。
df[u'品目分類表'].unique() # array([u'集計世帯数', # u'世帯数分布(抽出率調整)', # u'世帯数分布(抽出率調整)(1万分比)', # ..., u'仕送り金', # u'国内遊学仕送り金', # u'他の仕送り金'], dtype=object)
データから "すし" を含むレコードを抽出してみる。...弁当とは? この調査票には詳細が記載されていないようだが、他の調査を見ると スーパーで買うパックの寿司を指しているものと思う (おにぎりは別項目)。
filtered = df[df[u'品目分類表'].str.contains(u'すし')] filtered

抽出されたレコードの
"value" には 数値として不正な文字列は含まれないため、pd.to_numeric で数値に変換できる。
filtered['value'] = pd.to_numeric(filtered['value']) filtered.dtypes # value int64 # 世帯区分 object # 品目分類表 object # 地域 object # 表章項目 object # 男女 object # 年齢階級 object # 購入形態 object # dtype: object
Series.nunique で各列に含まれるユニークな値の数を調べる。"男女", "年齢階級", "購入形態" でレコードが分かれているため、それら 3 つをキーにして集計してやればよい。
filtered.apply(lambda x: x.nunique()) # value 76 # 世帯区分 1 # 品目分類表 1 # 地域 1 # 表章項目 1 # 男女 3 # 年齢階級 8 # 購入形態 4 # dtype: int64 sushi = pd.pivot_table(filtered, index=[u'男女', u'年齢階級'], columns=u'購入形態', values='value', aggfunc='sum') sushi

"購入形態" (支払い方法) には興味がないので、"合計" の値だけを抽出してプロットする。
60 代 男性 単身者 は "すし(弁当)" への消費金額が比較的多いようだ。金額的に月 1 〜 2 回買っている感じだろうか。また女性も一部男性と比べ高い。
sushi = sushi[[u'合計']] sushi.plot.bar(ylim=(0, 1000))

追加データでの確認
同じ統計調査に "二人以上の世帯" のデータも含まれているので、同項目をみてみる。先ほどと同じように、まず 対象の統計表 ID を調べる。世帯別の集計になるため、男女/年齢といった区分はないが、品目別の支出がわかるデータを探す。
indexer2 = tables.index.str.contains(u'品目別1世帯当たり1か月間の支出') tables[indexer2]

table2 = tables[indexer2].index[3] target2 = dlist[dlist[u'統計表題名及び表番号'] == table2] target2

見つけた 統計表 ID から実データを取得する。
df2 = jpd.DataReader(target2, 'estat', appid=key)
df2

カラム名を変更し、"すし" かつ 地域が "全国" のデータのみを抽出する。
df2.columns = [u'value', u'世帯区分', u'品目分類表', u'地域', u'表章項目'] sushi2 = df2[df2[u'品目分類表'].str.contains(u'すし') & (df2[u'地域'] == u'全国')] sushi2[u'value'] = pd.to_numeric(sushi2[u'value']) sushi2

この数値が二人以上世帯での消費金額の平均になる。先のグラフに重ねてプロットする。
ax = sushi.sum(axis=1).plot.bar(ylim=(0, 1000)) ax.axhline(y=sushi2.iloc[1, 0], color='red')

単身者で 二人以上世帯の消費金額とほぼ同じ金額を使っていれば消費が多いと言ってよさそうだ。単純な見方をすると 料理は面倒だし外食は気疲れする...と感じる頻度が高い層が買っているのだろうか。被調査者によってかなり偏りがあると考えられるので、ここまで細項目を取るなら分布が見てみたい。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (12件) を見る
Python pandas + folium で Leaflet をもっと使いたい
先日参加させていただいた Japan.R でこんな話を聞いた。
Python でも folium というパッケージを使うと JavaScript を書かなくても Leaflet.js の一部機能が使えるのだがあまり情報がない。上の資料に書いてあるようなことが folium でもできるのか調べたい。
folium については前にこんなエントリを書いた。
データの準備
import numpy as np np.__version__ # '1.10.2' import pandas as pd pd.__version__ # u'0.17.1'
サンプルデータとして Wikipedia にある アメリカの国立公園 のデータを使う。まずは pd.read_html でデータを読みこむ。
url = "https://en.wikipedia.org/wiki/List_of_national_parks_of_the_United_States" df = pd.read_html(url, header=0)[0] df

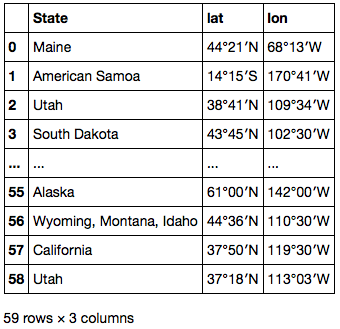
位置情報は "Location" カラムに文字列として保存されている。これを文字列処理して緯度・経度 別々の数値列に変換する。
df['Location'] # 0 Maine 44°21′N 68°13′W<feff> / <feff>44.35°N 68.21°W<feff> / 4... # 1 American Samoa 14°15′S 170°41′W<feff> / <feff>14.25°S 17... # 2 Utah 38°41′N 109°34′W<feff> / <feff>38.68°N 109.57°W<feff> / ... # 3 South Dakota 43°45′N 102°30′W<feff> / <feff>43.75°N 102.... # ... # 55 Alaska 61°00′N 142°00′W<feff> / <feff>61.00°N 142.00°W<feff> ... # 56 Wyoming, Montana, Idaho 44°36′N 110°30′W<feff> / <feff>4... # 57 California 37°50′N 119°30′W<feff> / <feff>37.83°N 119.50... # 58 Utah 37°18′N 113°03′W<feff> / <feff>37.30°N 113.05°W<feff> / ... # Name: Location, dtype: object
まずは .str.extract で Series 中の各要素から正規表現にマッチしたグループを取り出し、州名、緯度、経度 3 列の DataFrame に展開する。
locations = df['Location'].str.extract(u'(\D+) (\d+°\d+′[NS]) (\d+°\d+′[WE]).*') locations.columns = ['State', 'lat', 'lon'] locations
数値としてパースできるよう、.str.replace で記号を置換する。また、南半球 / 西半球の場合は緯度経度を負とするためマイナス符号をつけておく。
locations['lat'] = locations['lat'].str.replace(u'°', '.') locations['lon'] = locations['lon'].str.replace(u'°', '.') locations.loc[locations['lat'].str.endswith('S'), 'lat'] = '-' + locations['lat'] locations.loc[locations['lon'].str.endswith('W'), 'lon'] = '-' + locations['lon'] locations
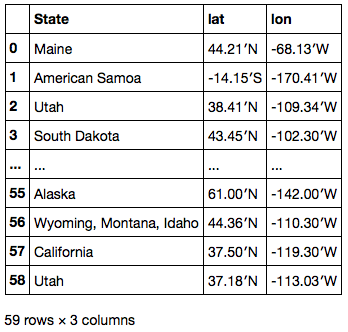
最後の 2 文字は不要のため、.str.slice_replace を使って削除する ( None と置換する )。これで float64 型に変換できるようになる。
locations['lat'] = locations['lat'].str.slice_replace(start=-2) locations['lon'] = locations['lon'].str.slice_replace(start=-2) locations[['lat', 'lon']] = locations[['lat', 'lon']].astype(float) locations

処理した DataFrame を pd.concat で元の DataFrame に追加する。
locations.dtypes # State object # lat float64 # lon float64 # dtype: object df = pd.concat([df, locations], axis=1)
地図の描画
folium で描画した地図は Jupyter Notebook に埋め込んで利用したい。Jupyter 上に描画するための関数を定義する。
import folium folium.__version__ # '0.1.6' from IPython.display import HTML def inline_map(m): m._build_map() iframe = '<iframe srcdoc=\"{srcdoc}\" style=\"width: 80%; height: 400px; border: none\"></iframe>' return HTML(iframe.format(srcdoc=m.HTML.replace('\"', '"')))
まずは シンプルなマーカー。これは前のエントリでやったことと同じ。
m = folium.Map(location=[55, -108], zoom_start=3.0) for i, row in df.iterrows(): m.simple_marker([row['lat'], row['lon']], popup=row['Name']) inline_map(m)

マーカーをクラスタとして表示するには、clustered_marker キーワードに True を渡すだけ。
m = folium.Map(location=[55, -108], zoom_start=3.0) for i, row in df.iterrows(): m.simple_marker([row['lat'], row['lon']], popup=row['Name'], clustered_marker=True) inline_map(m)

地図を拡大・縮小するとマーカーのクラスタ表示が適当に切り替わる。

サークルを表示するには circle_marker を使う。各国立公園の 2014 年の入園者数をサークルの大きさとしてプロットする。
m = folium.Map(location=[40, -95], zoom_start=4.0) for i, row in df.iterrows(): m.circle_marker([row['lat'], row['lon']], radius=np.sqrt(row['Recreation Visitors (2014)[5]']) * 100, popup=row['Name'], line_color='#DF5464', fill_color='#EDA098', fill_opacity=0.5) inline_map(m)

ポリラインは line で引ける。引数には、各点の緯度と経度からなるリストのリストを渡せばよい。グランドキャニオンからいくつかの国立公園を結ぶポリラインを描画してみる。
dests = ['Grand Canyon', 'Zion', 'Bryce Canyon', 'Capitol Reef', 'Arches'] loc_df = df.set_index('Name') locations = loc_df.loc[dests, ['lat', 'lon']].values.tolist() locations # [[36.04, -112.08], # [37.18, -113.03], # [37.34, -112.11], # [38.12, -111.1], # [38.41, -109.34]] m = folium.Map(location=[37.5, -111], zoom_start=7.0) m.line(locations=locations) for dest, loc in zip(dests, locations): m.simple_marker(loc, popup=dest) inline_map(m)

Google Maps Direction API の利用
2 点間の経路をポリラインで描画したい、といった場合は 上のやり方 / 現在のデータでは無理だ。Google Maps Direction API を使って取得した 2 点間の経路をポリラインとして描きたい。
Google Maps Direction API へのアクセスには googlemaps パッケージを利用する。インストールは pip でできる。
ドキュメントはなく、使い方はテストスクリプトを見ろ、とだいぶ硬派な感じだ。それでも自分で API 仕様を調べるよりは早いと思う。
import googlemaps googlemaps.__version__ # '2.4.2'
googlemaps は Google Map に関連したいくつかの API をサポートしている。Directions API を使うには Client.directions。 mode として渡せるのは "driving", "walking", "bicycling", "transit" いずれか。
key = "Your application key" client = googlemaps.Client(key) result = client.directions('Grand Canyon, AZ, USA', 'Arches, UT, USA', mode="driving", departure_time=pd.Timestamp.now()) import pprint pprint.pprint(result, depth=5) # [{u'bounds': {u'northeast': {u'lat': 38.6164979, u'lng': -109.336915}, # u'southwest': {u'lat': 35.8549308, u'lng': -112.1400703}}, # u'copyrights': u'Map data \xa92015 Google', # u'legs': [{u'distance': {u'text': u'333 mi', u'value': 535676}, # u'duration': {u'text': u'5 hours 36 mins', u'value': 20134}, # u'duration_in_traffic': {u'text': u'5 hours 31 mins', # u'value': 19847}, # u'end_address': u'Arches National Park, Utah 84532, USA', # u'end_location': {u'lat': 38.6164979, u'lng': -109.6157153}, # u'start_address': u'Grand Canyon Village, AZ 86023, USA', # u'start_location': {u'lat': 36.0542422, u'lng': -112.1400703}, # u'steps': [{...}, # ...
結果は経路上のポイントごとに step として含まれるようだ。各ステップ の end_location を取得して地図上にプロットすると、ざっくりとした経路がわかる。
steps = result[0]['legs'][0]['steps'] steps[:2] # [{u'distance': {u'text': u'344 ft', u'value': 105}, # u'duration': {u'text': u'1 min', u'value': 10}, # u'end_location': {u'lat': 36.054091, u'lng': -112.1412252}, # u'html_instructions': u'Head <b>west</b>', # u'polyline': {u'points': u'_z`{EljmkT\\fF'}, # u'start_location': {u'lat': 36.0542422, u'lng': -112.1400703}, # u'travel_mode': u'DRIVING'}, # {u'distance': {u'text': u'141 ft', u'value': 43}, # u'duration': {u'text': u'1 min', u'value': 8}, # u'end_location': {u'lat': 36.0544236, u'lng': -112.1414507}, # u'html_instructions': u'Turn <b>right</b> toward <b>Village Loop Drive</b>', # u'maneuver': u'turn-right', # u'polyline': {u'points': u'ay`{EtqmkTI@GBGBIDGFIDKL'}, # u'start_location': {u'lat': 36.054091, u'lng': -112.1412252}, # u'travel_mode': u'DRIVING'}] locs = [step['end_location'] for step in steps] locs = [[loc['lat'], loc['lng']] for loc in locs] locs # [[36.054091, -112.1412252], # [36.0544236, -112.1414507], # [36.05547, -112.1384223], # [36.0395224, -112.1216684], # [36.0520635, -112.1055832], # [35.8550626, -111.4251481], # [36.0755773, -111.3918428], # [36.9304583, -109.5745837], # [37.2655159, -109.6257182], # [37.6254146, -109.4780126], # [37.6254311, -109.4754401], # [38.5724833, -109.5507785], # [38.6109465, -109.6081511], # [38.6164979, -109.6157153]] m = folium.Map(location=[37.5, -111], zoom_start=7.0) m.line(locations=locs) m.simple_marker(locs[0], popup='Grand Canyon, AZ, USA') m.simple_marker(locs[-1], popup='Arches, UT, USA') inline_map(m)

各ステップ中のより詳細な座標は polyline 中に Encoded Polyline Algorithm Format で記録されている。これは googlemaps.convert.decode_polyline 関数でデコードできる。
steps[0]['polyline'] # {u'points': u'_z`{EljmkT\\fF'} googlemaps.convert.decode_polyline(steps[0]['polyline']['points']) # [{'lat': 36.05424, 'lng': -112.14007000000001}, # {'lat': 36.05409, 'lng': -112.14123000000001}]
全ての step から polyline 中の座標を取得すればよさそうだ。適当な関数を書いて地図上にプロットする。
def get_polylines_from_steps(steps): results = [] decode = googlemaps.convert.decode_polyline for step in steps: pl = step['polyline'] locs = decode(pl['points']) locs = [[loc['lat'], loc['lng']] for loc in locs] results.extend(locs) return results locs = get_polylines_from_steps(steps) locs[:10] # [[36.05424, -112.14007000000001], # [36.05409, -112.14123000000001], # [36.05409, -112.14123000000001], # [36.054140000000004, -112.14124000000001], # [36.05418, -112.14126], # [36.05422, -112.14128000000001], # [36.05427, -112.14131], # [36.05431, -112.14135], # [36.05436, -112.14138000000001], # [36.05442, -112.14145]] m = folium.Map(location=[37.5, -111], zoom_start=7.0) m.line(locations=locs) m.simple_marker(locs[0], popup='Grand Canyon, AZ, USA') m.simple_marker(locs[-1], popup='Arches, UT, USA') inline_map(m)

まとめ
folium から Leaflet の以下の機能を利用する方法を記載した。
- シンプルマーカーの表示とクラスタ表示
- サークルの表示
- ポリラインの表示と Google Maps Direction API の利用

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (12件) を見る
Python Jupyter + pandas で DataFrame 表示をカスタマイズする
先日 pandas v0.17.1 がリリースされた。v0.17.0 に対するバグフィックスがメインだが、以下の追加機能もあるため その内容をまとめたい。
HTML 表示のカスタマイズ
Jupyer 上では pandasの DataFrame は自動的に HTML として描画される。この HTML に対して、さまざまな CSS を柔軟に設定できるようになった。
このエントリでは、添付した公式ドキュメントとは少し違う例を記載する。
重要 公式ドキュメントにも記載がされているが v0.17.1 時点で開発中 / Experimental な追加のため、今後 破壊的な変更が発生する可能性がある。ご要望やお気づきの点があれば GitHub issue か @ ください。
以降、Jupyter にて実行する。まず DataFrame を作成して表示する。
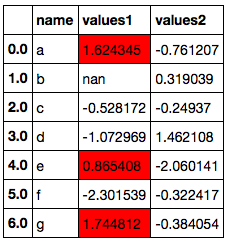
import pandas as pd import numpy as np np.__version__ # '1.10.1' pd.__version__ # u'0.17.1' np.random.seed(1) df = pd.DataFrame({'name': list('abcdefg'), 'values1': np.random.randn(7), 'values2': np.random.randn(7)}) df

v0.17.0 にて DataFrame に DataFrame.style アクセサが追加され、これを経由して種々のカスタマイズができる。例えば、各セルの値に応じてカラーマップを適用するには Styler.background_gradient()。
type(df.style) # pandas.core.style.Styler df.style.background_gradient(cmap='winter')

カラーマップの一部範囲の色のみ利用する場合は low, high キーワードで範囲を指定する。
df.style.background_gradient(cmap='winter', low=2.)

特定の列にのみ Styler を適用する場合は subset キーワード。
df.style.background_gradient(cmap='winter', low=2., subset=['values1'])

さらに特定のセルにのみ適用する場合は 対象セルを指定する pd.IndexSlice インスタンスを subset として指定する。
pd.IndexSlice[2:5, ['values1']] # (slice(2, 5, None), ['values1']) # .loc に渡した場合は対象セルのみをスライス df.loc[pd.IndexSlice[2:5, ['values1']]]

df.style.background_gradient(cmap='winter', low=2., subset=pd.IndexSlice[2:5, ['values1']])

Styler は background_gradient 以外にも 以下のようなメソッドをもつ。
Styler.highlight_max(): 列もしくは行の最大値を指定して色分け。Styler.highlight_min(): 列もしくは行の最小値を指定して色分け。Styler.highlight_null():NaNを指定して色分け。Styler.background_gradient(): 上述。Styler.bar(): 各セルの値に応じて棒グラフのように背景色表示。
df.iloc[1, 1] = np.nan df.style.highlight_null()

さらに、Styler.set_properties を利用すると任意の CSS を全セルに対して適用できる。数値以外のセルを色分けするにはこれを使えばよい。
df.style.set_properties(**{'background-color': '#00ff7f'})

また、Styler はチェインできる。
(df.style.
set_properties(**{'background-color': '#00ff7f'}).
background_gradient(cmap='winter', low=2.).
highlight_null())

より柔軟な条件分けのため、 Styler は .apply 系のメソッドも持つ。使い方は通常の DataFrame.apply や .applymap と同じだが、値そのものではなく CSS を返す関数を適用する。
Styler.applymap: 各セルに対して CSS を返す関数を適用 (関数への入力はスカラー)。Styler.apply: 各列もしくは各行に対して CSS を返す関数を適用 (関数への入力は 各列/各行の値からなるSeries)。
参考 Python pandas データのイテレーションと関数適用、pipe - StatsFragments
セルの値が 0 より小さい場合は 赤 / 太字で表示する場合は Styler.applymap。
def highlight_negative(val): if val < 0: return 'color: {0}; font-weight: bold'.format('red') else: return 'color: {0}'.format('black') df.style.applymap(highlight_negative)

各行について、values1 列の値が values2 列の値より大きいときに values1 列のみ 赤背景で表示したい。行ごとに関数を適用するため、axis=1 を指定する。highlight_values1 関数が返す リストの各要素が、各列に適用される CSS に対応している。
def highlight_values1(s): if s['values1'] > s['values2']: # return ['', 'background-color: red', ''] else: # スタイル変更しない場合は空の文字列のリストを返す return [''] * len(s) df.style.apply(highlight_values1, axis=1)
最後に、Jupyter の widget と組み合わせることで動的なスタイル変更ができる。以下ではスライドバーの位置によって フォントの大きさを変更している。
from IPython.html import widgets @widgets.interact def f(s=(5, 30)): return (df.style.background_gradient('winter', low=2.).set_properties(**{'font-size': '{0}px'.format(s)}))


Jupyter 以外に 同様の出力を HTML として埋め込みたい場合は、Styler.render() を利用する。
style = df.style.background_gradient('winter', low=2.) style.render() # 略 (HTML が文字列として出力される)
ピボットテーブル + グラフ表示
また Jupyter 上での DataFrame の扱いに関連して、データそのものを GUI でインタラクティブに操作 / グラフ表示ができるpivottablejs というパッケージがある。
まとめ
pandas v0.17.1 で追加された DataFrame の HTML 表示のカスタマイズ方法について記載した。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (11件) を見る
Python pandas プロット機能を使いこなす
pandas は可視化のための API を提供しており、折れ線グラフ、棒グラフといった基本的なプロットを簡易な API で利用することができる。一般的な使い方は公式ドキュメントに記載がある。
これらの機能は matplotlib に対する 薄い wrapper によって提供されている。ここでは pandas 側で一処理を加えることによって、ドキュメントに記載されているプロットより少し凝った出力を得る方法を書きたい。
補足 サンプルデータに対する見せ方として不適切なものがあるが、プロットの例ということでご容赦ください。
パッケージのインポート
import matplotlib.pyplot as plt plt.style.use('ggplot') import matplotlib as mpl mpl.__version__ # '1.5.0' import numpy as np np.__version__ # '1.10.1' import pandas as pd pd.__version__ # u'0.17.0'
折れ線グラフ、棒グラフ系
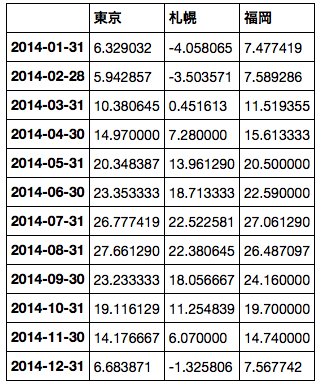
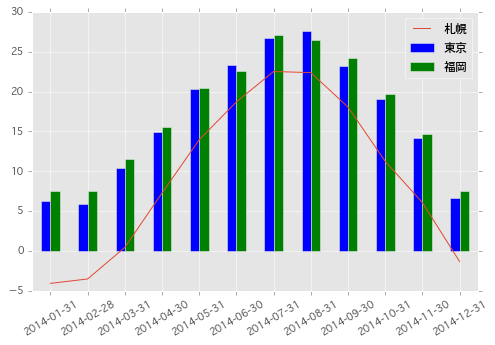
サンプルデータとして 気象庁から 東京、札幌、福岡の 2014 年の日別平均気温データをダウンロードし、pd.read_csv で読み込んだ。
df = pd.read_csv('data.csv', index_col=0, header=[0, 1, 2], skiprows=[0], encoding='shift-jis') df = df.iloc[:, [0, 3, 6]] df.columns = [u'東京', u'札幌', u'福岡'] df.index = pd.to_datetime(df.index) df.head()

まずは単純な折れ線グラフを描く。
df.plot()

次に、適当な単位 (ここでは月次) でグルーピングして棒グラフを書きたい。集計には pd.Grouper を利用する。
参考 Python pandas アクセサ / Grouperで少し高度なグルーピング/集計 - StatsFragments
monthly = df.groupby(pd.Grouper(level=0, freq='M')).mean() monthly
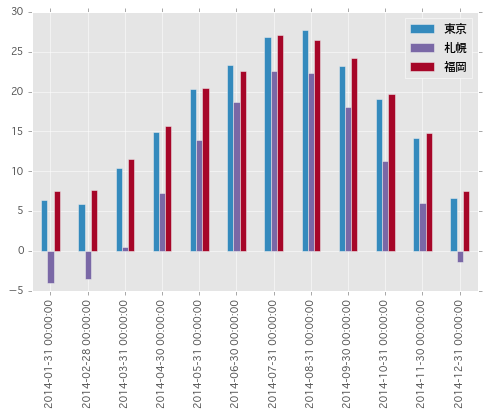
このとき、index が 時系列 ( DatetimeIndex ) の場合、そのままでは x 軸がタイムスタンプとして表示されてしまう。
monthly.plot.bar(color=['#348ABD', '#7A68A6', '#A60628'])
x軸 を簡単にフォーマットしたい場合、Index.format() メソッドを利用して index を文字列表現に変換するとよい。
monthly.index.format() # ['2014-01-31', '2014-02-28', '2014-03-31', '2014-04-30', '2014-05-31', '2014-06-30', # '2014-07-31', '2014-08-31', '2014-09-30', '2014-10-31', '2014-11-30', '2014-12-31'] monthly.index = monthly.index.format() monthly.plot.bar(color=['#348ABD', '#7A68A6', '#A60628'])

また、各棒の塗り分けには colormap を指定することもできる。
monthly.plot.bar(cmap='rainbow')
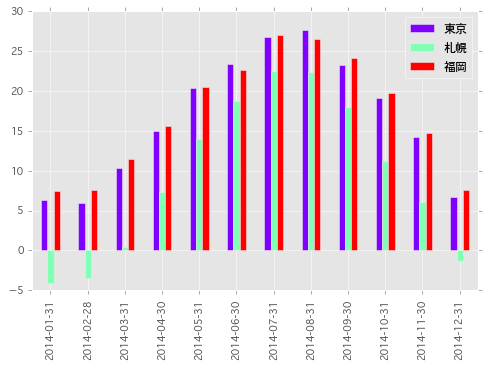
折れ線グラフ / 棒グラフを一つのプロットとして描画する場合は以下のようにする。.plot メソッドは matplotlib.axes.Axes インスタンスを返すため、続くプロットの描画先として その Axes を指定すればよい。
ax = monthly[u'札幌'].plot(legend=True) monthly[[u'東京', u'福岡']].plot.bar(ax=ax, rot=30)
また、matplotlib 側で 極座標の Axes を作成しておけば、レーダーチャートのような描画もできる。
補足 一部 線が切れているのは負値のため。
fig = plt.figure() ax = fig.add_axes([0.1, 0.1, 0.9, 0.9], polar=True) indexer = np.arange(1, 13) * 2 * np.pi / 12 monthly.index = indexer monthly.append(monthly.iloc[0]).plot(ax=ax) ax.set_xticks(indexer) ax.set_xticklabels(np.arange(1, 13));

分布描画系
各都市の気温の分布が見たい、といった場合にはヒストグラムを描画する。
df.plot.hist(bins=100, alpha=0.5)

より細かい単位でグループ分けしてグラフを描きたい場合は、まず plt.subplots で必要な Axes を生成する。続けて、pandas でグループ化したデータを各 Axes に対して描画すればよい。
ここでは月ごとに サブプロットにして カーネル密度推定したグラフを描画してみる。
fig, axes = plt.subplots(4, 3, figsize=(8, 6)) plt.subplots_adjust(wspace=0.5, hspace=0.5) for (ax, (key, group)) in zip(axes.flatten(), df.groupby(pd.Grouper(level=0, freq='M'))): ax = group.plot.kde(ax=ax, legend=False, fontsize=8) ax.set_ylabel('') ax.set_title(key, fontsize=8)

同様に箱ヒゲ図も描ける。
fig, axes = plt.subplots(4, 3, figsize=(8, 6)) plt.subplots_adjust(wspace=0.5, hspace=0.5) for (ax, (key, group)) in zip(axes.flatten(), df.groupby(pd.Grouper(level=0, freq='M'))): ax = group.plot.box(ax=ax) ax.set_ylabel('') ax.set_title(key, fontsize=8)

散布図系
通常の散布図は以下のようにして描ける。
df.plot(kind='scatter', x=u'札幌', y=u'福岡')

各点を適当に色分けしたい場合、c キーワードで各点の色を指定する。また、colormap も合わせて指定できる。ここで指定した値は連続値として扱われるため colorbar が表示されている (非表示にすることもできる)。
df.plot(kind='scatter', x=u'札幌', y=u'福岡', c=df.index.month, cmap='winter')

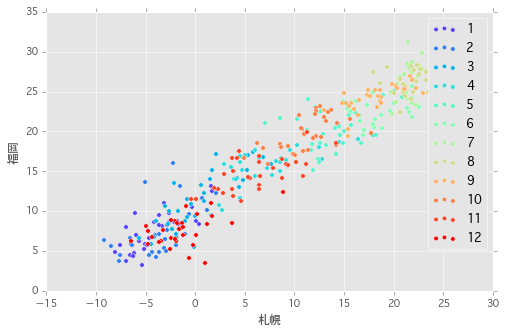
各点をグループ別に塗り分ける ( 離散値として扱う ) 場合は、単一の Axes に対して グループ化したデータを順番に描画していけばよい。
cmap = plt.get_cmap('rainbow') colors = [cmap(c / 12.0) for c in np.arange(1, 13)] colors # [(0.33529411764705885, 0.25584277759443558, 0.99164469551074275, 1.0), # (0.17058823529411765, 0.49465584339977881, 0.96671840426918743, 1.0), # ... # (1.0, 0.25584277759443586, 0.12899921653020341, 1.0), # (1.0, 1.2246467991473532e-16, 6.123233995736766e-17, 1.0)] fig, ax = plt.subplots(1, 1) for i, (key, group) in enumerate(df.groupby(pd.Grouper(level=0, freq='M')), start=1): group.plot(kind='scatter', x=u'札幌', y=u'福岡', color=cmap(i / 12.0), ax=ax, label=i)
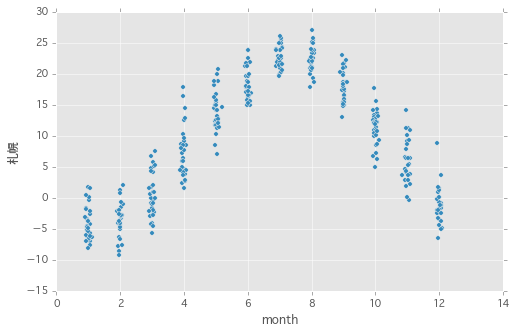
また、ダミーのカラムを作成することで月次の気温の分布を散布図としてみることもできる。
df['month'] = df.index.month df.plot(kind='scatter', x='month', y=u'札幌')

x 軸方向に散らす。
df['month2'] = df.index.month + np.random.normal(loc=0, scale=0.05, size=len(df.index.month)) df.plot(kind='scatter', x='month2', y=u'札幌')
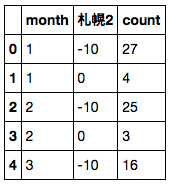
バブルチャートも描ける。前処理として札幌の気温を離散化し、月次のデータ数を集計する。
df2 = df.copy() df2[u'札幌2'] = df2[u'札幌'] // 10 * 10 df2['count'] = 1 df2 = df2.groupby(['month', u'札幌2'])['count'].count().reset_index() df2.head()
バブルの大きさは s キーワードで指定する。
df2.plot.scatter(x='month', y=u'札幌2', s=df2['count'] * 10) df.plot(kind='scatter', x='month2', y=u'札幌')

さらに変形して plt.imshow でヒートマップを描画する。imshow は pandas の API にはないため、DataFrame.pipe でデータを渡す。
piv = pd.pivot_table(df, index=u'札幌2', columns=df.index.month, values=u'札幌', aggfunc='count') piv = piv.fillna(0) piv

piv.pipe(plt.imshow, cmap='winter') ax = plt.gca() ax.invert_yaxis() ax.set_yticks(np.arange(4)) ax.set_yticklabels(piv.index);

まとめ
pandas での前処理 + 可視化機能の組み合わせを利用して、より柔軟にプロットを行う方法を記載した。pandas の裏側は ndarray のため、最後の例のように pandas 側に API がないプロットも簡単に描ける。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (11件) を見る
pandas 0.17.0 の主要な変更点
先日 10/9 に pandas 0.17.0 がリリースされた。直近のバージョンアップの中では かなり機能追加が多いリリースとなった。
重要な変更は リリースノート にハイライトとして列挙しているのだが、これらはある程度 pandas を使いこなしている方向けの記載となっている。
そのため、ここでは よりライトなユーザ向けに重要と思われる変更を書く。特に、ユーザ側のプログラムに影響がある以下の3点について記載する。
- ソート API の統合 (
sort_values/sort_index) - 重複削除 API の改善 (
drop_duplicates/duplicated) .plotアクセサの追加
準備
import numpy as np import pandas as pd np.__version__ # '1.10.1' pd.__version__ # u'0.17.0'
1. ソート API の統合 ( sort_values / sort_index )
これまで一貫性がなかったソート API が以下のように統合された。以前のメソッド / 引数は deprecate されており、将来のバージョン ( 通例では 0.19.0 以降 ) で削除される。
値によるソート ( sort_values として統合):
| 0.16.2以前 | 0.17.0以降 |
|---|---|
Series.order() |
Series.sort_values() |
Series.sort() |
Series.sort_values(inplace=True) |
DataFrame.sort(columns=...) |
DataFrame.sort_values(by=...) |
ラベル ( Index ) によるソート ( sort_index として統合):
| 0.16.2以前 | 0.17.0以降 |
|---|---|
Series.sort_index() |
Series.sort_index() |
Series.sortlevel(level=...) |
Series.sort_index(level=...) |
DataFrame.sort_index() |
DataFrame.sort_index() |
DataFrame.sortlevel(level=...) |
DataFrame.sort_index(level=...) |
DataFrame.sort() |
DataFrame.sort_index() |
それぞれ、引数として以下のオプションが利用できる。
inplace: 破壊的にソートするかどうかを指定する。既定はFalse(非破壊)axis: ソートの方向(軸)を指定するascending: ソート順序 (昇順/降順) を指定する。既定はTrue(昇順)
s = pd.Series(list('afcedb'), index=[1, 3, 2, 4, 0, 5]) s # 1 a # 3 f # 2 c # 4 e # 0 d # 5 b # dtype: object # 値によるソート s.sort_values() # 1 a # 5 b # 2 c # 0 d # 4 e # 3 f # dtype: object # ラベル (index) によるソート s.sort_index() # 0 d # 1 a # 2 c # 3 f # 4 e # 5 b # dtype: object
補足 もともとの仕様は NumPy (というか Python) の .sort メソッドの挙動 ( 破壊的なソート) にあわせたものだった。今回の変更により NumPy とは差異が発生する。
a = np.array([3, 2, 1]) a # array([3, 2, 1]) a.sort() a # array([1, 2, 3])
2. 重複削除 API の改善 ( drop_duplicates / duplicated )
重複した値の削除を行う drop_duplicates で、重複値の全削除ができるようになった。
| 0.16.2以前 | 0.17.0以降 |
|---|---|
.drop_duplicates() |
.drop_duplicates() |
.drop_duplicates(take_last=True) |
.drop_duplicates(keep='last') |
| - | .drop_duplicates(keep=False) |
削除方法の指定は これまでの take_last から keep オプション に変更された。
s = pd.Series(list('abebdcd')) s # 0 a # 1 b # 2 e # 3 b # 4 d # 5 c # 6 d # dtype: object # 重複がある場合、最初に出現した値を残す s.drop_duplicates() # 0 a # 1 b # 2 e # 4 d # 5 c # dtype: object # 重複がある場合、最後に出現した値を残す s.drop_duplicates(keep='last') # 0 a # 2 e # 3 b # 5 c # 6 d # dtype: object # 重複した値を残さない s.drop_duplicates(keep=False) # 0 a # 2 e # 5 c # dtype: object
また、値が重複しているかどうかを調べる .duplicated でも keep オプションが利用できる。
# 重複がある場合、最初に出現した値以外を True s.duplicated() # 0 False # 1 False # 2 False # 3 True # 4 False # 5 False # 6 True # dtype: bool # 重複がある場合、最後に出現した値以外を True s.duplicated(keep='last') # 0 False # 1 True # 2 False # 3 False # 4 True # 5 False # 6 False # dtype: bool # 重複 全てを True s.duplicated(keep=False) # 0 False # 1 True # 2 False # 3 True # 4 True # 5 False # 6 True # dtype: bool
Index の値から重複を全削除する場合は Index.duplicated を以下のように利用すればよい。
s = pd.Series(np.arange(6), index=list('abcbda')) s # a 0 # b 1 # c 2 # b 3 # d 4 # a 5 # dtype: int64 s[~s.index.duplicated(keep=False)] # c 2 # d 4 # dtype: int64
3. .plot アクセサの追加
DataFrame や Series をプロットする際のグラフの種類が、 .plot.bar() や .plot.hist() のように .plot をアクセサとして指定できるようになった。
これまでのメソッド呼び出し ( .plot(kind='bar') ) は引き続き利用できる。
df = pd.DataFrame(np.random.randn(5, 10)) # df.plot(kind='box') と同じ df.plot.box()

その他
また、上記ほどではないが重要な内容を列挙する。気になるものがあれば詳細はリンク先で。
- GIL 解放 (
Dask利用時のパフォーマンス向上) (詳細) pandas.io.dataを deprecate し、別パッケージpandas-datareaderとして分離 (詳細)pd.to_datetimeのエラー処理の変更、日時パース時の挙動統一 (詳細)Index同士の比較演算時の一部挙動の変更 (詳細)- ターミナル上での日本語データ表示時の位置補正オプションの追加 (詳細)
まとめ
pandas 0.17.0 での特に重要な変更点 3 点を記載した。
- ソート API の統合 (
sort_values/sort_index) - 重複削除 API の改善 (
drop_duplicates/duplicated) .plotアクセサの追加
これら以外の変更点については リリースノート を一読お願いします。
10/18 編集 コメントご指摘により誤記を修正

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (11件) を見る
PyConJP 2015: pandas/Daskについてお話させていただきました
10日、11日と PyCon JP に参加させていただきました。ご参加いただいた皆様、スタッフの皆様ありがとうございました。資料はこちらになります。
pandas internals
パフォーマンス向上のための pandas 内部実装の説明といくつかの TIPS について。そのうち翻訳するかもしれません。
Dask: 軽量並列分散フレームワーク (LT)
元ネタ
以下のエントリをベースに、それぞれ新しい内容を追加しています。
Python XGBoost + pandas 連携の改善
一部 こちらの続き。その後 いくつかプルリクを送り、XGBoost と pandas を連携させて使えるようになってきたため、その内容を書きたい。
できるようになったことは 以下 3 点。
DMatrixでのラベルと型の指定pd.DataFrameからのDMatrixの作成xgb.cvの結果をpd.DataFrameとして取得
補足 XGBoost では PyPI の更新をスクリプトで不定期にやっているようで、同一バージョンに見えても枝番が振られていたりして見分けにくい。記載は本日時点のこのコミットの情報。
%matplotlib inline import numpy as np import xgboost as xgb from sklearn import datasets import matplotlib.pyplot as plt plt.style.use('ggplot') xgb.__version__ # '0.4'
1. DMatrix でのラベルと型の指定
これは pandas 関係ない。DMatrix に以下 2 つのプロパティが追加され、任意のラベル/型が指定できるようにした (指定しない場合はこれまでと同じ挙動)。
feature_names: 変数のラベルを指定。ラベルは英数字のみ。feature_types: 変数の型を指定。指定できるのは、q: 量的変数 (既定)i: ダミー変数int: 整数型float: 浮動小数点型。
まずは feature_names のみを指定。
iris = datasets.load_iris() features = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth'] dm = xgb.DMatrix(iris.data, label=iris.target, feature_names=features) dm.feature_names # ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth']
DMatrix で指定した feature_names は xgb.Booster へも引き継がれ、.get_dump 時 やプロットに利用できる。
params={'objective': 'multi:softprob',
'eval_metric': 'mlogloss',
'eta': 0.3,
'num_class': 3}
np.random.seed(1)
bst = xgb.train(params, dm, num_boost_round=18)
xgb.plot_importance(bst)

次に feature_types。これは内部的な学習には全く関係がなく、.get_dump や xgb.plot_tree をする際の ラベルのフォーマットを決めるだけ。例えば以下のようなデータがあったとする。
data = np.array([[1, 0.1, 0], [2, 0.2, 1], [2, 0.2, 0]]) data # array([[ 1. , 0.1, 0. ], # [ 2. , 0.2, 1. ], # [ 2. , 0.2, 0. ]])
このとき、1 列目を int, 2 列目を float, 3 列目をダミー変数 i として扱いたければ以下のように指定すると、 plot_tree したときの見た目が若干変わる。全変数 既定 (q: 量的変数) でもとくに不自由ないので、強いこだわりのある方以外は指定する必要はない。
dm = xgb.DMatrix(data, feature_names=['A', 'B', 'C'], feature_types=['int', 'float', 'i']) dm.feature_types # ['int', 'float', 'i']
補足 これまで、変数のラベル/型の指定は "featmap.txt" のような設定ファイルから別途指定する必要があった。この指定が Python で直接できるようになった。
2 pd.DataFrame からの DMatrix の作成
pandas.DataFrame から DMatrix が作成できるようにした。このとき、feature_names と feature_types は DataFrame の定義から適当に設定される。
補足 DMatrix の label には pd.Series が渡せる (前からできた)。
import pandas as pd # 表示行数を指定 pd.set_option('display.max_rows', 8) # 一行に表示する文字数を指定 pd.set_option('display.width', 120) # pd.DataFrame を作成 train = pd.DataFrame(iris.data, columns=['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth']) train # SepalLength SepalWidth PetalLength PetalWidth # 0 5.1 3.5 1.4 0.2 # 1 4.9 3.0 1.4 0.2 # 2 4.7 3.2 1.3 0.2 # 3 4.6 3.1 1.5 0.2 # .. ... ... ... ... # 146 6.3 2.5 5.0 1.9 # 147 6.5 3.0 5.2 2.0 # 148 6.2 3.4 5.4 2.3 # 149 5.9 3.0 5.1 1.8 # # [150 rows x 4 columns] # DMatrix を作成 dm = xgb.DMatrix(train, label=pd.Series(iris.target)) dm.feature_names # ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth'] dm.feature_types # ['q', 'q', 'q', 'q']
DMatrix に変換できるのは、列が int64, float64, bool の DataFrame のみ。他の型を自動でダミー変数に変換したりはしない。
df = pd.DataFrame([[1, 0.1, False], [2, 0.2, True], [2, 0.2, False]], columns=['A', 'B', 'C']) df # A B C # 0 1 0.1 False # 1 2 0.2 True # 2 2 0.2 False df.dtypes # A int64 # B float64 # C bool # dtype: object dm = xgb.DMatrix(df) dm.feature_names # ['A', 'B', 'C'] dm.feature_types # ['int', 'q', 'i'] # object 型を含むためエラー df = pd.DataFrame([[1, 0.1, 'x'], [2, 0.2, 'y'], [2, 0.2, 'z']], columns=['A', 'B', 'C']) df # A B C # 0 1 0.1 x # 1 2 0.2 y # 2 2 0.2 z df.dtypes # A int64 # B float64 # C object # dtype: object xgb.DMatrix(df) # ValueError: DataFrame.dtypes must be int, float or bool
3. xgb.cv の結果を pd.DataFrame として取得
クロスバリデーションを行う xgb.cv はこれまで実行結果を文字列で返していたため、その結果をパースもしくは目視で確認する必要があった。この返り値を pd.DataFrame もしくは np.ndarray とし、プログラムで処理がしやすいようにした。
train = pd.DataFrame(iris.data, columns=['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth']) dm = xgb.DMatrix(train, label=pd.Series(iris.target)) cv = xgb.cv(params, dm, num_boost_round=50, nfold=10) cv # test-mlogloss-mean test-mlogloss-std train-mlogloss-mean train-mlogloss-std # 0 0.753459 0.027033 0.737631 0.003818 # 1 0.552303 0.048738 0.526929 0.005102 # 2 0.423481 0.066469 0.390115 0.005873 # 3 0.339942 0.082163 0.295637 0.006148 # .. ... ... ... ... # 46 0.208001 0.259161 0.018070 0.001759 # 47 0.208355 0.261166 0.017898 0.001724 # 48 0.208468 0.261520 0.017755 0.001703 # 49 0.208566 0.260967 0.017617 0.001686 # # [50 rows x 4 columns] cv.sort(columns='test-mlogloss-mean') # test-mlogloss-mean test-mlogloss-std train-mlogloss-mean train-mlogloss-std # 11 0.175506 0.177823 0.058496 0.004715 # 13 0.175514 0.194641 0.045222 0.004117 # 14 0.176150 0.203265 0.040493 0.004050 # 12 0.176277 0.186902 0.051074 0.004423 # .. ... ... ... ... # 3 0.339942 0.082163 0.295637 0.006148 # 2 0.423481 0.066469 0.390115 0.005873 # 1 0.552303 0.048738 0.526929 0.005102 # 0 0.753459 0.027033 0.737631 0.003818 # # [50 rows x 4 columns]
pandas がインストールされていない、もしくは as_pandas=False を指定した場合、返り値は np.ndarray となる。
cv = xgb.cv(params, dm, num_boost_round=50, nfold=10, as_pandas=False) cv # array([[ 0.7534586 , 0.02703308, 0.7376308 , 0.00381775], # ... # [ 0.2085664 , 0.26096721, 0.0176169 , 0.00168606]]) np.argmin(cv, axis=0) # array([11, 0, 49, 49])
まとめ
XGBoost と pandas をより便利に使うため、以下 3 点の修正を行った。
DMatrixでのラベルと型の指定pd.DataFrameからのDMatrixの作成xgb.cvの結果をpd.DataFrameとして取得
これ使って Kaggle で大勝利したい...と思ったのですが、他の日本勢に使っていただいたほうが勝率高いはずなのでシェアします。