PyConJP 2016: pandasでの時系列処理についてお話させていただきました
21日、22日と PyCon JP に参加させていただきました。ご参加いただいた皆様、スタッフの皆様ありがとうございました。資料はこちらになります。
pandas による時系列データ処理
pandas を使った時系列データの前処理と、statsmodels での時系列モデリングの触りをご紹介しました。
時系列モデルの考え方については全く説明していないので、以下書籍などをご参照ください。

経済・ファイナンスデータの計量時系列分析 (統計ライブラリー)
- 作者: 沖本竜義
- 出版社/メーカー: 朝倉書店
- 発売日: 2010/02/01
- メディア: 単行本
- 購入: 4人 クリック: 101回
- この商品を含むブログ (6件) を見る
元ネタ
以下のエントリをベースに新しい内容を追加しています。
時系列モデルを含む Python パッケージ
トーク中では ARIMA などの時系列モデルを含むパッケージとして statsmodels についてご説明、PyFlux をご紹介しました。一方 変化点検知や異常検知では、広く使われている Python パッケージはありません。
というわけで、作りました。現状、以下の2手法が実装されています。適当に手法追加しつつ、そのうちblogも書きます。
- 累積和法による変化点検知: R の
{changepoint}の実装と同一 - 成分分解 + Generalized ESD test による異常検知: R の
{AnomalyDetection}の実装に近いもの (同じではない)
補足 statsmodels v0.9ではマルコフ転換モデルが実装予定。また、skyline という異常検知アプリはあります。
Python pandas 欠損値/外れ値/離散化の処理
データの前処理にはいくつかの工程がある。書籍「データ分析プロセス」には 欠損など 前処理に必要なデータ特性の考慮とその対処方法が詳しく記載されている。
が、書籍のサンプルは R なので、Python でどうやればよいかよく分からない。同じことを pandas でやりたい。

- 作者: 福島真太朗,金明哲
- 出版社/メーカー: 共立出版
- 発売日: 2015/06/25
- メディア: 単行本
- この商品を含むブログ (2件) を見る
とはいえ、pandas 自身は統計的 / 機械学習的な前処理手法は持っていない。また Python には R と比べると統計的な前処理手法のパッケージは少なく、自分で実装しないと使えない方法も多い。ここではそういった方法は省略し、pandas でできる前処理 / 可視化を中心に書く。
また、方法自体の説明は記載しないので、詳細を知りたい方は 「データ分析プロセス」を読んでください。
データの要約
import numpy as np import pandas as pd pd.__version__ # u'0.17.1' import matplotlib.pyplot as plt import seaborn as sns
データの特徴をつかむため、要約統計量や相関係数が見たい。ここでは 「データ分析プロセス」と同じく Iris データ (scikit-learn に含まれているもの / 書籍とは少し値が違う) を例として使う。
import sklearn.datasets as datasets iris_data = datasets.load_iris() iris = pd.DataFrame(iris_data.data, columns=iris_data.feature_names) iris['species'] = iris_data.target iris.head()

変数の要約 (要約統計量)
pandas での要約統計量の表示は DataFrame.describe。5 列目
"species" も数値型だが、カテゴリ変数のため除外する。
iris.iloc[:, :4].describe()

"species" についてはラベルごとの頻度が見たいので Series.value_counts で集計する。
iris['species'].value_counts() # 2 50 # 1 50 # 0 50 # Name: species, dtype: int64
2 変数の関係 (相関係数と散布図行列)
また、相関係数の表示は DataFrame.corr。
iris.iloc[:, :4].corr()

散布図行列を描くには seaborn.pairplot。"species" に応じて色分けして描画する。
sns.pairplot(iris, hue='species');

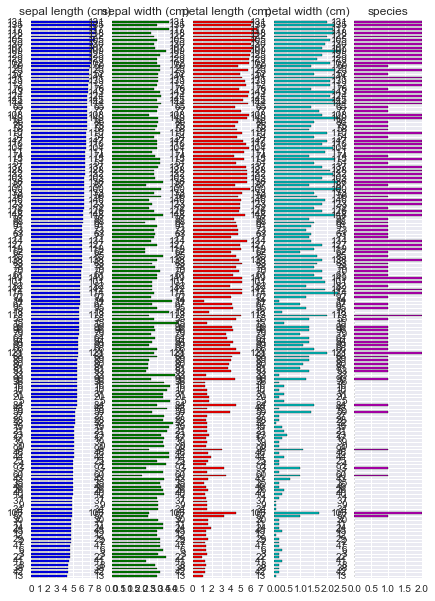
また、R には {tabplot} という data.frame 可視化のためのパッケージがある。これに近い出力は pandas でもかんたんに得られる。
(iris.sort_values('sepal length (cm)'). plot.barh(subplots=True, layout=(1, 5), sharex=False, legend=False));
欠損値
「データ分析プロセス」で使われているサンプルデータ employee_IQ_JP.csv を使う。ファイルは出版社のサポートページ からダウンロードできる。
データは知能指数 "IQ" と業務成果 "JobPerformance" 2 つの変数の関係を示している。3 列目以降は "JobPerformance" が以下いずれかのパターンで欠損した場合の例を示している。
| 欠損発生のパターン | 概要 |
|---|---|
| MCAR | ランダムに欠損している ( 欠損は "IQ" や "JobPerformance" の値に関係しない ) |
| MAR | 他の変数の値と関係して欠損している ( "IQ" が低いと "JobPerformance" の欠損が多い ) |
| MNAR | 欠損が発生しているデータ自身と関係して欠損している ( "JobPerformance" の真の値が低いと "JobPerformance" の欠損が多い ) |
df = pd.read_csv('employee_IQ_JP.csv')
df.head()

欠損パターンの可視化
欠損がそれぞれのパターンで発生した場合に、真の値 "JobPerformance" のうち欠損値となった箇所を 赤三角 "▲" で描く。
fig, axes = plt.subplots(1, 3) fig.tight_layout(w_pad=2.0) for col, ax in zip(['MCAR', 'MAR', 'MNAR'], axes): indexer = df[col].isnull() df[indexer].plot.scatter(x='IQ', y='JobPerformance', marker='^', color='red', label='missing', ax=ax) df[~indexer].plot.scatter(x='IQ', y='JobPerformance', ax=ax) ax.set_title(col)

次に、上よりもカラム数が多いサンプルデータを使って欠損のパターンを可視化する例を示す。R の {mice} パッケージから、nhanes データセットを CSV に出力し、pandas で読み込む。
nhances = pd.read_csv('nhanes.csv', index_col=0) nhances.head()

上の通り複数の変数で欠損が発生している。欠損がどのように発生しているかを調べるには以下のように集計すればよい。
missing = nhances.copy() # 欠損している場合に True とする missing = missing.apply(pd.isnull, axis=0) missing['count'] = 1 missing.groupby(['age', 'bmi', 'hyp', 'chl']).sum()

この結果から以下のことがわかる。
- 欠損がない (全て
False) レコードは 13件 ) - "chl" のみ欠損している ( "chl"のみ
True) レコードは 3 件 - 以下略
また、変数別に欠損しているレコード数を調べるには sum を取ればよい。
missing[['age', 'bmi', 'hyp', 'chl']].sum() # age 0 # bmi 9 # hyp 8 # chl 10 # dtype: int64
また、ある 2 つの変数 "bmi", "hyp" を選んで、欠損がどのように発生しているかを調べたい。DataFrame.pivot_table で集計する。
missing.pivot_table(index='hyp', columns='bmi', values='count', aggfunc='sum')

この結果から、
- 2 変数とも欠損なし: 16 件
- "bmi" のみ欠損: 1 件
- 以下略
上で調べた欠損値の発生状況をプロットすると以下のようになる。
- 左側: 各変数が欠損しているレコード数
- 右側: 欠損している変数の組み合わせごとのレコード数
fig, axes = plt.subplots(1, 3) missing[['age', 'bmi', 'hyp', 'chl']].sum().plot.bar(ax=axes[0]) missing.groupby(['age', 'bmi', 'hyp', 'chl']).sum().plot.barh(ax=axes[2]) axes[1].set_visible(False);

また、変数 "age" について、自身以外の変数 "bmi", "hyp", "chl" がそれぞれが欠損した / しなかった場合の分布を箱ヒゲ図で描く。 "age" の値が 他の変数の欠損とどのような関係にあるかがわかる。
missing['age'] = nhances['age'] fig, axes = plt.subplots(1, 4) fig.tight_layout(w_pad=3.0) sns.boxplot(data=missing, y='age', ax=axes[0]) sns.boxplot(data=missing, y='age', x='bmi', ax=axes[1]) sns.boxplot(data=missing, y='age', x='hyp', ax=axes[2]) sns.boxplot(data=missing, y='age', x='chl', ax=axes[3]);

欠損に対する処理
欠損値に対する対応にはいくつかの方法がある。うち、pandas, scikit-learn でできる方法を記載する。
| 方法 | 概要 |
|---|---|
| リストワイズ法 | 欠損レコードを除去 |
| ペアワイズ法 | 相関係数など 2 変数を用いて計算を行う際に、対象の変数が 欠損している場合に計算対象から除外 |
| 平均代入法 | 欠損を持つ変数の平均値を補完 |
| 回帰代入法 | 欠損を持つ変数の値を 回帰式をもとに補完 |
完全情報最尤推定、多重代入は Python にはなさそうなので、使うなら R のパッケージを呼び出すしかないと思う。
リストワイズ法
リストワイズ法では欠損を除去すれば良いため DataFrame.dropna でできる。
nhances.shape # (25, 4) nhances.dropna(subset=['bmi']).shape # (16, 4) nhances.dropna(subset=['bmi', 'chl'], how='any').shape # (13, 4)
ペアワイズ法
少し手間がかかるが、対象となる 2 変数について欠損しているレコードを除去 -> 計算を繰り返せばできる。
平均代入法
平均代入のように代表値で埋める場合は DataFrame.fillna。
nhances['bmi'] # 1 NaN # 2 22.7 # 3 NaN # 4 NaN # ... # 24 24.9 # 25 27.4 # Name: bmi, dtype: float64 nhances['bmi'].fillna(nhances['bmi'].mean()) # 1 26.5625 # 2 22.7000 # 3 26.5625 # 4 26.5625 # ... # 24 24.9000 # 25 27.4000 # Name: bmi, dtype: float64
回帰代入法
回帰代入では欠損が発生している変数と 欠損の発生に影響している変数とで回帰式を作り、作られた回帰式を使って欠損を補完する。欠損は MAR で発生していないとダメ。サンプルデータとしては 再び employee_IQ_JP.csv を使う。
回帰には scikit-learn を使う。当たり前だが補間された値は回帰直線 (灰色破線) 上に乗る。
import sklearn.linear_model as lm reg = lm.LinearRegression() indexer = df['MAR'].isnull() reg.fit(df.loc[~indexer, ['IQ']], df.loc[~indexer, 'MAR']) predicted = reg.predict(df.loc[indexer, ['IQ']]) df.loc[indexer, 'MAR'] = predicted # プロット ax = df[indexer].plot.scatter(x='IQ', y='MAR', marker='^', color='red', label='missing'); ax = df[~indexer].plot.scatter(x='IQ', y='MAR', ax=ax); x = np.linspace(*ax.get_xlim()) ax.plot(x, reg.coef_[0] * x + reg.intercept_, color='gray', linestyle='dashed')

外れ値
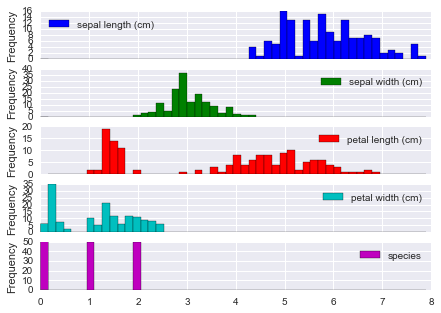
外れ値をみるにはまずデータの分布 / 箱ヒゲ図を描くのがかんたん。
iris.plot(kind='hist', bins=50, subplots=True);
四分位範囲での検出
seaborn.boxplot では 外れ値はダイヤ "♦︎" で描画される。外れ値とみなす閾値は whis オプションを利用して指定できる。既定は 1.5 で、四分位範囲 (IQR) = 第3四分位 - 第1四分位 の 1.5 倍を超えるレコードが外れ値となる。
ここでは "species" のラベルごとに、各変数の箱ヒゲ図を描く。
fig, axes = plt.subplots(3, 1) for i, (n, g) in enumerate(iris.groupby('species')): sns.boxplot(data=g.iloc[:, :4], ax=axes[i]) axes[i].set_ylabel(n)

「データ分析プロセス」に記載されているその他の方法のうち、LOF (Local Outlier Factor) には Python のパッケージがあるが、メンテされているか謎だ。
また、scikit-learn の 1 クラス SVM や ガウス過程 を使う方法もある。これらは 機械学習プロフェッショナルシリーズ「状態変化と異常検知」に記載がある。

- 作者: 井手剛,杉山将
- 出版社/メーカー: 講談社
- 発売日: 2015/08/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (1件) を見る
離散化
以下 2 つの方法については pandas でできる。
| 方法 | 概要 |
|---|---|
| 等間隔区間 (EWD) | 対象のカラムの値を等間隔の区分で分割する |
| 等頻度区間 (EFD) | 分割した区分に同程度の数のレコードが含まれるように分割する |
等間隔区間による離散化
等間隔区間による離散化は pd.cut。どのように分割されたかは categories として表示される。
pd.cut(iris['sepal length (cm)'], 5) # 0 (5.02, 5.74] # 1 (4.296, 5.02] # ... # 148 (5.74, 6.46] # 149 (5.74, 6.46] # Name: sepal length (cm), dtype: category # Categories (5, object): [(4.296, 5.02] < (5.02, 5.74] < (5.74, 6.46] < (6.46, 7.18] < (7.18, 7.9]]
区分ごとのレコード数を数えるには Series.value_counts。結果を Series.sort_index して区分の順番に並べている。
pd.cut(iris['sepal length (cm)'], 5).value_counts().sort_index() # (4.296, 5.02] 32 # (5.02, 5.74] 41 # (5.74, 6.46] 42 # (6.46, 7.18] 24 # (7.18, 7.9] 11 # dtype: int64
等頻度区間による離散化
等頻度区間による離散化は pd.qcut。
pd.qcut(iris['sepal length (cm)'], 5) # 0 (5, 5.6] # 1 [4.3, 5] # ... # 148 (6.1, 6.52] # 149 (5.6, 6.1] # Name: sepal length (cm), dtype: category # Categories (5, object): [[4.3, 5] < (5, 5.6] < (5.6, 6.1] < (6.1, 6.52] < (6.52, 7.9]] pd.qcut(iris['sepal length (cm)'], 5).value_counts().sort_index() # [4.3, 5] 32 # (5, 5.6] 33 # (5.6, 6.1] 30 # (6.1, 6.52] 25 # (6.52, 7.9] 30 # dtype: int64
「データ分析プロセス」に記載されているその他の方法のうち、最小記述長原理 (MDLP) での離散化は scikit-learn に PR が上がっている。
| 方法 | Python パッケージ / リンク |
|---|---|
| 最小記述長原理 (MDLP) | Discretization using Fayyad's MDLP stop criterion by hlin117 · Pull Request #4801 · scikit-learn/scikit-learn · GitHub |
まとめ
書籍「データ分析プロセス」の流れに沿って、欠損値/外れ値/離散化の処理を、pandas で行う方法を記載した。
上で記載した方法 = Python でできる方法は書籍の内容のうち比較的 簡単な方法だけだ。より網羅的に知りたい方は書籍を読んでいただくのがいい。R ユーザに限らずおすすめ。

- 作者: 福島真太朗,金明哲
- 出版社/メーカー: 共立出版
- 発売日: 2015/06/25
- メディア: 単行本
- この商品を含むブログ (2件) を見る
Python pandas で e-Stat のデータを取得したい
e-Stat とは
"「政府統計の総合窓口(e-Stat)」は、各府省が公表する統計データを一つにまとめ、統計データの検索をはじめとした、さまざまな機能を備えた政府統計のポータルサイト" だそうだ。このデータを pandas で読めるとうれしい...ということで対応した。
インストール
$ pip install japandas
パッケージのインポート
import numpy as np np.__version__ # '1.10.2' import pandas as pd pd.__version__ # u'0.17.1' import japandas as jpd jpd.__version__ # '0.2.0'
アプリケーション ID の取得
e-Stat を利用するには 利用登録とアプリケーション ID の取得が必要。利用ガイドに沿って登録する。
データの取得
japandas を利用してデータを取得する。データの取得は以下 2 ステップで行う
- "政府統計コード" を利用して、統計調査に含まれる統計表 ( 実データ ) の一覧とその ID ( 統計表 ID ) を取得する。
- 取得した統計表 ID を利用して、実データを取得する
1. 統計表一覧の取得
e-Stat 提供データ一覧 に含まれる統計調査のうち、今回は 00200564 全国消費実態調査 を利用する。
jpd.DataReader で ID "00200564" のデータを取得すると、以下のような DataFrame が返ってくる。各カラムの詳細は e-Stat API 仕様 に記載されている。
key = "Your application ID" dlist = jpd.DataReader("00200564", 'estat', appid=key) dlist

一つの "統計表題名及び表番号" は "調査年月" が異なる複数のデータを持つことがある。値をユニークにした方が中身を確認しやすい。
tables = dlist[u'統計表題名及び表番号'].value_counts().to_frame()
tables

ここでは "平成26年全国消費実態調査 > 全国 > 品目及び購入先・購入地域に関する結果 > 単身世帯" の "男女,年齢階級,購入形態,品目別1世帯当たり1か月間の支出" のデータを取得したい。
この時点では 正確な "統計表題名及び表番号" がわからないため、まずはそれらしい文字列でレコードを抽出する。
indexer = tables.index.str.contains(u'男女,年齢階級,購入形態,品目別1世帯当たり1か月間の支出') indexer # array([False, False, False, ..., False, False, False], dtype=bool) tables[indexer]

上の結果から 正確な "統計表題名及び表番号" が得られるため、元データから対象のレコードが抽出できる。
table = tables[indexer].index[0] table # [単身世帯]フロー編第149表 男女,年齢階級,購入形態,品目別1世帯当たり1か月間の支出 target = dlist[dlist[u'統計表題名及び表番号'] == table] target

2. 実データの取得
- で調べた "統計表 ID" (
"0003109612") をjpd.DataReaderに渡せばよい。
df = jpd.DataReader("0003109612", 'estat', appid=key) # 略
が、いちいち文字列を抽出したり再入力するのは面倒だ。そんな時は、上の結果 ( 取得対象の "統計表 ID" を含む DataFrame) をそのまま渡してもよい。複数のレコードがある場合は全データを連結して返す。
df = jpd.DataReader(target, 'estat', appid=key)
df

出典:「平成26年全国消費実態調査調査結果」(総務省統計局)
取得したデータを集計してみる。各属性に対応する値は "value" カラムに含まれている。まず、カラム名を簡潔なものに変更する。
df.columns =[u'value', u'世帯区分', u'品目分類表', u'地域', u'表章項目', u'男女', u'年齢階級', u'購入形態']
また、dtypes を見ると全ての列が object 型になっている。通常、"value" には数値が入るため、jpd.DataReader は "value" が数値に変換できる場合は自動で変換するのだが、このデータでは何らかの理由で失敗しているようだ。
df.dtypes # value object # 世帯区分 object # 品目分類表 object # 地域 object # 表章項目 object # 男女 object # 年齢階級 object # 購入形態 object # dtype: object
pd.to_numeric で数値に変換しようとすると ValueError が発生する。データを見ると "value" にハイフン "-" がいくつか使われているようだ (やめてくれ...)。
文字列処理してもよいが、今回は特に何もしなくても以降の処理で解決されるため、とりあえずそのままにしておく。
1/9 補足 v0.2.1 では "-" を欠損として扱うように修正済み。
pd.to_numeric(df['value']) # ValueError: Unable to parse string df['value'].str.isnumeric() # 時間軸(年次) # 2014-01-01 True # 2014-01-01 True # 2014-01-01 True # 2014-01-01 False # ... # 2014-01-01 False # 2014-01-01 False # 2014-01-01 False # 2014-01-01 False # Name: value, dtype: bool
"品目分類表" の値をみるとかなり細かい項目に分けられていることがわかる。
統計調査に利用された調査票 を参照すると、調査形式は家計簿のようなフォーマットに任意の品名を書き込むもののようだ。品目分類を被調査者が記入する欄はなく、集計時に家計簿を品目分類表に従って分類しているのだろう。
df[u'品目分類表'].unique() # array([u'集計世帯数', # u'世帯数分布(抽出率調整)', # u'世帯数分布(抽出率調整)(1万分比)', # ..., u'仕送り金', # u'国内遊学仕送り金', # u'他の仕送り金'], dtype=object)
データから "すし" を含むレコードを抽出してみる。...弁当とは? この調査票には詳細が記載されていないようだが、他の調査を見ると スーパーで買うパックの寿司を指しているものと思う (おにぎりは別項目)。
filtered = df[df[u'品目分類表'].str.contains(u'すし')] filtered

抽出されたレコードの
"value" には 数値として不正な文字列は含まれないため、pd.to_numeric で数値に変換できる。
filtered['value'] = pd.to_numeric(filtered['value']) filtered.dtypes # value int64 # 世帯区分 object # 品目分類表 object # 地域 object # 表章項目 object # 男女 object # 年齢階級 object # 購入形態 object # dtype: object
Series.nunique で各列に含まれるユニークな値の数を調べる。"男女", "年齢階級", "購入形態" でレコードが分かれているため、それら 3 つをキーにして集計してやればよい。
filtered.apply(lambda x: x.nunique()) # value 76 # 世帯区分 1 # 品目分類表 1 # 地域 1 # 表章項目 1 # 男女 3 # 年齢階級 8 # 購入形態 4 # dtype: int64 sushi = pd.pivot_table(filtered, index=[u'男女', u'年齢階級'], columns=u'購入形態', values='value', aggfunc='sum') sushi

"購入形態" (支払い方法) には興味がないので、"合計" の値だけを抽出してプロットする。
60 代 男性 単身者 は "すし(弁当)" への消費金額が比較的多いようだ。金額的に月 1 〜 2 回買っている感じだろうか。また女性も一部男性と比べ高い。
sushi = sushi[[u'合計']] sushi.plot.bar(ylim=(0, 1000))

追加データでの確認
同じ統計調査に "二人以上の世帯" のデータも含まれているので、同項目をみてみる。先ほどと同じように、まず 対象の統計表 ID を調べる。世帯別の集計になるため、男女/年齢といった区分はないが、品目別の支出がわかるデータを探す。
indexer2 = tables.index.str.contains(u'品目別1世帯当たり1か月間の支出') tables[indexer2]

table2 = tables[indexer2].index[3] target2 = dlist[dlist[u'統計表題名及び表番号'] == table2] target2

見つけた 統計表 ID から実データを取得する。
df2 = jpd.DataReader(target2, 'estat', appid=key)
df2

カラム名を変更し、"すし" かつ 地域が "全国" のデータのみを抽出する。
df2.columns = [u'value', u'世帯区分', u'品目分類表', u'地域', u'表章項目'] sushi2 = df2[df2[u'品目分類表'].str.contains(u'すし') & (df2[u'地域'] == u'全国')] sushi2[u'value'] = pd.to_numeric(sushi2[u'value']) sushi2

この数値が二人以上世帯での消費金額の平均になる。先のグラフに重ねてプロットする。
ax = sushi.sum(axis=1).plot.bar(ylim=(0, 1000)) ax.axhline(y=sushi2.iloc[1, 0], color='red')

単身者で 二人以上世帯の消費金額とほぼ同じ金額を使っていれば消費が多いと言ってよさそうだ。単純な見方をすると 料理は面倒だし外食は気疲れする...と感じる頻度が高い層が買っているのだろうか。被調査者によってかなり偏りがあると考えられるので、ここまで細項目を取るなら分布が見てみたい。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (12件) を見る
10 Minutes to DataFrames.jl
この記事は Julia Advent Calendar 2015 23 日目の記事です。
Julia で DataFrame を扱うパッケージ DataFrames.jl の使い方をまとめたい。
下の pandas ドキュメントにあるような処理が DataFrames.jl でどう書けるのかを整理する。
versioninfo() # Julia Version 0.4.2 # Commit bb73f34 (2015-12-06 21:47 UTC)
インストール
Pkg.add("DataFrames") Pkg.installed("DataFrames") # v"0.6.10" using DataFrames
データの作成
DataFrames.jl では 1 次元データ構造である DataArray ( DataArrays.jl ) と 2 次元データ構造である DataFrame を扱う。それぞれ、R や Python ( pandas ) では以下のようなクラスに対応する。
Julia (DataFrames.jl) |
R | Python (pandas) |
|---|---|---|
DataArrays.DataArray |
atomic ( vector ) |
Series |
DataFrames.DataFrame |
data.frame |
DataFrame |
DataArrayの作成
DataArray は直接作成せず、@data マクロを利用して作るのがよい。@data マクロを使うとでは欠損値 ( NA ) をよしなに処理して DataArray 化してくれる。
s = @data([1, NA, 3]) # 3-element DataArrays.DataArray{Int64,1}: # 1 # NA # 3 # データの実体 s.data # 3-element Array{Int64,1}: # 1 # 1 # 3 # NA に対応するマスク s.na # 3-element BitArray{1}: # false # true # false # NG! [1, NA, 3] # LoadError: MethodError: `convert` has no method matching convert(::Type{Int64}, ::DataArrays.NAtype)
DataFrame の作成
作成したいデータの "列名 = Array" のペアを渡す。
df = DataFrame(A = [1, 2, 3, 4, 5, 6, 7, 8, 9, 10], B = [10, 9, 8, 7, 6, 5, 4, 3, 2, 1], C = ["A", "B", "C", "A", "B", "C", "A", "B", "C", "A"]) # 10x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|----|-----| # | 1 | 1 | 10 | "A" | # | 2 | 2 | 9 | "B" | # | 3 | 3 | 8 | "C" | # | 4 | 4 | 7 | "A" | # | 5 | 5 | 6 | "B" | # | 6 | 6 | 5 | "C" | # | 7 | 7 | 4 | "A" | # | 8 | 8 | 3 | "B" | # | 9 | 9 | 2 | "C" | # | 10 | 10 | 1 | "A" |
DataFrame の確認
各列の型は以下のようにして確認できる。colwise は関数を列ごとに適用する generic function。
typeof(df) # DataFrames.DataFrame colwise(typeof, df) # 3-element Array{Any,1}: # [DataArrays.DataArray{Int64,1}] # [DataArrays.DataArray{Int64,1}] # [DataArrays.DataArray{ASCIIString,1}] eltype(df) # Any colwise(eltype, df) # 3-element Array{Any,1}: # [Int64] # [Int64] # [ASCIIString]
列名の表示は names。行名や index にあたるものはない (行番号のみ)。
names(df) # 3-element Array{Symbol,1}: # :A # :B # :C
先頭 / 末尾のレコードを確認したい場合は head もしくは tail。既定では 6 レコード表示。
head(df) # 6x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|---|----|-----| # | 1 | 1 | 10 | "A" | # | 2 | 2 | 9 | "B" | # | 3 | 3 | 8 | "C" | # | 4 | 4 | 7 | "A" | # | 5 | 5 | 6 | "B" | # | 6 | 6 | 5 | "C" | tail(df, 3) # 3x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|---|-----| # | 1 | 8 | 3 | "B" | # | 2 | 9 | 2 | "C" | # | 3 | 10 | 1 | "A" |
要約統計量の表示は describe。これは表示のためだけの関数のようで、返り値は Void になる。
describe(df) # A # Min 1.0 # 1st Qu. 3.25 # Median 5.5 # Mean 5.5 # 3rd Qu. 7.75 # Max 10.0 # NAs 0 # NA% 0.0% # # B # Min 1.0 # 1st Qu. 3.25 # ... ... typeof(describe(df)) # Void
入出力
DataFrames.jl では CSV のみ。
writetable("data.csv", df) df = readtable("data.csv")
データの選択
行や列を選択する操作を記載する。
補足 DataFrame の出力の表示が一部 ドット (...) で省略されているが、これは自分が手動でやった。既定では全レコードが表示されるようだ。
位置、ラベルによる選択
R や pandas と同じく、引数をスカラーで渡すと返り値の次元が減って DataArray になる。
列名を指定する場合、引数は文字列ではなく Symbol として渡す必要があるようだ。Int を渡した場合は列番号での選択になる。
df[:A] # 10-element DataArrays.DataArray{Int64,1}: # 1 # 2 # ... # 9 # 10 # NG! df["A"] # LoadError: MethodError: `getindex` has no method matching getindex(::DataFrames.DataFrame, ::ASCIIString) df[Symbol("A")] # 略 df[1] # 略
対象を Array や UnitRange で指定すると 返り値は DataFrame となる。
df[[:A]] # 10x1 DataFrames.DataFrame # | Row | A | # |-----|----| # | 1 | 1 | # | 2 | 2 | # ... .. # | 9 | 9 | # | 10 | 10 | df[[1]] # 略 df[2:3] # 10x2 DataFrames.DataFrame # | Row | B | C | # |-----|----|-----| # | 1 | 10 | "A" | # | 2 | 9 | "B" | # ... .. ... # | 9 | 2 | "C" | # | 10 | 1 | "A" | df[[:B, :C]] # 略
行 / 列それぞれで指定したい場合は順番に引数として渡す。行番号のみで選択したい場合、第二引数に : を渡す。
df[5:7, :] # 3x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|---|---|-----| # | 1 | 5 | 6 | "B" | # | 2 | 6 | 5 | "C" | # | 3 | 7 | 4 | "A" | df[3:4, [:A, :B]] # 2x2 DataFrames.DataFrame # | Row | A | B | # |-----|---|---| # | 1 | 3 | 8 | # | 2 | 4 | 7 | df[2, :A] # 2
条件式による選択
julia の通常の演算子は element-wise ではない。element-wise に操作したい場合はドットで始まる演算子を使う。この仕様は明示的で良いと思う。
df[:A] == 0 # false df[:A] .== 0 # 10-element DataArrays.DataArray{Bool,1}: # false # false # ..... # false # false
上で作成した Array を使えば 対応する行のみが選択できる。
df[df[:A] .> 5, :] # 5x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|---|-----| # | 1 | 6 | 5 | "C" | # | 2 | 7 | 4 | "A" | # | 3 | 8 | 3 | "B" | # | 4 | 9 | 2 | "C" | # | 5 | 10 | 1 | "A" |
対応する element-wise 演算子がない場合は 内包表記を使って Bool の Array を作ればよい。
[in(x, ("A", "B")) for x in df[:C]] # 10-element Array{Bool,1}: # true # true # ..... # false # true df[[in(x, ("A", "B")) for x in df[:C]], :] # 7x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|----|-----| # | 1 | 1 | 10 | "A" | # | 2 | 2 | 9 | "B" | # ... .. .. ...| # | 6 | 8 | 3 | "B" | # | 7 | 10 | 1 | "A" |
代入
データ選択した箇所に値を代入できる。
df[[1, 4], :A] = NA df # 10x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|----|-----| # | 1 | NA | 10 | "A" | # | 2 | 2 | 9 | "B" | # | 3 | 3 | 8 | "C" | # | 4 | NA | 7 | "A" | # | 5 | 5 | 6 | "B" | # | 6 | 6 | 5 | "C" | # | 7 | 7 | 4 | "A" | # | 8 | 8 | 3 | "B" | # | 9 | 9 | 2 | "C" | # | 10 | 10 | 1 | "A" |
ソート
generic function を利用してソートできる。ソート対象の列や 順序などが引数で指定できる。関数名が ! で終わるものは破壊的な操作を行うもの。
sort(df, cols = :B) # 10x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|----|-----| # | 1 | 10 | 1 | "A" | # | 2 | 9 | 2 | "C" | # | 3 | 8 | 3 | "B" | # | 4 | 7 | 4 | "A" | # | 5 | 6 | 5 | "C" | # | 6 | 5 | 6 | "B" | # | 7 | NA | 7 | "A" | # | 8 | 3 | 8 | "C" | # | 9 | 2 | 9 | "B" | # | 10 | NA | 10 | "A" |
欠損値
欠損値 NA は DataArrays.jl にて定義されており、Built-in にある非数 NaN とは異なる。
typeof(NA) # DataArrays.NAtype NA == NA # NA NA + 1 # NA NA | true # true is(NA, NA) # true typeof(NaN) # Float64
Array の各要素が NA かどうか調べるには isna を使う。
isna(df[:A]) # 10-element BitArray{1}: # true # false # false # true # ..... # false # false df[isna(df[:A]), :] # 2x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|----|-----| # | 1 | NA | 10 | "A" | # | 2 | NA | 7 | "A" | df[~isna(df[:A]), :] # 8x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|---|-----| # | 1 | 2 | 9 | "B" | # | 2 | 3 | 8 | "C" | # | 3 | 5 | 6 | "B" | # | 4 | 6 | 5 | "C" | # | 5 | 7 | 4 | "A" | # | 6 | 8 | 3 | "B" | # | 7 | 9 | 2 | "C" | # | 8 | 10 | 1 | "A" |
ある列について NA を除きたければ dropna。NA を含む列を集約する場合に有用。
dropna(df[:A]) # 8-element Array{Int64,1}: # 2 # 3 # 5 # .. # 9 # 10 mean(df[:A]) # NA mean(dropna(df[:A])) # 6.25
欠損値をパディングしたい場合は データ選択して代入。ただし DataFrame に異なる型の列が含まれている場合は (実際には代入が発生しなくても) エラーになるため、列名も明示したほうがよい。
df[isna(df[:A]), :] = 0 # LoadError: MethodError: `convert` has no method matching convert(::Type{ASCIIString}, ::Int64) df[isna(df[:A]), :A] = 0 df # 10x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|----|----|-----| # | 1 | 0 | 10 | "A" | # | 2 | 2 | 9 | "B" | # | 3 | 3 | 8 | "C" | # | 4 | 0 | 7 | "A" | # ... .. .. ... # | 9 | 9 | 2 | "C" | # | 10 | 10 | 1 | "A" |
演算、集計
演算
DataArray 同士の演算は element-wise な演算になる。
df[:A] - df[:B] # 10-element DataArrays.DataArray{Int64,1}: # -10 # -7 # ... # 7 # 9
集計、集約
ある一つの列を集約したい場合は そのまま集約関数に渡せばよい。
mean(df[:B])
# 5.5
複数の列を集約したい場合は aggregate もしくは colwise。aggregate の結果は DataFrame に、colwise は Array{Any,1} になる。
aggregate(df[[:A, :B]], sum) # 1x2 DataFrames.DataFrame # | Row | A_sum | B_sum | # |-----|-------|-------| # | 1 | 50 | 55 | colwise(sum, df[[:A, :B]]) # 2-element Array{Any,1}: # [50] # [55]
aggregate では複数列 / 複数の集約関数で集計を行うこともできる。
aggregate(df, :C, mean) # 3x3 DataFrames.DataFrame # | Row | C | A_mean | B_mean | # |-----|-----|--------|--------| # | 1 | "A" | 4.25 | 5.5 | # | 2 | "B" | 5.0 | 6.0 | # | 3 | "C" | 6.0 | 5.0 | aggregate(df, :C, [mean, sum]) # 3x5 DataFrames.DataFrame # | Row | C | A_mean | A_sum | B_mean | B_sum | # |-----|-----|--------|-------|--------|-------| # | 1 | "A" | 4.25 | 17 | 5.5 | 22 | # | 2 | "B" | 5.0 | 15 | 6.0 | 18 | # | 3 | "C" | 6.0 | 18 | 5.0 | 15 |
グループ別に集約したい場合は by もしくは aggregate。
by(df, :C, x -> mean(x[:A])) # 3x2 DataFrames.DataFrame # | Row | C | x1 | # |-----|-----|------| # | 1 | "A" | 4.25 | # | 2 | "B" | 5.0 | # | 3 | "C" | 6.0 | aggregate(df, :C, mean) # 3x3 DataFrames.DataFrame # | Row | C | A_mean | B_mean | # |-----|-----|--------|--------| # | 1 | "A" | 4.25 | 1.25 | # | 2 | "B" | 5.0 | 6.0 | # | 3 | "C" | 6.0 | 5.0 |
関数適用
また colwise を使うと ラムダ式を各列に適用することもできる ( apply のような操作)。
colwise(x -> mean(x[1:5]) - mean(6:10), df[[:A, :B]]) # 2-element Array{Any,1}: # [-6.0] # [0.0]
連結、結合、変形
連結
以下 2 つの関数で行う。昔は rbind, cbind という関数があったようだが、すでに削除されている。
vcat:DataFrameを縦方向に連結。hcat:DataFrameを横方向に連結。
df1 = DataFrame(A = [1, 2, 3], B = [4, 5, 6]) df2 = DataFrame(A = [11, 12, 13], B = [14, 15, 16]) vcat(df1, df2) # 6x2 DataFrames.DataFrame # | Row | A | B | # |-----|----|----| # | 1 | 1 | 4 | # | 2 | 2 | 5 | # | 3 | 3 | 6 | # | 4 | 11 | 14 | # | 5 | 12 | 15 | # | 6 | 13 | 16 | df3 = DataFrame(A = [1, 2, 3], B = [4, 5, 6]) df4 = DataFrame(C = [11, 12, 13], D = [14, 15, 16]) hcat(df3, df4) # 3x4 DataFrames.DataFrame # | Row | A | B | C | D | # |-----|---|---|----|----| # | 1 | 1 | 4 | 11 | 14 | # | 2 | 2 | 5 | 12 | 15 | # | 3 | 3 | 6 | 13 | 16 |
結合
特定の列の値で結合するには join。結合方法は kind で指定できる。一通りの結合方法は揃っている。
left = DataFrame(A = [1, 2, 3], B = [11, 12, 13]) right= DataFrame(A = [2, 3, 4], C = [12, 13, 14]) join(left, right, on = :A) # 2x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|---|----|----| # | 1 | 2 | 12 | 12 | # | 2 | 3 | 13 | 13 | join(left, right, on = :A, kind = :outer) # 4x3 DataFrames.DataFrame # | Row | A | B | C | # |-----|---|----|----| # | 1 | 2 | 12 | 12 | # | 2 | 3 | 13 | 13 | # | 3 | 1 | 11 | NA | # | 4 | 4 | NA | 14 |
変形
いわゆる tidy なデータへの変換と逆変換。
stack,melt: 列持ち → 行持ちへの変換 (unpivot)。unstack: 行持ち → 列持ちへの変換 (pivot)。
df5 = DataFrame(Name = ["A", "B", "C", "D"], width = [1, 2, 3, 4], height = [10, 20, 30, 40]) stacked = stack(df5, [:width, :height]) # 8x3 DataFrames.DataFrame # | Row | variable | value | Name | # |-----|----------|-------|------| # | 1 | width | 1 | "A" | # | 2 | width | 2 | "B" | # | 3 | width | 3 | "C" | # | 4 | width | 4 | "D" | # | 5 | height | 10 | "A" | # | 6 | height | 20 | "B" | # | 7 | height | 30 | "C" | # | 8 | height | 40 | "D" | melt(df5, :Name) # 略 unstack(stacked, :Name, :variable, :value) # 4x3 DataFrames.DataFrame # | Row | variable | height | width | # |-----|----------|--------|-------| # | 1 | "A" | 10 | 1 | # | 2 | "B" | 20 | 2 | # | 3 | "C" | 30 | 3 | # | 4 | "D" | 40 | 4 |
データ型固有の処理
データ型に固有の操作を記載する。pandas ではデータ型固有の操作をまとめた .str, .dt などのアクセサがあるが、DataFrames.jl にはそれらに相当するものはなさそうだ。
標準の関数は一部 Array にも適用できるが、そうでない場合は 内包表記を使ってデータを操作する。
日時型
標準には Datetime と Period 二つの型がある。Python でいうと datetime と timedelta + 一部 relativedelta に相当する型だ。これらは DataArray や DataFrame に値として含めることができる。
DataFrames.jl としてはリサンプリングやタイムゾーンなどは機能として持っていない。
dates = @data([DateTime(2013, 01, i) for i in 1:4]) # 6-element DataArrays.DataArray{DateTime,1}: # 2013-01-01T00:00:00 # 2013-01-02T00:00:00 # 2013-01-03T00:00:00 # 2013-01-04T00:00:00 # DateTime から日付を取得 Dates.day(dates) # 4-element Array{Union{DataArrays.NAtype,Int64},1}: # 1 # 2 # 3 # 4 # DateTime を Period に変換 periods = [Dates.Year(d) for d in dates] # 4-element Array{Base.Dates.Year,1}: # 2013 years # 2013 years # 2013 years # 2013 years [DateTime(p) for p in periods] # 4-element Array{Any,1}: # 2013-01-01T00:00:00 # 2013-01-01T00:00:00 # 2013-01-01T00:00:00 # 2013-01-01T00:00:00
時系列については別のパッケージがあるため、こちらを使うのがよいのかもしれない。
文字列型
それぞれ 内包表記で処理する。
lowercase(df[:C]) # LoadError: MethodError: `lowercase` has no method matching lowercase(::DataArrays.DataArray{ASCIIString,1}) [lowercase(x) for x in df[:C]] # 10-element Array{Any,1}: # "a" # "b" # ... # "c" # "a"
カテゴリカル型
いわゆるカテゴリカル型はない。Array の利用メモリを削減するための PooledDataArray というクラスがある。
まとめ
DataFrames.jl でのデータ操作を整理した。
API や 使い勝手は pandas よりも R の data.frame に近い感じだ (ただし dplyr は存在しない )。Julia にはパイプ演算子があるため、pandas よりは dplyr のようなパッケージがあると流行りそうだ。
Python Dask.Array で 並列 / Out-Of-Core 処理
この記事は Python Advent Calendar 2015 13 日目の記事です。
Python で手軽に並列 / Out-Of-Core 処理を行うためのパッケージである Dask について書きたい。Dask を使うと以下のようなメリットが得られる。
- 環境構築 / インストールが
pipで簡単にできる - 手軽に並列処理ができる
- Out-Of-Core (メモリに乗らないデータ) 処理ができる
補足 Dask は手持ちの PC の シングルコア / 物理メモリでは処理が少しきついかな、といった場合に利用するパッケージのため、より大規模 / 高速 / 安定した処理を行いたい場合には Hadoop や Spark を使ったほうがよい。
Dask は以下 3 つのサブパッケージを持つ。
| サブモジュール | ベースパッケージ |
|---|---|
dask.array |
NumPy |
dask.bag |
PyToolz (list, set, dict に対する処理) |
dask.dataframe |
pandas |
うち dask.dataframe については以前にエントリを書いた。
今回は NumPy API のサブセットをもつ dask.array について。dask.array でも基本的な考え方 / 挙動は dask.dataFrame と同じなので まず上のエントリを読んでください。
インストール
pip で。
# pip install dask
基本的な操作
import numpy as np np.__version__ # '1.10.1' import dask dask.__version__ # '0.7.5' import dask.array as da
dask.DataFrame はいくつかの行を chunk としてまとめて並列処理を行っていたが、dask.Array は各 axis それぞれを chunk として分割することができる。以下では 4 行 4 列 の ndarray を 2 行 2 列 計 4 つの chunk に分割する。
arr = np.array([[1, 2, 3, 4], [5, 6, 7, 8], [9, 10, 11, 12], [13, 14, 15, 16]]) arr # array([[ 1, 2, 3, 4], # [ 5, 6, 7, 8], # [ 9, 10, 11, 12], # [13, 14, 15, 16]]) darr = da.from_array(arr, chunks=2) darr # dask.array<from-ar..., shape=(4, 4), dtype=int64, chunksize=(2, 2)>
dask.Dataframe と同じく、dask.Array のメソッドを呼び出すことで 内部の Computational Graph を更新していく。評価 (計算の実行) を行うには .compute()。また、.visualize() で Computational Graph を描画することができる。
各行の合計を取る処理をみると、まずそれぞれの chunk ごとに 行の合計を計算 → その後 隣り合う chunk 同士の行の合計を取ることで最終的な出力を得ている。
darr.sum(axis=1) # dask.array<p_reduc..., shape=(4,), dtype=int64, chunksize=(2,)> darr.sum(axis=1).compute() # array([10, 26, 42, 58]) darr.sum(axis=1).visualize()

転置のように chunk の位置の入れ替えを伴う処理もできる。Computational Graph 中の四角形 (xxx, 0, 1) は (0, 1) 番目の chunk であることを表す。
darr.T.compute() # array([[ 1, 5, 9, 13], # [ 2, 6, 10, 14], # [ 3, 7, 11, 15], # [ 4, 8, 12, 16]]) darr.T.visualize()

dask.Array 同士の演算もできる。式中では darr.min(axis=1) が重複しているが、これらは Computational Graph 中でマージされ、評価時には 1 度だけ計算される。
((darr - darr.min(axis=1)) / (darr.max(axis=1) - darr.min(axis=1))).compute() # array([[ 0, -1, -2, -3], # [ 1, 0, -1, -2], # [ 2, 1, 0, -1], # [ 4, 3, 2, 1]]) ((darr - darr.min(axis=1)) / (darr.max(axis=1) - darr.min(axis=1))).visualize()

並列処理のメリット
各 chunk に対する処理は 自動的に並列化して実行されるため、ある程度データが大きい場合 / やりたい処理が並列で実行できる場合には速度メリットがある。以下では 長さ 1 億 の ndarray の合計を取る処理で比較した ( EC2 c4.2xlarge を利用)。
# NumPy arr = np.arange(100000000) %timeit arr.sum() # 10 loops, best of 3: 76.5 ms per loop # Dask darr = da.from_array(arr, chunks=10000000) %timeit darr.sum().compute() # 10 loops, best of 3: 25.3 ms per loop
補足 NumPy は主要な処理で GIL を解放しているため、Dask での並列処理は threading によって行われる。処理方法として multiprocessing を指定することもできる。
Out-Of-Core 処理のメリット
Dask では Computational Graph 中の各ノードを順に計算していくため、処理する全データが同時にメモリに乗っている必要がない。そのため、PC の物理メモリを超える大きさのデータも扱うことができる。以下のドキュメントでは複数の pkl ファイルを chunk として読み込む方法が記載されている。
関連パッケージ
以下のパッケージでは バックグラウンド処理を dask.array を利用して行うことができる。
Xray: 多次元ラベルデータをpandasのように処理できるパッケージ
まとめ
簡単に並列 / Out-Of-Core 処理を行うためのパッケージである Dask のサブモジュールのうち NumPy 互換の dask.array の使い方を記載した。

- 作者: Micha Gorelick,Ian Ozsvald,相川愛三
- 出版社/メーカー: オライリージャパン
- 発売日: 2015/11/20
- メディア: 大型本
- この商品を含むブログ (3件) を見る
{purrr} でリストデータを操作する <2>
前の記事に続けて、{purrr} で {rlist} 相当の処理を行う。今回はレコードの選択とソート。
サンプルデータは前回と同じものを利用する。リストの表示も同じく Hmisc::list.tree を使う。
library(rlist) library(pipeR) library(purrr) packages <- list( list(name = 'dplyr', star = 979L, maintainer = 'hadley' , authors = c('hadley', 'romain')), list(name = 'ggplot2', star = 1546L, maintainer = 'hadley' , authors = c('hadley')), list(name = 'knitr', star = 1047L, maintainer = 'yihui' , authors = c('yihui', 'hadley', '...and all')) ) packages ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[3]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
レコードの選択
rlist Tutorial: Filtering 後半の内容。
ある条件にあてはまるレコードのうち最初の N 件だけを取得したいとき、{rlist} には専用の関数 list.find がある。{purrr} には対応する関数はないが、keep %>% head で同じ結果が得られる。
packages %>>% rlist::list.find(star >= 1000, 1) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::keep(~ .$star >= 1000) %>% head(n = 1) # 略
また、{rlist} には list.findi という インデックスを返す関数がある。purrr::keep では、引数に logical 型のベクトルを渡せば TRUE に対応する要素のみを残すことができる。
packages %>>% rlist::list.findi(star >= 1000, 2) ## [1] 2 3 purrr::keep(seq_along(packages), flatmap(packages, ~ .$star >= 1000)) %>% head(n = 2) # 略
条件にあてはまる最初のレコードだけを取得したいときは rlist::first もしくは purrr::detect。
rlist::list.first(packages, star >= 1000) ## x = list 4 (792 bytes) ## . name = character 1= ggplot2 ## . star = integer 1= 1546 ## . maintainer = character 1= hadley ## . authors = character 1= hadley purrr::detect(packages, ~ .$star >= 1000) # 略
レコード数がそこまで多くない場合は keep %>% head(n = 1) でよいと思うが、結果に 1 レベル目が入れ子として残ってしまう。上と同じ結果を得るには purrr::flatten ( unlist(recursive = FALSE) と同じ ) を通す。
purrr::keep(packages, ~ .$star >= 1000) %>% head(n = 1) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley purrr::keep(packages, ~ .$star >= 1000) %>% head(n = 1) %>% purrr::flatten() ## x = list 4 (792 bytes) ## . name = character 1= ggplot2 ## . star = integer 1= 1546 ## . maintainer = character 1= hadley ## . authors = character 1= hadley
同じく、最後のレコードを取得したいときは rlist::last もしくは purrr::detect(.right = TRUE)。これも自分としては keep %>% tail(n = 1) のほうが覚えやすい。
list.last(packages, star >= 1000) ## x = list 4 (920 bytes) ## . name = character 1= knitr ## . star = integer 1= 1047 ## . maintainer = character 1= yihui ## . authors = character 3= yihui hadley ... purrr::detect(packages, ~ .$star >= 1000, .right = TRUE) # 略 purrr::keep(packages, ~ .$star >= 1000) %>% tail(n = 1) %>% purrr::flatten() # 略
類似の処理として、{rlist} には list.take という最初の N レコードを取得する関数と list.skip という最初の N レコードを除外する関数がある。{purrr} には対応する関数はないが、組み込みの head と tail でよいのでは...。
packages %>>% rlist::list.take(2) ## x = list 2 (1696 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% head(n = 2) # 略 packages %>% rlist::list.skip(1) ## x = list 2 (1768 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... packages %>% tail(length(.) - 1) # 略
ある条件が初めて偽となるまでレコードを取得する場合は rlist::list.takeWhile もしくは purrr::head_while。
packages %>>% rlist::list.takeWhile(star <= 1500) ## x = list 1 (896 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain packages %>% purrr::head_while(~ .$star <= 1500) # 略
ある条件が初めて偽となったレコード以降を取得したい場合は rlist::list.skipWhile。{purrr} には対応する処理はないが、purrr::detect_index を使って以下のように書ける。
packages %>>% rlist::list.skipWhile(star <= 1500) ## x = list 2 (1768 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... purrr::detect_index(packages, ~ .$star > 1500) ## [1] 2 packages[purrr::detect_index(packages, ~ .$star > 1500):length(packages)] # 略
一方、{purrr} には tail_while という末尾からみて条件にあてはまるレコードだけを取得する関数がある。
packages %>% purrr::tail_while(~ .$star <= 1500) ## x = list 1 (968 bytes) ## . [[1]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
ある条件を満たす要素がいくつあるかを調べるには rlist::list.count。{purrr} なら keep %>% length でよい。
list.count(packages, star > 1000) ## [1] 2 purrr::keep(packages, ~ .$star > 1000) %>% length() # 略
{rlist} には リストの要素の名前を正規表現で抽出する list.match がある。{purrr} なら keep でできる。stringr::str_match はマッチした結果を matrix で返すため、complete.cases で logical に変換する。
data <- list(p1 = 1, p2 = 2, a1 = 3, a2 = 4) list.match(data, "p[12]") ## x = list 2 (416 bytes) ## . p1 = double 1= 1 ## . p2 = double 1= 2 complete.cases(stringr::str_match(names(data), 'p[12]')) ## [1] TRUE TRUE FALSE FALSE keep(data, complete.cases(stringr::str_match(names(data), 'p[12]'))) # 略
logical の処理
条件に当てはまるかどうかを logical ベクトルとして返すには rlist::list.is。これは rlist::list.mapv と同じなのでは...? {purrr} なら flatmap もしくは map_lgl。
packages %>>% rlist::list.is(star < 1500) ## [1] TRUE FALSE TRUE packages %>>% rlist::list.mapv(star < 1500) # 略 packages %>% purrr::flatmap(~ .$star < 1500) # 略 packages %>% purrr::map_lgl(~ .$star < 1500) # 略
また、logical のリスト全体の真偽値を以下のように取得できる
- 要素全てが
TRUEかどうか:rlist::list.allもしくはpurrr::every。 - 要素いずれかが
TRUEかどうか:rlist::list.anyもしくはpurrr::some。
自分は purrr::flatmap %>% all もしくは purrr::flatmap %>% any のほうが覚えやすい。
ただし、{purrr} v0.1.0 の every, some にはバグがあり正しく動作しない。下のスクリプトを実行するためには GitHub から最新の master をインストールする必要がある。
rlist::list.all(packages, star >= 500) ## [1] TRUE # install_github('hadley/purrr') # が必要 purrr::every(packages, ~ .$star >= 500) # 略 packages %>% purrr::flatmap(~ .$star >= 500) %>% all() # 略 rlist::list.any(packages, star >= 1500) ## [1] TRUE # install_github('hadley/purrr') # が必要 purrr::some(packages, ~ .$star >= 1500) packages %>% purrr::flatmap(~ .$star >= 1500) %>% any() # 略
要素の削除
{rlist} では list.remove で指定した名前をリストから除外できる。{purrr} には discard という条件にマッチする要素をリストから除外する関数がある ( purrr::keep とは逆 )。条件の否定をとれば keep でも書ける。
rlist::list.remove(data, c("p1", "p2")) ## x = list 2 (416 bytes) ## . a1 = double 1= 3 ## . a2 = double 1= 4 purrr::discard(data, names(data) %in% c("p1", "p2")) # 略 purrr::keep(data, !(names(data) %in% c("p1", "p2"))) # 略
{rlist} で、名前ではなく 条件式を使ってリストから除外したい場合は list.exclude。{purrr} の場合は 上と同じく discard もしくは keep。
packages %>>% rlist::list.exclude("yihui" %in% authors) ## x = list 2 (1696 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::discard(~ "yihui" %in% .$authors) # 略
また、{rlist} には 欠損などの異常値を リストから除去する list.clean という関数がある。下の例では リストの 1 レベル目にある欠損 "b" を除去している。{purrr} では同じく discard。
x <- list(a = 1, b = NULL, c = list(x = 1, y = NULL, z = logical(0L), w = c(NA, 1))) x ## x = list 3 (1040 bytes) ## . a = double 1= 1 ## . b = NULL 0 ## . c = list 4 ## . . x = double 1= 1 ## . . y = NULL 0 ## . . z = logical 0 ## . . w = double 2= NA 1 rlist::list.clean(x) ## x = list 2 (960 bytes) ## . a = double 1= 1 ## . c = list 4 ## . . x = double 1= 1 ## . . y = NULL 0 ## . . z = logical 0 ## . . w = double 2= NA 1 purrr::discard(x, ~ is.null(.)) # 略
rlist::list.clean は recursive = TRUE を指定することで 処理を再帰的に適用できる。{purrr} ではこういった再帰的な処理は (自分で関数を書かない限り) できないと思う。
rlist::list.clean(x, recursive = TRUE) ## x = list 2 (912 bytes) ## . a = double 1= 1 ## . c = list 3 ## . . x = double 1= 1 ## . . z = logical 0 ## . . w = double 2= NA 1
要素の更新
rlist Tutorial: Updating に相当する内容。
rlist::list.map や purrr::map を用いたリストの選択では、結果に含める属性を明示的に指定する必要があった。
これは対象のリストに選択したい属性が多い場合はすこし面倒だ。
rlist::list.update を使うと、もともとのリストの属性を残した上で 指定した要素を追加 / 更新できる。{purrr} で同じことをするには map + update_list。
packages %>>% rlist::list.update(lang = 'R', star = star + 1) ## x = list 3 (3160 bytes) ## . [[1]] = list 5 ## . . name = character 1= dplyr ## . . star = double 1= 980 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . . lang = character 1= R ## . [[2]] = list 5 ## . . name = character 1= ggplot2 ## . . star = double 1= 1547 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . . lang = character 1= R ## . [[3]] = list 5 ## . . name = character 1= knitr ## . . star = double 1= 1048 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . . lang = character 1= R packages %>% purrr::map(~ purrr::update_list(., lang = 'R', star = ~ .$star + 1)) # 略
一部の要素を取り除きたい場合は NULL を指定する。これは {purrr} も同じ。
packages %>>% rlist::list.update(maintainer = NULL, authors = NULL) ## x = list 3 (1464 bytes) ## . [[1]] = list 2 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . [[2]] = list 2 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . [[3]] = list 2 ## . . name = character 1= knitr ## . . star = integer 1= 1047 packages %>% purrr::map(~ purrr::update_list(., maintainer = NULL, authors = NULL)) # 略
ソート
rlist Tutorial: Sorting に相当する内容。
{rlist}, {purrr} とも 標準の sort ( 値のソート ) と order ( インデックスのソート ) それぞれに対応した関数を持つ。
x = c('a', 'c', 'd', 'b') sort(x) ## [1] "a" "b" "c" "d" order(x) ## [1] 1 4 2 3
sort のようにソートした値を得るためには rlist::list.sort もしくは purrr::sort_by。
packages %>>% rlist::list.sort(star) ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . [[3]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::sort_by('star') # 略
{rlist} では 列名を括弧でくくると逆順となる。{purrr} には対応する記法がないが、結果を rev で逆順にすればよい。
packages %>>% rlist::list.sort((star)) ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . [[3]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain packages %>% purrr::sort_by('star') %>% rev() # 略
order のように結果をインデックスで得るためには rlist::list.order もしくは purrr::order_by。
rlist::list.order(packages, star) ## [1] 1 3 2 purrr::order_by(packages, 'star') ## [1] 1 3 2
まとめ
前回と同じく、{purrr} では map (flatmap)、keep (discard) でだいたいのことはできる。処理を少し変えたい場合は 組み込み関数を適当に組み合わせればよいため、追加での学習コストは低いと思う。
一方、{rlist} はパッケージの中だけで処理が完結するので、そちらにメリットを感じる方は {rlist} を使ったほうがよさそう。
さらに続きます。

- 作者: Hadley Wickham,石田基広,市川太祐,高柳慎一,福島真太朗
- 出版社/メーカー: 共立出版
- 発売日: 2016/01/23
- メディア: 単行本
- この商品を含むブログ (25件) を見る
{purrr} でリストデータを操作する <1>
R で関数型プログラミングを行うためのパッケージである {purrr}、すこし使い方がわかってきたので整理をしたい。RStudio のブログの記載をみると、とくにデータ処理フローを関数型のように記述することが目的のようだ。
The core of purrr is a set of functions for manipulating vectors (atomic vectors, lists, and data frames). The goal is similar to dplyr: help you tackle the most common 90% of data manipulation challenges.
ここでいう"関数型プログラミング言語"とは Haskell のような静的型の言語を想定しており、型チェック、ガード、リフトなど断片的に影響を受けたと思われる関数群をもつ。ただし、同時に Haskell を目指すものではない、とも明言されている。
そもそも R は Scheme の影響をうけているためか、関数型らしい言語仕様やビルトイン関数群 Map, Reduce, Filter などをすでに持っている。ただ、これらの関数群は引数の順序からパイプ演算子と組み合わせて使いにくい。{purrr} でこのあたりが使いやすくなっているとうれしい。
R: Common Higher-Order Functions in Functional Programming...
※ 上記の Map など、S のリファレンスに見当たらなかったため R 独自だと思っていますが、間違っていたら教えてください。
{purrr} によるリストの操作
{purrr} を使うとリストやベクトルの処理が書けるのかをまとめたい。リスト操作のためのパッケージである {rlist} とできること、記法をくらべてみる。
{purrr} を利用する目的は 処理を簡単に、見やすく書くことであるため、{rlist} と比べて可読性が悪いもの、複雑になるものは "できない" 処理として扱う。サンプルデータとして、R の著名パッケージに関連した情報のリストを用意した。
packages <- list( list(name = 'dplyr', star = 979L, maintainer = 'hadley' , authors = c('hadley', 'romain')), list(name = 'ggplot2', star = 1546L, maintainer = 'hadley' , authors = c('hadley')), list(name = 'knitr', star = 1047L, maintainer = 'yihui' , authors = c('yihui', 'hadley', '...and all')) ) packages ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[3]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
1レベル目がレコード、以降のレベルが各レコードの要素となっている。この記事では主に以下二つの操作に関連した機能について記載する。
- 要素に対する操作: 特定の要素を選択する。特定の要素を元に新しい要素をつくる (マッピングする)。
- レコードに対する操作: 特定のレコードを選択する。
準備: リストの表示
既定の print ではリストの階層構造がわかりにくく、出力も長い。わかりやすくするため Hmisc::list.tree を利用した出力を記載する。また、出力が同一のものは省略する。
library(Hmisc) l1 <- list(a = 1, b = c(3L, 4L), c = list(x = c(5, 6), y = 'AAA')) # 既定での表示 l1 ## $a ## [1] 1 ## ## $b ## [1] 3 4 ## ## $c ## $c$x ## [1] 5 6 ## ## $c$y ## [1] "AAA" # list.tree での表示 (名前 = 型 要素数 = 値 と読む) Hmisc::list.tree(l1) ## l1 = list 3 (968 bytes) ## . a = double 1= 1 ## . b = integer 2= 3 4 ## . c = list 2 ## . . x = double 2= 5 6 ## . . y = character 1= AAA
パッケージのロード
library(rlist) library(pipeR) library(purrr) library(dplyr)
要素の選択とマッピング
rlist Tutorial: Mapping に相当する内容。
要素の選択
R 標準の lapply 他 apply 系の関数に対応するのは rlist::list.map と purrr::map。purrr::map では第二引数に文字列を渡すとその要素の抽出、ラムダ式 ( ~ .$name )を渡すとその式の適用になる。
lapply(packages, function(x) { x$name }) ## x = list 3 (360 bytes) ## . [[1]] = character 1= dplyr ## . [[2]] = character 1= ggplot2 ## . [[3]] = character 1= knitr rlist::list.map(packages, name) # 略 purrr::map(packages, 'name') # 略 purrr::map(packages, ~ .$name) # 略
複数の要素を一度に選択したい場合、{rlist} には rlist::list.select という選択専用の関数がある。{purrr} では渡せる式が一つのため、ラムダ式を利用する。
rlist::list.select(packages, maintainer, star) ## x = list 3 (1488 bytes) ## . [[1]] = list 2 ## . . maintainer = character 1= hadley ## . . star = integer 1= 979 ## . [[2]] = list 2 ## . . maintainer = character 1= hadley ## . . star = integer 1= 1546 ## . [[3]] = list 2 ## . . maintainer = character 1= yihui ## . . star = integer 1= 1047 purrr::map(packages, ~ .[c('maintainer', 'star')]) # 略
補足 第二引数にベクトルを渡した場合の処理は、以下のように階層的な選択になる ( l[[c('a', 'b')]] と一緒)。
l2 <- list(list(a=list(b=1)), list(a=list(b=2))) l2 ## x = list 2 (1176 bytes) ## . [[1]] = list 1 ## . . a = list 1 ## . . . b = double 1= 1 ## . [[2]] = list 1 ## . . a = list 1 ## . . . b = double 1= 2 purrr::map(l2, c('a', 'b')) ## x = list 2 (152 bytes) ## . [[1]] = double 1= 1 ## . [[2]] = double 1= 2
{rlist}, {purrr} でのラムダ式
f <- function(.) {. + 1} に対応する無名関数を {rlist}, {purrr} それぞれの記法で書く。形式はチルダの有無以外は同じ。ドットが引数に対応する。
nums <- c(a = 3, b = 2, c = 1) rlist::list.map(nums, . + 1) ## x = list 3 (544 bytes) ## . a = double 1= 4 ## . b = double 1= 3 ## . c = double 1= 2 purrr::map(nums, ~ . + 1) # 略
{rilst} では ラムダ式中の .i で元のリストの位置 (インデックス) を、.name で名前 (names) をそれぞれ参照できる。purrr のラムダ式には対応する記法がないため、近いことをやるためには直接 map の引数として渡す必要がある。
rlist::list.map(nums, .i) ## x = list 3 (544 bytes) ## . a = integer 1= 1 ## . b = integer 1= 2 ## . c = integer 1= 3 purrr::map(seq_along(nums), ~ .) # namesは変わってしまう ## x = list 3 (216 bytes) ## . [[1]] = integer 1= 1 ## . [[2]] = integer 1= 2 ## . [[3]] = integer 1= 3
rlist::list.map(nums, paste0("Name: ", .name)) ## x = list 3 (688 bytes) ## . a = character 1= Name: a ## . b = character 1= Name: b ## . c = character 1= Name: c purrr::map(names(nums), ~ paste0("Name: ", .)) # namesは変わってしまう ## x = list 3 (360 bytes) ## . [[1]] = character 1= Name: a ## . [[2]] = character 1= Name: b ## . [[3]] = character 1= Name: c
また、内部的にはラムダ式を eval で評価をしているため、変数として扱われない位置にあるドットは評価されない。
purrr::map(nums, ~ list(. = 1)) ## x = list 3 (1312 bytes) ## . a = list 1 ## . . . = double 1= 1 ## . b = list 1 ## . . . = double 1= 1 ## . c = list 1 ## . . . = double 1= 1
要素の追加、変更
元となるリストの値から新しいリストを作りたい場合はラムダ式でリストを返す。
rlist::list.map(packages, list(star = star, had = 'hadley' %in% authors)) ## x = list 3 (1320 bytes) ## . [[1]] = list 2 ## . . star = integer 1= 979 ## . . had = logical 1= TRUE ## . [[2]] = list 2 ## . . star = integer 1= 1546 ## . . had = logical 1= TRUE ## . [[3]] = list 2 ## . . star = integer 1= 1047 ## . . had = logical 1= TRUE purrr::map(packages, ~ list(star = .$star, had = 'hadley' %in% .$authors)) # 略
結果の型の変更
結果をベクトルで取得したい場合、{rilst} では list.mapv。{purrr} では flatmap もしくは map_int ( map_xxx のように結果の型を指定できる関数群がある )。
rlist::list.mapv(packages, star) ## [1] 979 1546 1047 purrr::flatmap(packages, 'star') # 略 purrr::map_int(packages, 'star') # 略
data.frame としたい場合は rlist::list.stack。{purrr} では dplyr::bind_rows に渡す。
packages %>>% rlist::list.select(name, star) %>>% rlist::list.stack() ## name star ## 1 dplyr 979 ## 2 ggplot2 1546 ## 3 knitr 1047 packages %>% purrr::map(~ .[c('name', 'star')]) %>% dplyr::bind_rows()) # 略
関数の適用
返り値を変更せずに関数を適用するには rlist::list.iter と purrr::walk。パイプ処理の間に出力やプロットなど意図する返り値を持たない処理を挟み込む場合に利用する。
r <- rlist::list.iter(packages, cat(name, ":", star, "\n")) ## dplyr : 979 ## ggplot2 : 1546 ## knitr : 1047 r <- purrr::walk(packages, ~ cat(.$name, ":", .$star, "\n")) # 略
レコードの選択
rlist Tutorial: Filtering 前半の単純な条件での選択。
{rilst} では list.filter。{purrr} では keep。ラムダ式が使えるのは map などと同じ。
packages %>>% list.filter(star >= 1500) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::keep(~ .$star >= 1500) # 略
packages %>>% list.filter("yihui" %in% authors) ## x = list 1 (968 bytes) ## . [[1]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... packages %>% purrr::keep(~ "yihui" %in% .$authors) # 略
まとめ
{purrr} によるリストデータの属性、レコードに対する操作を記載した。purrr::map と purrr::keep だけでもパイプ演算子 + ラムダ式と組み合わせて幅広い処理ができそうだ。
11/28追記 続きです。

- 作者: Hadley Wickham,石田基広,市川太祐,高柳慎一,福島真太朗
- 出版社/メーカー: 共立出版
- 発売日: 2016/01/23
- メディア: 単行本
- この商品を含むブログ (25件) を見る
Python xray で 多次元データを pandas ライクに扱う
はじめに
pandas では 2 次元、表形式のデータ ( DataFrame ) を主な対象としているが、ときには 3 次元以上のデータを扱いたい場合がある。そういった場合 以下のような方法がある。
MultiIndexを使い、2 次元のデータにマッピングする。- 3 次元データ構造である
Panel、4 次元のPanel4D、もしくは任意の次元のデータ構造 (PanelND) をファクトリ関数 で定義して使う。 numpy.ndarrayのまま扱う。
自分は MultiIndex を使うことが多いが、データを 2 次元にマップしなければならないため 種類によっては直感的に扱いにくい。Panel や PanelND は DataFrame と比べると開発が活発でなく、特に Panel4D、PanelND は 現時点で Experimental 扱いである。また、今後の扱いをどうするかも議論がある。numpy.ndarray では データのラベル付けができない。
xray とは
ラベル付きの多次元データを多次元のまま 直感的に扱えるパッケージとして xray がある。作者は pandas 開発チーム仲間の shoyer だ。そのため、API は pandas にかなり近いものになっている。
xray は大きく以下ふたつのデータ構造を持つ。これらを使うと多次元データをより直感的に操作することができる。
xray.DataArray:numpyの多次元配列にラベルでのアクセスを追加したもの。データは任意の次元を持つことができる。xray.Dataset: 複数のxray.DataArrayをまとめるセット。
インストール
pip で。
$ pip install xray
データの準備
まず、必要なパッケージをインポートする。
import numpy as np import pandas as pd pd.__version__ # '0.16.2' import xray xray.__version__ # '0.5.2'
サンプルデータとして、気象庁から 2015年7月20日〜25日の東京、八王子、大島の最高気温のデータを使いたい。以下のサイトからダウンロードした。データは 日付、場所 2 次元の配列となる。
- 出典:気象庁ホームページ (URL: http://www.data.jma.go.jp/gmd/risk/obsdl/index.php)

data2 = np.array([[34.2, 30.2, 33.5], [36.0, 29.3, 34.9], [35.3, 29.7, 32.8], [30.1, 27.6, 30.4], [33.6, 30.1, 33.9], [34.1, 28.0, 33.1]])
この numpy.ndarray から xray.DataArray インスタンスを作成する。データへアクセスするためのラベル ( pandas でいう index のようなもの ) は coords キーワードで指定する。また、dims キーワードを使って各次元それぞれにも名前をつけることができる。ここでは 以下のような DataArray を作成している。
- データは 2 つの次元
date,locationを持つ。 date次元はdatesで指定される 6 つの日付のラベルを持つ。location次元はlocsで指定される 3 つの場所のラベルを持つ。
2 次元のため、pandas.DataFrame でいうと date が indexに、 location が columns に対応しているイメージ。
locs = [u'八王子', u'大島', u'東京'] dates = pd.date_range('2015-07-20', periods=6, freq='D') da2 = xray.DataArray(data2, coords=[dates, locs], dims=['date', 'location']) da2 # <xray.DataArray (date: 6, location: 3)> # array([[ 34.2, 30.2, 33.5], # [ 36. , 29.3, 34.9], # [ 35.3, 29.7, 32.8], # [ 30.1, 27.6, 30.4], # [ 33.6, 30.1, 33.9], # [ 34.1, 28. , 33.1]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 各次元の名前 da2.dims # ('date', 'location') # 各次元の詳細 da2.coords # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # date 次元のラベル da2.coords['date'] # <xray.DataArray 'date' (date: 6)> # array(['2015-07-20T09:00:00.000000000+0900', # '2015-07-21T09:00:00.000000000+0900', # '2015-07-22T09:00:00.000000000+0900', # '2015-07-23T09:00:00.000000000+0900', # '2015-07-24T09:00:00.000000000+0900', # '2015-07-25T09:00:00.000000000+0900'], dtype='datetime64[ns]') # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ...
データの選択
xray でのデータ選択は pandas と類似の方法で行える。pandas でのデータ選択についてはこちらを。
ある次元から、特定のラベルをもつデータを選択したい場合、pandas と同じく .loc が使える。
# 7/25 のデータを選択 da2.loc[pd.Timestamp('2015-07-25')] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
また、対象の次元が datetime64 型の場合は、pandas と同じく日時文字列でも指定が可能 (部分文字列によるスライシング、詳細以下)。
# 7/25 のデータを選択 da2.loc['2015-07-25'] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
また、ラベルではなく位置 ( n番目など ) によって選択する場合は __getitem__ を使う。pandas では .iloc に対応。
# 末尾 = 最新の日付のデータを取得 da2[-1] # <xray.DataArray (location: 3)> # array([ 34.1, 28. , 33.1]) # Coordinates: # date datetime64[ns] 2015-07-25 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
2 次元目以降をラベル / 位置によって指定する場合は、引数をカンマで区切り、選択に利用する次元の位置に対応する値を渡す。
# date は全選択、location が東京のデータを選択 da2.loc[:, u'東京'] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # date は全選択、location が 3 番目 = 東京のデータを選択 da2[:, 2] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac'
データ選択に利用する次元自体もラベルで指定したい場合は .sel を使う。.sel では次元の順序に関係なく値を指定できるため、高次元になった場合もシンプルだ。
# 東京のデータを選択 da2.sel(location=u'東京') # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # 7/22, 7/23 の 東京のデータを選択 da2.sel(location=u'東京', date=pd.DatetimeIndex(['2015-07-22', '2015-07-23'])) # <xray.DataArray (date: 2)> # array([ 32.8, 30.4]) # Coordinates: # * date (date) datetime64[ns] 2015-07-22 2015-07-23 # location <U3 u'\u6771\u4eac'
もしくは .loc, __getitem__ に以下のような辞書を渡してもよい。
# 東京のデータを選択 da2.loc[dict(location=u'東京')] # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac'
公式ドキュメント では、xray.DataArray からのデータ選択を以下のような表で整理している。
| 次元の指定 | 値の指定 | DataArray のメソッド |
|---|---|---|
| 位置 | 位置 | da2[:, 0] |
| 位置 | ラベル | da2.loc[:, u'東京'] |
| ラベル | 位置 | da2.isel(location=0), da2[dict(space=0)] |
| ラベル | ラベル | da2.sel(location=u'東京'), da2.loc[dict(location=u'東京')] |
次元の追加
ここまでは 2 次元のデータを使っていたが、最低気温と平均気温のデータを追加して 3 次元のデータとする。
# 最低気温 data3 = np.array([[24.7, 25.1, 25.8], [23.4, 24.3, 25.4], [22.7, 23.8, 25.4], [24.5, 24.4, 24.7], [24.9, 24.6, 25.0], [23.7, 24.8, 24.8]]) # 平均気温 data4 = np.array([[27.8, 26.5, 28.8], [29.7, 26.3, 29.4], [29.5, 26.5, 28.9], [26.9, 25.3, 27.0], [27.7, 26.4, 28.1], [29.0, 26.1, 28.6]]) data = np.dstack([data2, data3, data4]) # 日時、場所、データの種類 の 3 次元 data.shape # (6, 3, 3) da3 = xray.DataArray(data, coords=[dates, locs, [u'最高', u'最低', u'平均']], dims=['date', 'location', 'type']) da3 # <xray.DataArray (date: 6, location: 3, type: 3)> # array([[[ 34.2, 24.7, 27.8], # [ 30.2, 25.1, 26.5], # [ 33.5, 25.8, 28.8]], # # ... # # [[ 34.1, 23.7, 29. ], # [ 28. , 24.8, 26.1], # [ 33.1, 24.8, 28.6]]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
3 次元以上の場合もデータ選択のルールは一緒なのでわかりやすい。
# 7/24 のデータを選択 da3.loc['2015-07-24'] # <xray.DataArray (location: 3, type: 3)> # array([[ 33.6, 24.9, 27.7], # [ 30.1, 24.6, 26.4], # [ 33.9, 25. , 28.1]]) # Coordinates: # date datetime64[ns] 2015-07-24 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # 7/24 の 最高気温のデータを選択 da3.loc['2015-07-24', :, u'最高'] # <xray.DataArray (location: 3)> # array([ 33.6, 30.1, 33.9]) # Coordinates: # date datetime64[ns] 2015-07-24 # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # type <U2 u'\u6700\u9ad8' # 東京 の 最高気温のデータを選択 da3.sel(location=u'東京', type=u'最高') # <xray.DataArray (date: 6)> # array([ 33.5, 34.9, 32.8, 30.4, 33.9, 33.1]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u6771\u4eac' # type <U2 u'\u6700\u9ad8'
算術演算
pandas と同じく、xray.DataArray 同士での算術演算が可能。各日の最高気温と最低気温の差を求めると、
da3.sel(type=u'最高') - da3.sel(type=u'最低') # <xray.DataArray (date: 6, location: 3)> # array([[ 9.5, 5.1, 7.7], # [ 12.6, 5. , 9.5], # [ 12.6, 5.9, 7.4], # [ 5.6, 3.2, 5.7], # [ 8.7, 5.5, 8.9], # [ 10.4, 3.2, 8.3]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 結果から 八王子 のデータだけを選択 (da3.sel(type=u'最高') - da3.sel(type=u'最低')).sel(location=u'八王子') # <xray.DataArray (date: 6)> # array([ 9.5, 12.6, 12.6, 5.6, 8.7, 10.4]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location <U3 u'\u516b\u738b\u5b50'
データのグループ化 / 集約
グループ化 / 集約も pandas とほぼ同じ形式でできる。location によってグループ化し、期間中の最高気温を出してみる。
da3.groupby('location') # <xray.core.groupby.DataArrayGroupBy at 0x109a56f90> da3.groupby('location').max() # <xray.DataArray (location: 3)> # array([ 36. , 30.2, 34.9]) # Coordinates: # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
グループ化した結果は、イテレーションによって順番に処理することもできる。
for name, g in da3.groupby('location'): print(name) print(g) # 八王子 # <xray.DataArray (date: 6, type: 3)> # array([[ 34.2, 24.7, 27.8], # [ 36. , 23.4, 29.7], # [ 35.3, 22.7, 29.5], # [ 30.1, 24.5, 26.9], # [ 33.6, 24.9, 27.7], # [ 34.1, 23.7, 29. ]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # location <U4 u'\u516b\u738b\u5b50' # # 以降略
データの結合 / 連結
上で作成した DataArray に、別のデータを追加したい。適当なデータを探したところ、ある成人男性のお住まいの気温データ を見つけた。これを日別で集計して連結したい。
# GitHub からデータを取得 df = pd.read_csv('https://raw.githubusercontent.com/dichika/mydata/master/room.csv') df['time'] = pd.to_datetime(df['time']) # light は光量、temperatur は気温 df.head() # time light temperature # 0 2015-02-12 01:45:04 463.0 26.784 # 1 2015-02-12 02:00:03 473.0 26.630 # 2 2015-02-12 02:15:04 467.9 25.983 # 3 2015-02-12 02:30:04 0.0 25.453 # 4 2015-02-12 02:45:04 0.0 23.650 # 期間中にフィルタ df = df[df['time'] >= pd.Timestamp('2015-07-20')] # 日時でグループ化 / 集約 agg = df.groupby(pd.Grouper(key='time', freq='D'))['temperature'].agg(['max', 'min', 'mean']) agg # max min mean # time # 2015-07-20 28.374 26.450 27.484604 # 2015-07-21 33.790 26.800 30.132792 # 2015-07-22 34.180 27.070 30.134375 # 2015-07-23 28.779 27.412 28.290918
xray でデータを連結するためには、連結する次元 (ここでは location) 以外のデータの要素数を一致させる必要がある。上記のデータは数日遅れで公開されているため、直近を NaN でパディングして xray.DataArray を作成する。
agg.loc[pd.Timestamp('2015-07-24'), :] = np.nan agg.loc[pd.Timestamp('2015-07-25'), :] = np.nan agg # max min mean # time # 2015-07-20 28.374 26.450 27.484604 # 2015-07-21 33.790 26.800 30.132792 # 2015-07-22 34.180 27.070 30.134375 # 2015-07-23 28.779 27.412 28.290918 # 2015-07-24 NaN NaN NaN # 2015-07-25 NaN NaN NaN d = xray.DataArray(agg.values.reshape(6, 1, 3), coords=[agg.index, [u'誰かの家'], [u'最高', u'最低', u'平均']], dims=['date', 'location', 'type']) d # <xray.DataArray (date: 6, location: 1, type: 3)> # array([[[ 28.374 , 26.45 , 27.48460417]], # [[ 33.79 , 26.8 , 30.13279167]], # [[ 34.18 , 27.07 , 30.134375 ]], # [[ 28.779 , 27.412 , 28.29091765]], # [[ nan, nan, nan]], # [[ nan, nan, nan]]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U4 u'\u8ab0\u304b\u306e\u5bb6' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
データの連結は xray.concat で可能。新しい場所のデータを追加 (連結) したいので、対象の次元として location を指定する。
da3 = xray.concat([da3, d], dim='location') da3.sel(location=u'誰かの家') # <xray.DataArray (date: 6, type: 3)> # array([[ 28.374 , 26.45 , 27.48460417], # [ 33.79 , 26.8 , 30.13279167], # [ 34.18 , 27.07 , 30.134375 ], # [ 28.779 , 27.412 , 28.29091765], # [ nan, nan, nan], # [ nan, nan, nan]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # location <U4 u'\u8ab0\u304b\u306e\u5bb6'
ほか、merge で結合もできる。
Dataset の利用
xray では、DataArray クラスを複数まとめて Dataset クラスとして扱うことができる。元となる DataArray は同じ次元でなくてもよい。
Dataset を作成するため、降水量、湿度 それぞれ 2 次元の DataArray を用意する。
# 降水量 precip = np.array([[0, np.nan, 0], [0, np.nan, np.nan], [0, 0, np.nan], [6.5, 1, 4.5], [30.0, 0, 7.0], [0, np.nan, np.nan]]) precip = xray.DataArray(precip, coords=[dates, locs], dims=['date', 'location']) precip # <xray.DataArray (date: 6, location: 3)> # array([[ 0. , nan, 0. ], # [ 0. , nan, nan], # [ 0. , 0. , nan], # [ 6.5, 1. , 4.5], # [ 30. , 0. , 7. ], # [ 0. , nan, nan]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # 湿度 humid = np.array([[np.nan, 88, 75], [np.nan, 85, 65], [np.nan, 87, 61], [np.nan, 91, 80], [np.nan, 86, 83], [np.nan, 88, 80]]) humid = xray.DataArray(humid, coords=[dates, locs], dims=['date', 'location']) humid # <xray.DataArray (date: 6, location: 3)> # array([[ nan, 88., 75.], # [ nan, 85., 65.], # [ nan, 87., 61.], # [ nan, 91., 80.], # [ nan, 86., 83.], # [ nan, 88., 80.]]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
これらと気温データをあわせて xray.Dataset を作成する。Dataset が持つ次元は Dimenstions, Coordinates に表示される。
Dataset に含まれる DataArray は Data variables 中に表示され、それぞれどの次元を含んでいるかがわかる。元データと同じく、気温は 3 次元、ほかは 2 次元のデータとなっている。
ds = xray.Dataset({'temperature': da3,
'precipitation': precip,
'humidity': humid})
ds
# <xray.Dataset>
# Dimensions: (date: 6, location: 4, type: 3)
# Coordinates:
# * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ...
# * location (location) object u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ...
# * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747'
# Data variables:
# precipitation (date, location) float64 0.0 nan 0.0 nan 0.0 nan nan nan ...
# temperature (date, location, type) float64 34.2 24.7 27.8 30.2 25.1 ...
# humidity (date, location) float64 nan 88.0 75.0 nan nan 85.0 65.0 ...
ds.data_vars
# Data variables:
# precipitation (date, location) float64 0.0 nan 0.0 nan 0.0 nan nan nan ...
# temperature (date, location, type) float64 34.2 24.7 27.8 30.2 25.1 ...
# humidity (date, location) float64 nan 88.0 75.0 nan nan 85.0 65.0 ...
Dataset からのデータ選択は、Dataset に含まれるすべての DataArray に対して行われる。DataArray とは異なり、次元は必ずラベルで指定する必要がある。
| 次元の指定 | 値の指定 | Dataset のメソッド |
|---|---|---|
| 位置 | 位置 | なし |
| 位置 | ラベル | なし |
| ラベル | 位置 | ds.isel(location=2), ds[dict(location=2)] |
| ラベル | ラベル | ds.sel(location=u'東京'), ds.loc[dict(location=u'東京')] |
# 東京 のデータを選択 ds.sel(location=u'東京') # <xray.Dataset> # Dimensions: (date: 6, type: 3) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u6771\u4eac' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # Data variables: # precipitation (date) float64 0.0 nan nan 4.5 7.0 nan # temperature (date, type) float64 33.5 25.8 28.8 34.9 25.4 29.4 32.8 ... # humidity (date) float64 75.0 65.0 61.0 80.0 83.0 80.0 # 東京 の 平均気温 を選択 ds.sel(location=u'東京', type=u'平均')['temperature'] # <xray.DataArray 'temperature' (date: 6)> # array([ 28.8, 29.4, 28.9, 27. , 28.1, 28.6]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # type <U2 u'\u5e73\u5747' # location object u'\u6771\u4eac' # 東京 の 湿度 を選択 ds.sel(location=u'東京')['humidity'] # <xray.DataArray 'humidity' (date: 6)> # array([ 75., 65., 61., 80., 83., 80.]) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u6771\u4eac' # 対応する DataArray がない場合は NaN となる ds.sel(location=u'誰かの家') # <xray.Dataset> # Dimensions: (date: 6, type: 3) # Coordinates: # * date (date) datetime64[ns] 2015-07-20 2015-07-21 2015-07-22 ... # location object u'\u8ab0\u304b\u306e\u5bb6' # * type (type) <U2 u'\u6700\u9ad8' u'\u6700\u4f4e' u'\u5e73\u5747' # Data variables: # precipitation (date) float64 nan nan nan nan nan nan # temperature (date, type) float64 28.37 26.45 27.48 33.79 26.8 30.13 ... # humidity (date) float64 nan nan nan nan nan nan
そのほかの操作も DataArray と同じようにできる。
# location ごとに期間中の最大値を計算 ds.groupby('location').max() # <xray.Dataset> # Dimensions: (location: 3) # Coordinates: # * location (location) <U3 u'\u516b\u738b\u5b50' u'\u5927\u5cf6' ... # Data variables: # precipitation (location) float64 30.0 1.0 7.0 # temperature (location) float64 36.0 30.2 34.9 # humidity (location) float64 nan 91.0 83.0
pandas のデータ形式への変換
DataArray, Dataset はそれぞれ pandas のデータに変換できる。元データが 3 次元以上の場合、変換後の pandas のデータは MultiIndex を持つことになる。
また、上の例では 次元が異なる DataArray から Dataset を作成した。このとき、存在しない次元 ( ここでは type ) のデータはすべて同じ値でパディングされる。
ds.to_dataframe()
パディングしたくない場合は、個々の DataArray ごとに DataFrame に変換すればよい。
ds['humidity'].to_dataframe()
まとめ
xray を使えば 多次元のラベル付きデータを多次元のまま、pandas に近い方法で扱うことができる。
簡単な集約/変換処理を PySpark & pandas の DataFrame で行う
こちらの続き。
準備
サンプルデータは iris 。今回は HDFS に csv を置き、そこから読み取って DataFrame を作成する。
# HDFS にディレクトリを作成しファイルを置く $ hadoop fs -mkdir /data/ $ hadoop fs -put iris.csv /data/ $ hadoop fs -ls / Found 1 items drwxr-xr-x - ec2-user supergroup 0 2015-04-28 20:01 /data # Spark のパスに移動 $ echo $SPARK_HOME /usr/local/spark $ cd $SPARK_HOME $ pwd /usr/local/spark $ bin/pyspark
補足 前回同様に pandas から直接 PySpark の DataFrame を作成した場合、groupBy 時に java.lang.OutOfMemoryError: Java heap space エラーが発生してシェルごと落ちる。
CSV ファイルの読み込み
pandas では前回同様 read_csv。
import numpy as np import pandas as pd # 表示する行数を設定 pd.options.display.max_rows=10 names = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth', 'Species'] # pandas pdf = pd.read_csv('~/iris.csv', header=None, names=names) pdf # 略
PySpark は標準では csv から直接 DataFrame を作成できないため、一度 Row のリストを作成して DataFrame に変換する。
from pyspark.sql import Row lines = sc.textFile("hdfs://127.0.0.1:9000/data/iris.csv") cells = lines.map(lambda l: l.split(",")) rows = cells.map(lambda x: Row(SepalLength=float(x[0]), SepalWidth=float(x[1]), PetalLength=float(x[2]), PetalWidth=float(x[3]), Species=x[4])) sdf = sqlContext.createDataFrame(rows) sdf.show() # 略
グルーピング/集約
ある列の値ごとに集計
pandas, PySpark で多少 文法は異なる。
列の値でグループ分けし、一列の合計を取得する場合:
# pandas pdf.groupby('Species')['SepalLength'].sum() # Species # setosa 250.3 # versicolor 296.8 # virginica 329.4 # Name: SepalLength, dtype: float64 # PySpark sdf.groupBy('Species').sum('SepalLength').show() # Species SUM(SepalLength) # virginica 329.3999999999999 # versicolor 296.8 # setosa 250.29999999999998
指定した複数列の合計を取得する場合:
# pandas pdf.groupby('Species')[['PetalWidth', 'PetalLength']].sum() # PetalWidth PetalLength # Species # setosa 12.2 73.2 # versicolor 66.3 213.0 # virginica 101.3 277.6 # PySpark sdf.groupBy('Species').sum('PetalWidth', 'PetalLength').show() # Species SUM(PetalWidth) SUM(PetalLength) # virginica 101.29999999999998 277.59999999999997 # versicolor 66.30000000000001 213.0 # setosa 12.199999999999996 73.2
全列の合計を取得する場合:
# pandas pdf.groupby('Species').sum() # SepalLength SepalWidth PetalLength PetalWidth # Species # setosa 250.3 170.9 73.2 12.2 # versicolor 296.8 138.5 213.0 66.3 # virginica 329.4 148.7 277.6 101.3 # PySpark sdf.groupBy('Species').sum().show() # Species SUM(PetalLength) SUM(PetalWidth) SUM(SepalLength) SUM(SepalWidth) # virginica 277.59999999999997 101.29999999999998 329.3999999999999 148.7 # versicolor 213.0 66.30000000000001 296.8 138.5 # setosa 73.2 12.199999999999996 250.29999999999998 170.90000000000003
補足 pandas では グループ化したデータも DataFrame と同じようにスライシングできたりする。
一方、PySpark の GroupedData は集約系のAPI しか持っていない。
# pandas pdf.groupby('Species')['PetalWidth'] # <pandas.core.groupby.SeriesGroupBy object at 0x7f62f4218d50> # PySpark (NG!) sdf.groupBy('Species')[['Species']] # TypeError: 'GroupedData' object has no attribute '__getitem__' sdf.groupBy('Species').select('PetalWidth') # AttributeError: 'GroupedData' object has no attribute 'select'
また、pandas では apply で自作の集約関数 (UDAF) を利用することができるが、PySpark 1.3.1 時点 では非対応らしい。PySpark の udf を利用して定義した自作関数を集約時に使うと以下のエラーになる。
# pandas pdf.groupby('Species')[['PetalWidth', 'PetalLength']].apply(np.sum) # PetalWidth PetalLength # Species # setosa 12.2 73.2 # versicolor 66.3 213.0 # virginica 101.3 277.6 # PySpark (NG!) import pyspark.sql.functions np_sum = pyspark.sql.functions.udf(np.sum, pyspark.sql.types.FloatType()) sdf.groupBy('Species').agg(np_sum(sdf.PetalWidth)) # py4j.protocol.Py4JJavaError: An error occurred while calling o334.agg. # : org.apache.spark.sql.AnalysisException: expression 'pythonUDF' is neither present in the group by, nor is it an aggregate function. Add to group by or wrap in first() if you don't care which value you get.;
行持ち / 列持ち変換
複数列持ちの値を行持ちに展開 (unpivot / melt)
pandas では pd.melt。 DataFrame.melt ではないので注意。
# pandas pmelted = pd.melt(pdf, id_vars=['Species'], var_name='variable', value_name='value') pmelted # Species variable value # 0 setosa SepalLength 5.1 # 1 setosa SepalLength 4.9 # 2 setosa SepalLength 4.7 # 3 setosa SepalLength 4.6 # 4 setosa SepalLength 5.0 # .. ... ... ... # 595 virginica PetalWidth 2.3 # 596 virginica PetalWidth 1.9 # 597 virginica PetalWidth 2.0 # 598 virginica PetalWidth 2.3 # 599 virginica PetalWidth 1.8 # # [600 rows x 3 columns]
同様の処理を PySpark でやるには、DataFrame.flatMap。1行の入力に対して複数行 (この例では4行) のデータを返すことができる。fratMap の返り値は RDD インスタンスになるため、必要なら再度 DataFrame 化する。
# PySpark def mapper(row): return [Row(Species=row[4], variable='PetalLength', value=row[0]), Row(Species=row[4], variable='PetalWidth', value=row[1]), Row(Species=row[4], variable='SepalLength', value=row[2]), Row(Species=row[4], variable='SepalWidth', value=row[3])] smelted = sqlContext.createDataFrame(sdf.flatMap(mapper)) smelted.show() # Species value variable # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.1 SepalLength # setosa 3.5 SepalWidth # ... .. ... # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.0 SepalLength # setosa 3.6 SepalWidth smelted.count() # 600L
複数行持ちの値を列持ちに変換 (pivot)
pandas では DataFrame.pivot。pivotするデータは列にする値 (以下では Species ) と行にする値 (以下では variable ) の組がユニークになっている必要がある。そのため、まず pivot 用データを作成 -> その後 pivot する。
# pandas # pivot 用データを作成 punpivot = pmelted.groupby(['Species', 'variable']).sum() punpivot = punpivot.reset_index() punpivot # Species variable value # 0 setosa PetalLength 73.2 # 1 setosa PetalWidth 12.2 # 2 setosa SepalLength 250.3 # 3 setosa SepalWidth 170.9 # 4 versicolor PetalLength 213.0 # .. ... ... ... # 7 versicolor SepalWidth 138.5 # 8 virginica PetalLength 277.6 # 9 virginica PetalWidth 101.3 # 10 virginica SepalLength 329.4 # 11 virginica SepalWidth 148.7 # # [12 rows x 3 columns] # pivot punpivot.pivot(index='variable', columns='Species', values='value') # Species setosa versicolor virginica # variable # PetalLength 73.2 213.0 277.6 # PetalWidth 12.2 66.3 101.3 # SepalLength 250.3 296.8 329.4 # SepalWidth 170.9 138.5 148.7
PySpark の DataFrame のままでは同じ処理はできないようなので、一度 RDD に変換してから、 groupBy -> map
# PySpark # pivot 用データを作成 sunpivot = smelted.groupBy('Species', 'variable').sum() sunpivot.show() # Species variable SUM(value) # versicolor SepalWidth 138.5 # versicolor SepalLength 296.8 # setosa PetalLength 73.2 # virginica PetalWidth 101.29999999999998 # versicolor PetalWidth 66.30000000000001 # setosa SepalWidth 170.90000000000003 # virginica PetalLength 277.59999999999997 # setosa SepalLength 250.29999999999998 # versicolor PetalLength 213.0 # setosa PetalWidth 12.199999999999996 # virginica SepalWidth 148.7 # virginica SepalLength 329.3999999999999 def reducer(obj): # variable : value の辞書を作成 result = {o[1]:o[2] for o in obj[1]} return Row(Species=obj[0], **result) # pivot spivot = sunpivot.rdd.groupBy(lambda x: x[0]).map(reducer) spivot.collect() # [Row(PetalLength=277.59999999999997, PetalWidth=101.29999999999998, SepalLength=329.3999999999999, SepalWidth=148.7, Species=u'virginica'), # Row(PetalLength=73.2, PetalWidth=12.199999999999996, SepalLength=250.29999999999998, SepalWidth=170.90000000000003, Species=u'setosa'), # Row(PetalLength=213.0, PetalWidth=66.30000000000001, SepalLength=296.8, SepalWidth=138.5, Species=u'versicolor')] sqlContext.createDataFrame(spivot).show() # PetalLength PetalWidth SepalLength SepalWidth Species # 277.59999999999997 101.29999999999998 329.3999999999999 148.7 virginica # 73.2 12.199999999999996 250.29999999999998 170.90000000000003 setosa # 213.0 66.30000000000001 296.8 138.5 versicolor
列の分割 / 結合
列の値を複数列に分割
ある列の値を適当に文字列処理して、新しい列を作成したい。pandas には 文字列処理用のアクセサがあるため、 assign と組み合わせて以下のように書ける。
# pandas psplitted = pmelted.assign(Parts=pmelted['variable'].str.slice(0, 5), Scale=pmelted['variable'].str.slice(5)) psplitted # Species variable value Parts Scale # 0 setosa SepalLength 5.1 Sepal Length # 1 setosa SepalLength 4.9 Sepal Length # 2 setosa SepalLength 4.7 Sepal Length # 3 setosa SepalLength 4.6 Sepal Length # 4 setosa SepalLength 5.0 Sepal Length # .. ... ... ... ... ... # 595 virginica PetalWidth 2.3 Petal Width # 596 virginica PetalWidth 1.9 Petal Width # 597 virginica PetalWidth 2.0 Petal Width # 598 virginica PetalWidth 2.3 Petal Width # 599 virginica PetalWidth 1.8 Petal Width # # [600 rows x 5 columns]
PySparkには上記のようなメソッドはないので map で処理する。
# PySpark def splitter(row): parts = row[2][:5] scale = row[2][5:] return Row(Species=row[0], value=row[1], Parts=parts, Scale=scale) ssplitted = sqlContext.createDataFrame(smelted.map(splitter)) ssplitted.show() # Parts Scale Species value # Petal Length setosa 1.4 # Petal Width setosa 0.2 # Sepal Length setosa 5.1 # Sepal Width setosa 3.5 # Petal Length setosa 1.4 # .. .. ... .. # Petal Length setosa 1.4 # Petal Width setosa 0.2 # Sepal Length setosa 5.0 # Sepal Width setosa 3.6
複数列の値を一列に結合
pandas では普通に文字列結合すればよい。
# pandas psplitted['variable2'] = psplitted['Parts'] + psplitted['Scale'] psplitted # Species variable value Parts Scale variable2 # 0 setosa SepalLength 5.1 Sepal Length SepalLength # 1 setosa SepalLength 4.9 Sepal Length SepalLength # 2 setosa SepalLength 4.7 Sepal Length SepalLength # 3 setosa SepalLength 4.6 Sepal Length SepalLength # 4 setosa SepalLength 5.0 Sepal Length SepalLength # .. ... ... ... ... ... ... # 595 virginica PetalWidth 2.3 Petal Width PetalWidth # 596 virginica PetalWidth 1.9 Petal Width PetalWidth # 597 virginica PetalWidth 2.0 Petal Width PetalWidth # 598 virginica PetalWidth 2.3 Petal Width PetalWidth # 599 virginica PetalWidth 1.8 Petal Width PetalWidth # # [600 rows x 6 columns]
PySpark では map。
# PySpark def unite(row): return Row(Species=row[2], value=row[3], variable=row[0] + row[1]) sqlContext.createDataFrame(splitted.map(unite)).show() # Species value variable # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.1 SepalLength # setosa 3.5 SepalWidth # .. .. .. # setosa 1.4 PetalLength # setosa 0.2 PetalWidth # setosa 5.0 SepalLength # setosa 3.6 SepalWidth
補足 withColumn の場合、オペレータは 数値の演算として扱われてしまうようなのでここでは使えない。
# PySpark (NG!) ssplitted.withColumn('variable', splitted.Parts + splitted.Scale).show() # Parts Scale Species value variable # Petal Length setosa 1.4 null # Petal Width setosa 0.2 null # .. .. .. .. ..
まとめ
PySpark と pandas のデータ集約/変形処理を整理した。
データ分析用途で利用したい場合、(ごく当たり前だが) データ量が少なく手元でさっといろいろ試したい場合は pandas、データ量が比較的多く 単純な処理を全体にかけたい場合は Spark がよい。
Spark は map 系の API が充実するとさらに使いやすくなりそうだ。が、小回りの効く文法/機能が充実していくことは考えにくいので 完全に Spark だけでデータ分析をする、、という状態には将来もならないのではないかと思う。小さいデータは pandas 使いましょう。

Learning Spark: Lightning-Fast Big Data Analysis
- 作者: Holden Karau,Andy Konwinski,Patrick Wendell,Matei Zaharia
- 出版社/メーカー: O'Reilly Media
- 発売日: 2015/01/28
- メディア: Kindle版
- この商品を含むブログを見る
簡単なデータ操作を PySpark & pandas の DataFrame で行う
Spark v1.3.0 で追加された DataFrame 、結構いいらしいという話は聞いていたのだが 自分で試すことなく時間が過ぎてしまっていた。ようやく PySpark を少し触れたので pandas との比較をまとめておきたい。内容に誤りや よりよい方法があればご指摘 下さい。
過去に基本的なデータ操作について 以下 ふたつの記事を書いたことがあるので、同じ処理のPySpark 版を加えたい。今回は ひとつめの "簡単なデータ操作〜" に相当する内容。
pandas 版
準備
環境は EC2 に作る。Spark のインストールについてはそのへんに情報あるので省略。サンプルデータは iris を csv でダウンロードしてホームディレクトリにおいた。以降の操作は PySpark のコンソールで行う。
# Spark のパスに移動 $ echo $SPARK_HOME /usr/local/spark $ cd $SPARK_HOME $ pwd /usr/local/spark # 既定だとログの出力が邪魔なので抑制 $ cp conf/log4j.properties.template conf/log4j.properties $ vi conf/log4j.properties # INFO をすべて WARN に変える $ bin/pyspark # Welcome to # ____ __ # / __/__ ___ _____/ /__ # _\ \/ _ \/ _ `/ __/ '_/ # /__ / .__/\_,_/_/ /_/\_\ version 1.3.1 # /_/ # # Using Python version 2.7.9 (default, Apr 1 2015 19:28:03) # SparkContext available as sc, HiveContext available as sqlContext.
ここでは HDFS は使わず、pandas の read_csv で pandas.DataFrame を作成する。列名に "." が入っていると PySpark でうまく動かないようだったため以下のカラム名にした。
import pandas as pd # 表示する行数を設定 pd.options.display.max_rows=10 names = ['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth', 'Species'] # pandas pdf = pd.read_csv('~/iris.csv', header=None, names=names) pdf # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns]
これを PySpark の DataFrame に変換する。pandas と PySpark の基本的な違いとして、
PySparkにはpandas.IndexにあたるものがないPySparkにはpandas.Seriesにあたるものがない ( 代わり? にRowがあるが、どの程度 柔軟な操作ができるかは未知 )
# PySpark sdf = sqlContext.createDataFrame(pdf) sdf # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double, Species: string] # データの中身を表示するには DataFrame.show() sdf.show() # SepalLength SepalWidth PetalLength PetalWidth Species # 5.1 3.5 1.4 0.2 setosa # 4.9 3.0 1.4 0.2 setosa # 4.7 3.2 1.3 0.2 setosa # 4.6 3.1 1.5 0.2 setosa # .. ... ... ... ... # 5.4 3.9 1.3 0.4 setosa # 5.1 3.5 1.4 0.3 setosa # 5.7 3.8 1.7 0.3 setosa # 5.1 3.8 1.5 0.3 setosa
以降、pdf が pandas、sdf が PySpark の DataFrame をあらわす。
type(pdf) # <class 'pandas.core.frame.DataFrame'> type(sdf) # <class 'pyspark.sql.dataframe.DataFrame'>
基本
まず基本的な操作を。先頭いくつかのデータを確認するには head。
PySpark での返り値は Row インスタンスのリストになる。
# pandas pdf.head(5) # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # PySpark sdf.head(5) # [Row(SepalLength=5.1, SepalWidth=3.5, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.9, SepalWidth=3.0, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.7, SepalWidth=3.2, PetalLength=1.3, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.6, SepalWidth=3.1, PetalLength=1.5, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=5.0, SepalWidth=3.6, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa')] # pandas type(pdf.head(5)) # <class 'pandas.core.frame.DataFrame'> # PySpark type(sdf.head(5)) # <type 'list'>
各カラムのデータ型の確認には dtypes。
# pandas pdf.dtypes # SepalLength float64 # SepalWidth float64 # PetalLength float64 # PetalWidth float64 # Species object # dtype: object # PySpark sdf.dtypes # [('SepalLength', 'double'), ('SepalWidth', 'double'), # ('PetalLength', 'double'), ('PetalWidth', 'double'), # ('Species', 'string')]
要約統計量の確認は describe。
# pandas pdf.describe() # SepalLength SepalWidth PetalLength PetalWidth # count 150.000000 150.000000 150.000000 150.000000 # mean 5.843333 3.054000 3.758667 1.198667 # std 0.828066 0.433594 1.764420 0.763161 # min 4.300000 2.000000 1.000000 0.100000 # 25% 5.100000 2.800000 1.600000 0.300000 # 50% 5.800000 3.000000 4.350000 1.300000 # 75% 6.400000 3.300000 5.100000 1.800000 # max 7.900000 4.400000 6.900000 2.500000 # PySpark sdf.describe().show() # summary SepalLength SepalWidth PetalLength PetalWidth # count 150 150 150 150 # mean 5.843333333333334 3.0540000000000003 3.758666666666666 1.1986666666666668 # stddev 0.8253012917851317 0.4321465800705415 1.7585291834055206 0.760612618588172 # min 4.3 2.0 1.0 0.1 # max 7.9 4.4 6.9 2.5
列操作
列名操作
参照と変更。PySpark ではプロパティへの代入は不可。
# pandas pdf.columns # Index([u'SepalLength', u'SepalWidth', u'PetalLength', u'PetalWidth', u'Species'], dtype='object') # PySpark sdf.columns # [u'SepalLength', u'SepalWidth', u'PetalLength', u'PetalWidth', u'Species'] # pandas pdf.columns = [1, 2, 3, 4, 5] pdf.columns # Int64Index([1, 2, 3, 4, 5], dtype='int64') # PySpark sdf.columns = [1, 2, 3, 4, 5] # AttributeError: can't set attribute
マッピングによって列名を変更するには、pandas では DataFrame.rename。
# pandas pdf.columns # Int64Index([1, 2, 3, 4, 5], dtype='int64') pdf = pdf.rename(columns={1:'SepalLength', 2:'SepalWidth', 3:'PetalLength', 4:'PetalWidth', 5:'Species'}) pdf # SepalLength SepalWidth PetalLength PetalWidth Species # 0 5.1 3.5 1.4 0.2 setosa # 1 4.9 3.0 1.4 0.2 setosa # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [150 rows x 5 columns]
PySpark では DataFrame.withColumnRenamed。
# PySpark sdf.withColumnRenamed('Species', 'xxx') # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double, xxx: string]
補足 非破壊的な処理のため、反映には代入が必要。ここでは列名変更したくないので代入しない。
列名による列選択
pandas.DataFrame の列選択では 当該の列のデータを含む Series が返ってくる。
# pandas pdf.Species # 0 setosa # 1 setosa # 2 setosa # 3 setosa # 4 setosa # ... # 145 virginica # 146 virginica # 147 virginica # 148 virginica # 149 virginica # Name: Species, dtype: object pdf['Species'] # 略
PySpark では、列の属性を表現する Column インスタンスが返ってくる。
# PySpark sdf.Species # Column<Species> sdf['Species'] # Column<Species>
pandas、PySpark いずれも、文字列ではなくリストを渡せば その列を DataFrame としてスライシングする。
# pandas pdf[['PetalWidth']] # PetalWidth # 0 0.2 # 1 0.2 # 2 0.2 # 3 0.2 # 4 0.2 # .. ... # 145 2.3 # 146 1.9 # 147 2.0 # 148 2.3 # 149 1.8 # # [150 rows x 1 columns] # PySpark sdf[['PetalWidth']] # DataFrame[PetalWidth: double] # pandas pdf[['PetalWidth', 'PetalLength']] # PetalWidth PetalLength # 0 0.2 1.4 # 1 0.2 1.4 # 2 0.2 1.3 # 3 0.2 1.5 # 4 0.2 1.4 # .. ... ... # 145 2.3 5.2 # 146 1.9 5.0 # 147 2.0 5.2 # 148 2.3 5.4 # 149 1.8 5.1 # # [150 rows x 2 columns] # PySpark sdf[['PetalWidth', 'PetalLength']] # DataFrame[PetalWidth: double, PetalLength: double]
PySpark では DataFrame.select でもよい。
# PySpark sdf.select(sdf['PetalWidth'], sdf['PetalLength']) # DataFrame[PetalWidth: double, PetalLength: double]
真偽値リストによる列選択
自分はあまり使わないのだが、R {dplyr} との比較という観点で。
indexer = [False, False, True, True, False] # pandas pdf.loc[:, indexer] # PetalLength PetalWidth # 0 1.4 0.2 # 1 1.4 0.2 # 2 1.3 0.2 # 3 1.5 0.2 # 4 1.4 0.2 # .. ... ... # 145 5.2 2.3 # 146 5.0 1.9 # 147 5.2 2.0 # 148 5.4 2.3 # 149 5.1 1.8 # # [150 rows x 2 columns] # PySpark sdf[[c for c, i in zip(sdf.columns, indexer) if i is True]] # DataFrame[PetalLength: double, PetalWidth: double]
列の属性による列選択
列の型が pandas の場合は float、PySpark の場合は double の列のみ取り出す。
# pandas pdf.loc[:, pdf.dtypes == float] # SepalLength SepalWidth PetalLength PetalWidth # 0 5.1 3.5 1.4 0.2 # 1 4.9 3.0 1.4 0.2 # 2 4.7 3.2 1.3 0.2 # 3 4.6 3.1 1.5 0.2 # 4 5.0 3.6 1.4 0.2 # .. ... ... ... ... # 145 6.7 3.0 5.2 2.3 # 146 6.3 2.5 5.0 1.9 # 147 6.5 3.0 5.2 2.0 # 148 6.2 3.4 5.4 2.3 # 149 5.9 3.0 5.1 1.8 # # [150 rows x 4 columns] # PySpark sdf[[c for c, type in sdf.dtypes if type == 'double']] # DataFrame[SepalLength: double, SepalWidth: double, PetalLength: double, # PetalWidth: double]
行操作
値の条件による行選択
# pandas pdf[pdf['Species'] == 'virginica'] # SepalLength SepalWidth PetalLength PetalWidth Species # 100 6.3 3.3 6.0 2.5 virginica # 101 5.8 2.7 5.1 1.9 virginica # 102 7.1 3.0 5.9 2.1 virginica # 103 6.3 2.9 5.6 1.8 virginica # 104 6.5 3.0 5.8 2.2 virginica # .. ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica # 146 6.3 2.5 5.0 1.9 virginica # 147 6.5 3.0 5.2 2.0 virginica # 148 6.2 3.4 5.4 2.3 virginica # 149 5.9 3.0 5.1 1.8 virginica # # [50 rows x 5 columns] # PySpark sdf[sdf['Species'] == 'virginica'].show() # SepalLength SepalWidth PetalLength PetalWidth Species # 6.3 3.3 6.0 2.5 virginica # 5.8 2.7 5.1 1.9 virginica # 7.1 3.0 5.9 2.1 virginica # 6.3 2.9 5.6 1.8 virginica # .. ... ... ... ... # 6.5 3.0 5.5 1.8 virginica # 7.7 3.8 6.7 2.2 virginica # 7.7 2.6 6.9 2.3 virginica # 6.0 2.2 5.0 1.5 virginica
PySpark では DataFrame.filter でもよい。
# PySpark sdf.filter(sdf['Species'] == 'virginica').show() # 略
行番号による行選択
PySpark では 冒頭の n 行を抽出したりはできるが、適当な行選択ではできない、、、と思う。PySpark でどうしてもやりたければ index にあたる列を追加 -> 列の値で選択。
4/27追記 DataFrame.collect で Row のリストに変換 -> 再度 DataFrame 化すればスキーマ変更せずにできる、、、が、マスタでデータを処理することになるのでよい方法ではなさそう。
# pandas pdf.loc[[2, 3, 4]] # SepalLength SepalWidth PetalLength PetalWidth Species # 2 4.7 3.2 1.3 0.2 setosa # 3 4.6 3.1 1.5 0.2 setosa # 4 5.0 3.6 1.4 0.2 setosa # PySpark [c for i, c in enumerate(sdf.collect()) if i in (2, 3 ,4)] # [Row(SepalLength=4.7, SepalWidth=3.2, PetalLength=1.3, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=4.6, SepalWidth=3.1, PetalLength=1.5, PetalWidth=0.2, Species=u'setosa'), # Row(SepalLength=5.0, SepalWidth=3.6, PetalLength=1.4, PetalWidth=0.2, Species=u'setosa')] sqlContext.createDataFrame([c for i, c in enumerate(sdf.collect()) if i in (2, 3 ,4)]).show() # SepalLength SepalWidth PetalLength PetalWidth Species # 4.7 3.2 1.3 0.2 setosa # 4.6 3.1 1.5 0.2 setosa # 5.0 3.6 1.4 0.2 setosa
ランダムサンプリング
pandas ではindex をサンプリングしてスライス。
import random # pandas pdf.loc[random.sample(pdf.index, 5)] # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 64 6.1 2.9 4.7 1.4 versicolor # 17 5.4 3.9 1.3 0.4 setosa # 14 4.3 3.0 1.1 0.1 setosa # 4 4.6 3.1 1.5 0.2 setosa # 146 6.7 3.0 5.2 2.3 virginica
PySpark では DataFrame.sample。この関数、fraction の確率で行ごとに選択しているようで、サンプリングするたびに抽出される行数が変わる ( 行数指定でランダムサンプリング、という処理はできなさそう )。
# PySpark sdf.sample(False, 5.0 / sdf.count()).show() # SepalLength SepalWidth PetalLength PetalWidth Species # 5.4 3.4 1.7 0.2 setosa # 5.7 2.8 4.5 1.3 versicolor # 6.2 2.8 4.8 1.8 virginica # 6.1 3.0 4.9 1.8 virginica
補足 今後、pandas にもサンプリングメソッド DataFrame.sample が実装予定 #9666。
列の追加
pandas では DataFrame に対して 直接 新しい列を追加できるが、PySpark ではできない。
# 破壊的な処理になるのでコピー pdf_temp = pdf.copy() # pandas pdf_temp['PetalMult'] = pdf_temp['PetalWidth'] * pdf_temp['PetalLength'] pdf_temp # SepalLength SepalWidth PetalLength PetalWidth Species PetalMult # 0 5.1 3.5 1.4 0.2 setosa 0.28 # 1 4.9 3.0 1.4 0.2 setosa 0.28 # 2 4.7 3.2 1.3 0.2 setosa 0.26 # 3 4.6 3.1 1.5 0.2 setosa 0.30 # 4 5.0 3.6 1.4 0.2 setosa 0.28 # .. ... ... ... ... ... ... # 145 6.7 3.0 5.2 2.3 virginica 11.96 # 146 6.3 2.5 5.0 1.9 virginica 9.50 # 147 6.5 3.0 5.2 2.0 virginica 10.40 # 148 6.2 3.4 5.4 2.3 virginica 12.42 # 149 5.9 3.0 5.1 1.8 virginica 9.18 # # [150 rows x 6 columns] # PySpark sdf['PetalMult'] = sdf['PetalWidth'] * sdf['PetalLength'] # TypeError: 'DataFrame' object does not support item assignment
PySpark での列追加は DataFrame.withColumn。
# PySpark sdf.withColumn('PetalMult', sdf.PetalWidth * sdf.PetalLength).show() # SepalLength SepalWidth PetalLength PetalWidth Species PetalMult # 5.1 3.5 1.4 0.2 setosa 0.27999999999999997 # 4.9 3.0 1.4 0.2 setosa 0.27999999999999997 # 4.7 3.2 1.3 0.2 setosa 0.26 # 4.6 3.1 1.5 0.2 setosa 0.30000000000000004 # .. .. ... ... ... ... # 5.4 3.9 1.3 0.4 setosa 0.52 # 5.1 3.5 1.4 0.3 setosa 0.42 # 5.7 3.8 1.7 0.3 setosa 0.51 # 5.1 3.8 1.5 0.3 setosa 0.44999999999999996
補足 pandas v0.16.0 以降では 類似のメソッド DataFrame.assign が追加。
# pandas pdf.assign(PetalMult=pdf['PetalWidth'] * pdf['PetalLength']) # 略
まとめ
PySpark と pandas のデータ操作を整理した。
PySpark、上のような基本的な処理は pandas と似たやり方で直感的に使える感じだ。
大規模処理は PySpark、細かい取り回しが必要なものは pandas でうまく併用できるとよさそう。
4/29追記 続きはこちら。

Learning Spark: Lightning-Fast Big Data Analysis
- 作者: Holden Karau,Andy Konwinski,Patrick Wendell,Matei Zaharia
- 出版社/メーカー: O'Reilly Media
- 発売日: 2015/01/28
- メディア: Kindle版
- この商品を含むブログを見る