Chainer + Dask で 並列 Deep Learning したい <1>
この記事は Chainer Advent Calendar 2015 17 日目の記事です。
はじめに
サイズが大きいデータを Deep Learning すると学習に時間がかかってつらい。時間がかかってつらいので並列処理して高速化したい。
並列化するのに良さそうなパッケージないかな? と探してみると、Dask という並列 / Out-Of-Core 計算パッケージを見つけた。これと Chainer を組み合わせると並列処理が簡単に書けそうな気がする。
最初は MNIST を並列化してみたが、データが小さすぎるせいか むしろ遅くなってしまった。もう少し大きいデータである CIFAR-10 を使い、より深いネットワーク構造でその効果を確かめたい。
最終的には以下二つの処理を並列化することを目指す。
- Data Augmentation
- DNN の学習
1. Data Augmentation
層の深い DNN をうまく学習させるため、学習データに対して クロッピング等の画像処理を行い学習データを増やす。予測も Augmentation した画像群に対して行いその平均をとる。具体的な処理は id:ultraist さんのエントリに詳しい。
これは Dask を使って CPU 側で並列処理したい。Dask についてはこちらを。
- Python Dask で 並列 DataFrame 処理 - StatsFragments
- Python Dask.Array で 並列 / Out-Of-Core 処理 - StatsFragments
補足 もっとも、学習データ/テストデータはそれぞれ前もって 1 度だけ Augmentation しておけばよいため、この並列化の重要度は低い。Daskは むしろ 2. DNN の学習の並列化をシンプルに書くために使う (予定)。
2. DNN の学習
Chainer を使って GPU 側で並列処理したい。Chainer のドキュメントにも記載されているが、学習を並列化する方法は大きく 2 種類ある。
| 2-1 | Model Parallel | ある DNN の処理ごとに異なる GPU を割り当てて並列化する。 |
| 2-2 | Data Parallel | ある DNN を複数のデータ/GPUで同時に学習させ、学習したパラメータをなんらかの方法で統合する。 |
2-1. Model Parallel は モデルごとに実装する必要がある。2-2. Data Parallel はある程度 汎用的に書けそうなので、今回は Data Parallel をやりたい。
2-2. Data Parallel での DNN の学習
Data Parallel にもいくつか方法がある。詳細は "確率的最適化 (機械学習プロフェッショナルシリーズ)" や PFI 岡野原さんの資料に記載されている。

- 作者: 鈴木大慈
- 出版社/メーカー: 講談社
- 発売日: 2015/08/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログを見る
Data Parallel について自分の理解を簡単に整理すると、
| 手法 | 概要 |
|---|---|
| Parameter Mixture (単純平均) | パラメータを各ノードで並列に最適化し、最後にパラメータの平均をとる。期待誤差が強凸 / 損失関数が十分滑らかなら上手くいくらしい。 |
| Distributed Gradient (同期型・ミニバッチ法) | 勾配の計算を各ノードで並列に行い、パラメータの更新は 1台のノードで行う。勾配計算のたびにパラメータの同期が発生する。Chainer のドキュメントに記載されているのはこの方式。 |
| Asynchronous Update (非同期分散) | 勾配の計算、パラメータ更新を各ノードで並列に行う。パラメータ更新時にロックしないため、古いパラメータを元に勾配計算 / 更新がされることがある。実装の一種である Hogwild! は スパースな問題を対象とし、パラメータ更新時に勾配が 0 でない部分だけを更新する。 |
| Iterative Parameter Mixture | パラメータを各ノードで並列に最適化する。特定の期間ごと (epochごと等) にパラメータの平均をとり、更新されたパラメータを元に各ノードで最適化を繰り返す。 |
目指す姿
トラブルや金銭的な課題 (EC2) がなければ、以下 5 ステップに分けてやってみるつもり。
| 1 | データの準備 & Distributed Gradient での DNN 学習並列化 (←今回) |
| 2 | Iterative Parameter Mixture での DNN 学習並列化 |
| 3 | Dask による Data Augmentation の並列化 |
| 4 | ステップ 2 + ステップ 3 の処理を統合 |
| 5 | blaze/distributed ( 旧 dask.distributed )によるノード間分散処理 |
利用環境
- EC2 g2.8xlarge (GPU 4, vCPU 32, メモリ 60 GiB)
- Amazon Linux AMI with NVIDIA GRID GPU Driver
- cuDNN v2 (March 17,2015)
データのダウンロード
CIFAR-10 は以下サイトから入手できる。スクリプトでダウンロードする。
import numpy as np np.__version__ # '1.10.2' import os import requests fname = 'cifar-10-python.tar.gz' datadir = 'data' os.mkdir(datadir) reader = requests.get('http://www.cs.toronto.edu/~kriz/{0}'.format(fname), stream=True) with open(os.path.join(datadir, fname), 'wb') as f: for chunk in reader.iter_content(chunk_size=1024): if chunk: f.write(chunk) f.flush()
ダウンロードした tar ファイルを解凍する。以下のスクリプトを実行すると、 data/cifar-10-batches-py ディレクトリの下に 訓練用、テスト用のデータを含む pickle ファイルができる (拡張子はない)。
import tarfile tf = tarfile.open(os.path.join(datadir, fname), 'r') tf.extractall(datadir) tarpath = tf.getnames()[0] tarpath # 'cifar-10-batches-py' files = os.listdir(os.path.join(datadir, tarpath)) datafiles = [os.path.join(datadir, tarpath, f) for f in files if f.startswith('data')] datafiles # ['data/cifar-10-batches-py/data_batch_5', # 'data/cifar-10-batches-py/data_batch_4', # 'data/cifar-10-batches-py/data_batch_1', # 'data/cifar-10-batches-py/data_batch_3', # 'data/cifar-10-batches-py/data_batch_2']
データの準備
データは Dask で読み込む。もっとも、今回は学習時には Dask ではなく np.ndarray を直接使うので普通に読み込んでもいい ( 次回以降は Dask が活躍する予定...)。
まずは pickle ファイルから 画像データ / ラベルデータを取得する関数を定義する。
import dask import dask.array as da dask.__version__ # '0.7.5' from six.moves import cPickle as pickle def load(fn): # https://www.cs.toronto.edu/~kriz/cifar.html with open(os.path.join(datadir, tarpath, fn), 'rb') as f: result = pickle.load(f) return result def load_data(fn): return load(fn)['data'] def load_labels(fn): # list から np.ndarray に変換 return np.array(load(fn)['labels'])
これらを使って、訓練用データを読み込む Computational Graph を定義する。詳細はこちらのドキュメントを。
dsk_data = {('data', i, 0): (load_data, 'data_batch_{0}'.format(i + 1)) for i in range(5)}
dsk_data
# {('data', 0, 0): (<function __main__.load_data>, 'data_batch_1'),
# ('data', 1, 0): (<function __main__.load_data>, 'data_batch_2'),
# ('data', 2, 0): (<function __main__.load_data>, 'data_batch_3'),
# ('data', 3, 0): (<function __main__.load_data>, 'data_batch_4'),
# ('data', 4, 0): (<function __main__.load_data>, 'data_batch_5')}
data = da.Array(dsk_data, 'data', chunks=(10000, 3 * 32 * 32),
dtype=np.float32, shape=(50000, 3 * 32 * 32))
data
# dask.array<data, shape=(50000, 3072), dtype=float32, chunksize=(10000, 3072)>
data.compute()
# array([[ 59, 43, 50, ..., 140, 84, 72],
# [154, 126, 105, ..., 139, 142, 144],
# [255, 253, 253, ..., 83, 83, 84],
# ...,
# [ 35, 40, 42, ..., 77, 66, 50],
# [189, 186, 185, ..., 169, 171, 171],
# [229, 236, 234, ..., 173, 162, 161]], dtype=uint8)
ラベルについても同様。
dsk_labels = {('labels', i): (load_labels, 'data_batch_{0}'.format(i + 1)) for i in range(5)}
labels = da.Array(dsk_labels, 'labels', chunks=(10000, ), shape=(50000, ))
labels
# dask.array<labels, shape=(50000,), dtype=None, chunksize=(10000,)>
labels.compute()
# array([6, 9, 9, ..., 9, 1, 1])
テスト用データは np.ndarray として読み込んだ。
test_data = load_data('test_batch') test_labels = np.array(load_labels('test_batch'))
データの確認
データの中身を確かめるため各画像を描画してみたい。各レコード中には (チャネル, 縦, 横) という次元に対応する画素が順に 1 次元に並んでいる。これを matplotlib で描画するため (縦, 横, チャネル) の 3 次元となるように reshape → transpose する。
nrows = 3 ncols = 10 images = data[0:nrows*ncols, :].compute() images = images.reshape(nrows*ncols, 3, 32, 32).transpose(0, 2, 3, 1) images[0].shape # (32, 32, 3) import matplotlib.pyplot as plt fig, axes = plt.subplots(nrows, ncols, figsize=(10, 4)) for im, ax in zip(images, axes.flatten()): ax.imshow(im) ax.axis('off')

DNN の学習
Single GPU での学習
比較のため、まずは並列化せずに一つの GPU だけで学習 / テストがしたい。Chainer をインポートし、 CUDA, cuDNN が利用できることを確かめる。
import chainer chainer.__version__ # '1.5.1' import chainer.cuda as cuda cuda.available # True cuda.cudnn_enabled # True
学習するモデルとしては、@mitmul さんが GitHub 上で公開されている CIFAR-10 用のモデルを使わせていただく。
今回は Data Augmentation をしておらずデータ数が少ないため、比較的単純なモデルである models/Cifar10.py を利用した。API は後の処理にあわせて少し変更している。
from chainer import optimizers from chainer import Variable import chainer.functions as F import chainer.links as L class Cifar10(chainer.Chain): def __init__(self): super(Cifar10, self).__init__( conv1=L.Convolution2D(3, 32, 5, stride=1, pad=2), conv2=L.Convolution2D(32, 32, 5, stride=1, pad=2), conv3=L.Convolution2D(32, 64, 5, stride=1, pad=2), fc4=F.Linear(1344, 4096), fc5=F.Linear(4096, 10), ) def _internal_call(self, x, t, train=True): h = F.max_pooling_2d(F.relu(self.conv1(x)), 3, stride=2) h = F.max_pooling_2d(F.relu(self.conv2(h)), 3, stride=2) h = F.relu(self.conv3(h)) h = F.spatial_pyramid_pooling_2d(h, 3, F.MaxPooling2D) h = F.dropout(F.relu(self.fc4(h)), ratio=0.5, train=train) h = self.fc5(h) return h def __call__(self, x, t): h = self._internal_call(x, t, train=True) self.loss = F.softmax_cross_entropy(h, t) self.accuracy = F.accuracy(h, t) return self.loss def predict(self, x, t): """ データに対する予測値 (ラベル) を返す """ h = self._internal_call(x, t, train=False) self.pred = F.softmax(h) return self.pred.data.argmax(axis=1) optimizer = optimizers.Adam(alpha=0.001) model = Cifar10() xp = cuda.cupy model.to_gpu() optimizer.setup(model) optimizer.target # <__main__.Cifar10 at 0x7f7e9c586150>
ここで 訓練データ/テストデータを np.ndarray に変換する。
def data_transformer(x): # 各レコード 1 次元のデータを 3次元 (3, 32, 32) に変換する return x.astype(np.float32).reshape(len(x), 3, 32, 32) def labels_transformer(y): return y.astype(np.int32) tr_data = data_transformer(data.compute()) tr_labels = labels_transformer(labels.compute()) te_data = data_transformer(test_data) te_labels = labels_transformer(test_labels)
学習 / テストを行う。ログの出力など実行に不要な箇所は省略した。
def run_epoch(optimizer, model, data, labels, train=True, batch_size=100): sum_accuracy = 0 sum_loss = 0 total = len(labels) if train: indexer = np.random.permutation(total) data = data[indexer] labels = labels[indexer] for index in range(0, total, batch_size): x = data[index:index+batch_size] t = labels[index:index+batch_size] volatile = 'off' if train else 'on' x = Variable(xp.asarray(x), volatile=volatile) t = Variable(xp.asarray(t), volatile=volatile) if train: optimizer.update(model, x, t) sum_loss += float(model.loss.data) * len(t) sum_accuracy += float(model.accuracy.data) * len(t) else: predicted = model.predict(x, t) acc = float((predicted == t.data).sum()) sum_loss = np.nan sum_accuracy += acc return sum_accuracy, sum_loss for epoch in range(0, 20): print('******************** Epoch {:02d} (Train) ********************'.format(epoch + 1)) acc, loss = run_epoch(optimizer, model, tr_data, tr_labels, train=True) print('******************** Epoch {:02d} (Test) *********************'.format(epoch + 1)) acc, loss = run_epoch(optimizer, model, te_data, te_labels, train=False)
出力されたログを上に添付した。最左列は学習開始からの時間 (秒) である。20 Epoch 経過時点で 355 秒、テストデータに対して 60 % 程度の精度だった。
# ******************** Epoch 01 (Train) ******************** # 4.091: 10000/50000 loss=10.164 acc=0.123 # 7.329: 20000/50000 loss=6.162 acc=0.150 # 10.570: 30000/50000 loss=4.788 acc=0.177 # 13.812: 40000/50000 loss=4.064 acc=0.205 # 17.056: 50000/50000 loss=3.612 acc=0.231 # ******************** Epoch 01 (Test) ********************* # 18.140: 10000/10000 loss=nan acc=0.380 # # ... ... ... ... # # ******************** Epoch 20 (Train) ******************** # 340.570: 10000/50000 loss=0.993 acc=0.650 # 343.832: 20000/50000 loss=0.993 acc=0.650 # 347.095: 30000/50000 loss=1.001 acc=0.650 # 350.350: 40000/50000 loss=1.005 acc=0.650 # 353.611: 50000/50000 loss=1.014 acc=0.649 # ******************** Epoch 20 (Test) ********************* # 354.691: 10000/10000 loss=nan acc=0.621
Multi GPU での学習 (Distributed Gradient)
上に記載のとおり、今回は Distributed Gradient (同期型・ミニバッチ法) を利用した並列学習を行いたい。勾配計算は各 GPU で行い、パラメータの更新は 1 つの GPU で行う。
実装は Chainer のドキュメントに記載されている内容をなぞればよい。利用するメソッドの概要を整理する。
| メソッド | 概要 |
|---|---|
Link.zerograds() |
Link に含まれる各 Variable が持つ勾配 ._grad を 0 で埋める。 |
Link.__call__() |
最適化する Variable を返す。Link を継承したクラスで定義する。 |
Variable.backward() |
Variable から誤差逆伝播を行う |
Link.addgrads(link) |
引数の Link に含まれる Variable の勾配を自身の対応する Variable に加算する。 |
Optimizer.update() |
(引数なしで呼ばれた場合) 計算済みの勾配をもとに、モデルの Variable を更新する。実装は GradientMethod.update() 中にある。 |
Link.copyparams() |
引数の Link に含まれる Variable の値を自身の対応する Variable にコピーする。 |
とはいえ、ドキュメントの書き方だと GPU 数が増えると少し手間だ。簡単な wrapper を作り、 Distributed Gradient を以下のように定型的に書けるようにしたい。
with model as m: # 各 GPU 上のモデルで .zerograds() を実行 m(x, t) # 各 GPU 上のモデルで .__call__() を実行 # 各 GPU 上のモデルで .backward() を実行し、 # 計算された勾配を .addgrads() でマスターに加算 optimizer.update() model.syncparams() # マスターのパラメータを .copyparams() で各 GPU 上のモデルにコピー
そのための wrapper として GPUDistributedGradient クラスを定義する。
class GPUDistributedGradient(chainer.Chain): def __init__(self, chain, ngpus): if isinstance(ngpus, int): ngpus = list(range(ngpus)) self.ngpus = ngpus self._chains = [chain.copy().to_gpu(i) for i in ngpus] @property def master(self): """ 最初の GPU にあるモデルをマスターとする """ return self._chains[0] def __call__(self, *args): """ 引数毎に 'Variable のリスト' を渡す""" self._losses = [chain(*arg) for chain, arg in zip(self._chains, zip(*args))] return self._losses[0] def predict(self, x, t): """ 予測値はマスターで計算し、(まだ) 並列化しない """ return self.master.predict(x, t) def zerograds(self): for chain in self._chains: chain.zerograds() def backward(self): for loss in self._losses: loss.backward() def syncgrads(self): """ 各 GPU からの勾配をマスターに加算する """ for chain in self._chains[1:]: self.master.addgrads(chain) def __enter__(self): self.zerograds() return self def __exit__(self, exception_type, exception_value, traceback): self.backward() self.syncgrads() return True def syncparams(self): """ マスターのパラメータを他 GPU のモデルにコピーする """ for chain in self._chains[1:]: chain.copyparams(self.master)
上で作成したクラスを利用して、並列化したいモデルを wrap する。optimizer へは マスターとなるモデル model.master を渡す。
NGPU = 4 optimizer = optimizers.Adam(alpha=0.001) model = Cifar10() model = GPUDistributedGradient(model, NGPU) optimizer.setup(model.master) optimizer.target # <__main__.Cifar10 at 0x7f7e9c6ede10>
run_epoch を書き換えて実行してみる。run_epoch の呼び出しは先ほどと同じため省略する。
def run_epoch(optimizer, model, data, labels, train=True, batch_size=100): sum_accuracy = 0 sum_loss = 0 total = len(labels) if train: indexer = np.random.permutation(total) data = data[indexer] labels = labels[indexer] batch_size = batch_size * NGPU for index in range(0, total, batch_size): x = data[index:index+batch_size] t = labels[index:index+batch_size] if train: # train 時には 利用する GPU と同じ長さの Variable のリストを渡す x = [Variable(cuda.to_gpu(v, i)) for i, v in enumerate(np.array_split(x, NGPU))] t = [Variable(cuda.to_gpu(v, i)) for i, v in enumerate(np.array_split(t, NGPU))] else: x = Variable(xp.asarray(x), volatile='on') t = Variable(xp.asarray(t), volatile='on') if train: with model as m: m(x, t) optimizer.update() model.syncparams() n = sum([len(_t) for _t in t]) # マスターで計算された loss / acc をレコード数倍して近似 sum_loss += float(model.master.loss.data) * n sum_accuracy += float(model.master.accuracy.data) * n else: predicted = model.predict(x, t) acc = float((predicted == t.data).sum()) sum_loss = np.nan sum_accuracy += acc return sum_accuracy, sum_loss
Single GPU の場合と同じくログを添付する。20 Epoch までの処理時間は、並列化なしでの 355 秒に対し 並列化した場合は 277 秒と 20% 程度 速くなっている。また、テストデータに対する精度も並列化なしの場合と同程度で問題はなさそうだ。
# ******************** Epoch 01 (Train) ******************** # 2.870: 10000/50000 loss=1.438 acc=0.479 # 5.439: 20000/50000 loss=1.431 acc=0.480 # 8.008: 30000/50000 loss=1.417 acc=0.488 # 10.595: 40000/50000 loss=1.416 acc=0.491 # 13.155: 50000/50000 loss=1.405 acc=0.492 # ******************** Epoch 01 (Test) ********************* # 14.243: 10000/10000 loss=nan acc=0.517 # # ... ... ... ... # # ******************** Epoch 20 (Train) ******************** # 265.769: 10000/50000 loss=0.498 acc=0.824 # 268.249: 20000/50000 loss=0.509 acc=0.821 # 270.738: 30000/50000 loss=0.518 acc=0.818 # 273.230: 40000/50000 loss=0.524 acc=0.814 # 275.717: 50000/50000 loss=0.528 acc=0.813 # ******************** Epoch 20 (Test) ********************* # 276.785: 10000/10000 loss=nan acc=0.634
処理中に watch nvidia-smi すると 全 GPU が利用されていることが確かめられる。眺めていると GPU 1 〜 3 の利用率はしばしば 0% になっている (パラメータ更新のため)。
$ watch nvidia-smi
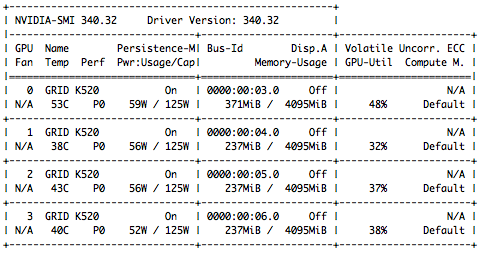
GPU の利用率が少ないのは モデルが単純だった / バッチサイズが小さかったせいだと思う。このあたりを適切に設定すればパフォーマンス向上の余地がありそうだ。
結果のプロット
縦軸をテストデータにおける精度、横軸を Epoch の経過 ( 100 Ticks で 1 Epoch ) としてグラフを描いた。
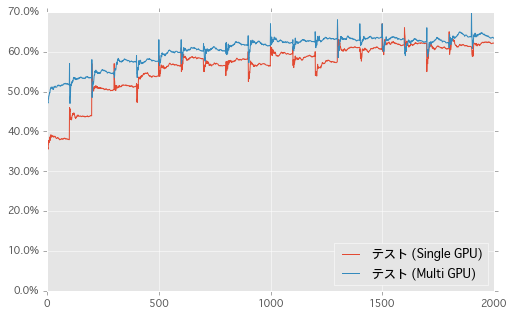
同じく、横軸に経過時間 (秒) として学習の過程をプロットした。訓練データの情報も追加した。形が異なるのは Multi-GPU で訓練データへの精度を正確に計算していないためかと思う。こうしてみると、テスト部分は並列化しなくてもあまり影響なさそうだ。
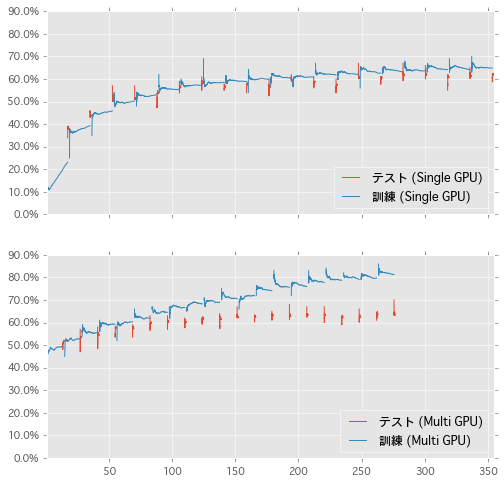
まとめ
Chainer のドキュメントの手順に従い、DNN の学習を 4 GPU / Distributed Gradient で並列化した。次は Dask の処理も使って Iterative Parameter Mixture をやりたい。
| 1 | データの準備 & Distributed Gradient での DNN 学習並列化 (←済み) |
| 2 | Iterative Parameter Mixture での DNN 学習並列化 (←次回) |
| 3 | Dask による Data Augmentation の並列化 |
| 4 | ステップ 2 + ステップ 3 の処理を統合 |
| 5 | blaze/distributed ( 旧 dask.distributed )によるノード間分散処理 |
利用したスクリプトは以下のリポジトリに置いた。Jupyter Notebook なので GitHub 上でレンダリングして確認することができる。
何かおかしいことやっていたらご指摘ください。

- 作者: 岡谷貴之
- 出版社/メーカー: 講談社
- 発売日: 2015/04/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (6件) を見る

- 作者: 鈴木大慈
- 出版社/メーカー: 講談社
- 発売日: 2015/08/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログを見る
Python Dask.Array で 並列 / Out-Of-Core 処理
この記事は Python Advent Calendar 2015 13 日目の記事です。
Python で手軽に並列 / Out-Of-Core 処理を行うためのパッケージである Dask について書きたい。Dask を使うと以下のようなメリットが得られる。
- 環境構築 / インストールが
pipで簡単にできる - 手軽に並列処理ができる
- Out-Of-Core (メモリに乗らないデータ) 処理ができる
補足 Dask は手持ちの PC の シングルコア / 物理メモリでは処理が少しきついかな、といった場合に利用するパッケージのため、より大規模 / 高速 / 安定した処理を行いたい場合には Hadoop や Spark を使ったほうがよい。
Dask は以下 3 つのサブパッケージを持つ。
| サブモジュール | ベースパッケージ |
|---|---|
dask.array |
NumPy |
dask.bag |
PyToolz (list, set, dict に対する処理) |
dask.dataframe |
pandas |
うち dask.dataframe については以前にエントリを書いた。
今回は NumPy API のサブセットをもつ dask.array について。dask.array でも基本的な考え方 / 挙動は dask.dataFrame と同じなので まず上のエントリを読んでください。
インストール
pip で。
# pip install dask
基本的な操作
import numpy as np np.__version__ # '1.10.1' import dask dask.__version__ # '0.7.5' import dask.array as da
dask.DataFrame はいくつかの行を chunk としてまとめて並列処理を行っていたが、dask.Array は各 axis それぞれを chunk として分割することができる。以下では 4 行 4 列 の ndarray を 2 行 2 列 計 4 つの chunk に分割する。
arr = np.array([[1, 2, 3, 4], [5, 6, 7, 8], [9, 10, 11, 12], [13, 14, 15, 16]]) arr # array([[ 1, 2, 3, 4], # [ 5, 6, 7, 8], # [ 9, 10, 11, 12], # [13, 14, 15, 16]]) darr = da.from_array(arr, chunks=2) darr # dask.array<from-ar..., shape=(4, 4), dtype=int64, chunksize=(2, 2)>
dask.Dataframe と同じく、dask.Array のメソッドを呼び出すことで 内部の Computational Graph を更新していく。評価 (計算の実行) を行うには .compute()。また、.visualize() で Computational Graph を描画することができる。
各行の合計を取る処理をみると、まずそれぞれの chunk ごとに 行の合計を計算 → その後 隣り合う chunk 同士の行の合計を取ることで最終的な出力を得ている。
darr.sum(axis=1) # dask.array<p_reduc..., shape=(4,), dtype=int64, chunksize=(2,)> darr.sum(axis=1).compute() # array([10, 26, 42, 58]) darr.sum(axis=1).visualize()

転置のように chunk の位置の入れ替えを伴う処理もできる。Computational Graph 中の四角形 (xxx, 0, 1) は (0, 1) 番目の chunk であることを表す。
darr.T.compute() # array([[ 1, 5, 9, 13], # [ 2, 6, 10, 14], # [ 3, 7, 11, 15], # [ 4, 8, 12, 16]]) darr.T.visualize()

dask.Array 同士の演算もできる。式中では darr.min(axis=1) が重複しているが、これらは Computational Graph 中でマージされ、評価時には 1 度だけ計算される。
((darr - darr.min(axis=1)) / (darr.max(axis=1) - darr.min(axis=1))).compute() # array([[ 0, -1, -2, -3], # [ 1, 0, -1, -2], # [ 2, 1, 0, -1], # [ 4, 3, 2, 1]]) ((darr - darr.min(axis=1)) / (darr.max(axis=1) - darr.min(axis=1))).visualize()

並列処理のメリット
各 chunk に対する処理は 自動的に並列化して実行されるため、ある程度データが大きい場合 / やりたい処理が並列で実行できる場合には速度メリットがある。以下では 長さ 1 億 の ndarray の合計を取る処理で比較した ( EC2 c4.2xlarge を利用)。
# NumPy arr = np.arange(100000000) %timeit arr.sum() # 10 loops, best of 3: 76.5 ms per loop # Dask darr = da.from_array(arr, chunks=10000000) %timeit darr.sum().compute() # 10 loops, best of 3: 25.3 ms per loop
補足 NumPy は主要な処理で GIL を解放しているため、Dask での並列処理は threading によって行われる。処理方法として multiprocessing を指定することもできる。
Out-Of-Core 処理のメリット
Dask では Computational Graph 中の各ノードを順に計算していくため、処理する全データが同時にメモリに乗っている必要がない。そのため、PC の物理メモリを超える大きさのデータも扱うことができる。以下のドキュメントでは複数の pkl ファイルを chunk として読み込む方法が記載されている。
関連パッケージ
以下のパッケージでは バックグラウンド処理を dask.array を利用して行うことができる。
Xray: 多次元ラベルデータをpandasのように処理できるパッケージ
まとめ
簡単に並列 / Out-Of-Core 処理を行うためのパッケージである Dask のサブモジュールのうち NumPy 互換の dask.array の使い方を記載した。

- 作者: Micha Gorelick,Ian Ozsvald,相川愛三
- 出版社/メーカー: オライリージャパン
- 発売日: 2015/11/20
- メディア: 大型本
- この商品を含むブログ (3件) を見る
Rust で k-means クラスタリング
この記事は Rust Advent Calendar 2015 7 日目の記事です。
簡単な統計/機械学習のアルゴリズムを実装しつつ Rust を学びたい。こちらの続き。
環境:
- Rust(Nightly) rustc 1.6.0-nightly (d49e36552 2015-12-05)
- rust-csv 0.14.3 : CSV の読み込みに利用
- nalgebra 0.3.2 : 行列/ベクトルの処理に利用
- rand 0.3.12 : 乱数生成に利用
- gnuplot 0.0.19 : プロットに利用
k-means クラスタリングとは
非階層型クラスタリングの一種。下のアニメーションがわかりやすい。
Rust でプロット
これまでは 結果を数値で表示するだけだったが、味気ないのでプロットを出力したい。検索したところ gnuplot の Rust 用 wrapper があった。
extern crate gnuplot; use gnuplot::{Figure, Color}; fn main() { let mut fg = Figure::new(); fg.axes2d() .lines(&[1, 2, 3], &[4, 5, 6], &[Color("blue")]) .lines(&[1, 2, 3], &[7, 6, 5], &[Color("red")]); fg.set_terminal("png", "test.png"); fg.show(); }

描画するオブジェクト (上の例では .lines) は fg.axes2d() からチェインさせて呼び出すことが推奨されているようだ。 let ax = fg.axes2d() など変数に代入してしまうと fg の所有権が move したままとなり、 fg.show() で描画できない状態になってしまう。
が、それではデータを逐次描画していくことができないため、以下の例ではすこし変わった書き方をしている。
Rust で k-means
前回まで作ったものを Crate にして、必要なクラス、関数を再利用している。
extern crate csv; extern crate gnuplot; extern crate nalgebra; extern crate num; extern crate rand; extern crate brasswheels; use gnuplot::{Figure, Color}; use nalgebra::{DVec, DMat, RowSlice, Iterable}; use rand::sample; use std::collections::HashMap; use std::f64; use std::ops::Index; use brasswheels::io::read_csv_f64; // CSV から f64 型のみを読みこむ use brasswheels::pca::PCA; // 主成分分析 use brasswheels::mathfunc::euc_dist; // 2 つのベクトルのユークリッド距離を求める fn main() { // http://aima.cs.berkeley.edu/data/iris.csv let path = "./data/iris.csv"; let mut reader = csv::Reader::from_file(path).unwrap().has_headers(false); let dx = read_csv_f64(&mut reader); // k-means (クラスタ数, 最大のイテレーション数) let mut kmeans = KMeans::new(3, 300); kmeans.fit(&dx); println!("各クラスタの中心点"); for (_, cluster) in &kmeans.centroids { println!("{:?}", cluster.centroid.at); } let predicted = kmeans.predict(&dx); println!("クラスタリング結果\n{:?}", &predicted.at); // 2 次元に描画するため主成分分析を行う let mut pca = PCA::new(4, true); pca.fit(&dx); let transformed = pca.transform(&dx); // プロット // http://siegelord.github.io/RustGnuplot/doc/gnuplot/struct.Axes2D.html let mut fg = Figure::new(); let colors = ["blue", "red", "green"]; // fg.axes2d() によって fg が move するため、 // 同一ブロック中で呼び出すと fg.show() が使えなくなる (0..kmeans.nclusters).fold(fg.axes2d(), |ax, c| { let mut xvals: Vec<f64> = vec![]; let mut yvals: Vec<f64> = vec![]; for (rownum, &predc) in predicted.iter().enumerate() { if predc == c { xvals.push(*transformed.index((rownum, 0))); yvals.push(*transformed.index((rownum, 1))); } } return ax.points(&xvals, &yvals, &[Color(colors[c])]); }); fg.set_terminal("png", "kmeans.png"); fg.show(); } pub struct KMeans { pub nclusters: usize, // クラスタ数 max_iter: usize, // イテレーション回数 pub centroids: HashMap<usize, Cluster>, } impl KMeans { pub fn new(nclusters: usize, max_iter: usize) -> KMeans { KMeans { nclusters: nclusters, max_iter: max_iter, centroids: HashMap::new(), } } pub fn fit(&mut self, data: &DMat<f64>) { let mut rng = rand::thread_rng(); // データからクラスタの初期値をサンプリング (非復元抽出) let inits: Vec<usize> = sample(&mut rng, 0..data.nrows(), self.nclusters); for (i, rownum) in inits.into_iter().enumerate() { let mut c = Cluster::new(data.ncols()); let row = data.row_slice(rownum, 0, data.ncols()); c.add_element(row); self.centroids.insert(i, c); } let mut cindexer = self.predict(data); // 最大 max_iter 回繰り返し for _ in 0..self.max_iter { // 中心点の更新 self.update_centroids(&data, &cindexer); // 各レコードを クラスタの中心点にもっとも近いものに分類 let cindexer_new = self.predict(data); // Eq での比較結果は element-wise ではなく bool になる if cindexer_new == cindexer { // 変化がなくなったら終了 break; } else { cindexer = cindexer_new; } } } /// 各レコードが所属するクラスタのベクトルを返す pub fn predict(&self, data: &DMat<f64>) -> DVec<usize> { return DVec::from_fn(data.nrows(), |x| self.get_nearest(&data.row_slice(x, 0, data.ncols()))); } /// レコードにもっとも近い中心点をもつクラスタのラベルを返す fn get_nearest(&self, values: &DVec<f64>) -> usize { let mut tmp_i = 0; let mut current_dist = f64::MAX; for (cnum, cluster) in &self.centroids { let d = euc_dist(values, &cluster.centroid); if d < current_dist { current_dist = d; tmp_i = *cnum; } } return tmp_i; } /// クラスタの中心点を更新する fn update_centroids(&mut self, data: &DMat<f64>, cindexer: &DVec<usize>) { self.centroids.clear(); for i in 0..self.nclusters { let c = Cluster::new(data.ncols()); self.centroids.insert(i, c); } for (rownum, cnum) in cindexer.iter().enumerate() { let row = data.row_slice(rownum, 0, data.ncols()); let mut c = self.centroids.remove(&cnum).unwrap(); c.add_element(row); self.centroids.insert(*cnum, c); } for cnum in 0..self.nclusters { let mut c = self.centroids.remove(&cnum).unwrap(); c.finalize(); self.centroids.insert(cnum, c); } } } pub struct Cluster { pub centroid: DVec<f64>, n: f64 } impl Cluster { fn new(ncols: usize) -> Cluster { Cluster { centroid: DVec::from_elem(ncols, 0.), n: 0. } } /// 中心点を更新 (レコードを合計に追加する) fn add_element(&mut self, values: DVec<f64>) { self.centroid = self.centroid.clone() + values; self.n = self.n + 1.; } /// 中心点を更新 (レコード数で割り、中心点を求める) fn finalize(&mut self) { if self.n != 0. { self.centroid = self.centroid.clone() / self.n; self.n = 0.; } } }
結果の出力。
各クラスタの中心点 # [5.005999999999999, 3.4180000000000006, 1.464, 0.2439999999999999] # [5.88360655737705, 2.740983606557377, 4.388524590163935, 1.4344262295081966] # [6.853846153846153, 3.0769230769230766, 5.715384615384615, 2.053846153846153] クラスタリング結果 # [2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, # 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, # 0, 1, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, # 1, 1, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, # 0, 1, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 1, 0, 0, 0, 0, 1, 0, 1, 0, 1, 0, # 0, 1, 1, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 1]
クラスタごとに色分けしてプロットすると以下のようになる。うまくクラスタリングできているようだ。
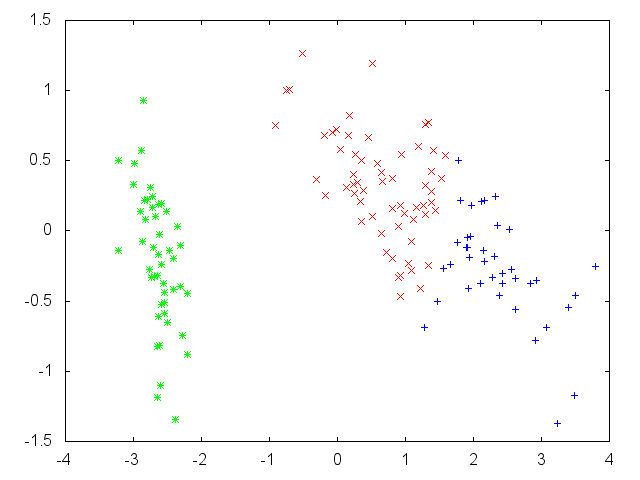
R の結果。初期値の選ばれ方次第で中心点の順序が変わる / 差異がより大きくなる場合もある。
biris = read.csv("iris.csv", header=FALSE) kmeans(biris[-5], 3)$centers # V1 V2 V3 V4 # 1 5.006000 3.418000 1.464000 0.244000 # 2 5.901613 2.748387 4.393548 1.433871 # 3 6.850000 3.073684 5.742105 2.071053
R {ggplot2} で 少し複雑なサブプロットが描きたい
この記事は R Advent Calendar 2015 4 日目の記事です。
{ggplot2} でのサブプロット
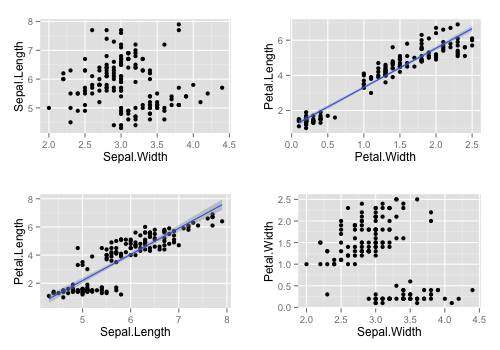
{ggplot2} でサブプロットを描画したいことがある。同じ種類のプロットを水準別に描画するなど、単純なものであれば facet で描ける。例えば 適当な散布図を Species 別に描きたい場合、
library(dplyr) library(ggplot2) ggplot(iris, aes(Sepal.Width, Sepal.Length)) + geom_point() + facet_wrap(~ Species)

facet では異なる種類のプロットをサブプロットとすることはできない。例えば散布図の縦軸/横軸をそれぞれ変えたい、といった場合には gridExtra::grid.arrange を使う。
library(gridExtra) p1 <- ggplot(iris, aes(Sepal.Width, Sepal.Length)) + geom_point() p2 <- ggplot(iris, aes(Petal.Width, Petal.Length)) + geom_point() p3 <- ggplot(iris, aes(Sepal.Length, Petal.Length)) + geom_point() p4 <- ggplot(iris, aes(Sepal.Width, Petal.Width)) + geom_point() grid.arrange(p1, p2, p3, p4)

が、grid.arrange は引数を ドット ... で受け取ること、また呼び出すと直ちに描画を行うことから、以下のような場合は少し面倒だ。
- 描画が直ちに行われるため、サブプロットの描画設定をまとめて変更できない。単一のテーマを使う場合でも、個々のプロットそれぞれについてあらかじめ処理を行っておく必要がある。
- リストが渡せない。サブプロットの数を動的に変更したい場合は、リストに入れて
do.callが必要である。
というわけで、自作パッケージ {ggfortify} でこの辺の操作を少し便利にできるようにした。サブプロット周りの処理は ggmultiplot クラスで行う。
コンストラクタの引数にはリストが渡せるため、サブプロット数が変わる場合でも楽だと思う。また、インスタンスに対して + 演算子を用いることで ggplot と同じ処理を各プロットに対して適用できる。
# install.packages('ggfortify') library(ggfortify) p <- new('ggmultiplot', plots = list(p1, p2, p3, p4)) p # 略。上記 grid.arrange と同じ出力 # theme_bw を全サブプロットに適用 p + theme_bw()

サブプロットの要素には リストと同じようにアクセスできる。[ でのアクセスは ggmultiplot インスタンスに、[[ での アクセスは ggplot インスタンスになる。代入もできるため、一部のプロットにだけ処理を適用したい場合は以下のように書ける。
p[2:3] <- p[2:3] + stat_smooth(method = 'lm') p
+ 演算子の右項に ggplot インスタンスを指定するとサブプロットが追加できる。
p + (ggplot(iris, aes(Sepal.Width, fill = Species)) + geom_density())
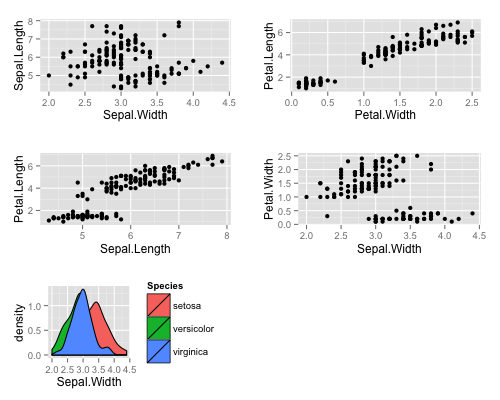
ggmultiplot 作成時に ncol もしくは nrow キーワードを指定することで、サブプロットの列数 / 行数が指定できる。
p <- new('ggmultiplot', plots = list(p1, p2, p3, p4), ncol = 3) p

autoplot との組み合わせ
{ggfortify} を使うと主要なクラスを ggplot2::autoplot 関数で描画できるようになる。autoplot にリストを渡した場合は、リストの各要素が autoplot 可能であればそれらをサブプロットとして描画する。
サンプルとして、{broom} パッケージの vignettes にある例をやってみたい。ここでは、適当なデータに対して クラスタ数を変えながら kmeans クラスタリングを行った結果をプロットしている。
これは autoplot を使って以下のように書ける。
kclusts <- data.frame(k=1:9) %>% dplyr::group_by(k) %>% dplyr::do(kclust = kmeans(iris[-5], .$k)) autoplot(kclusts$kclust, data = iris[-5], ncol = 3) + theme(legend.position = "none")
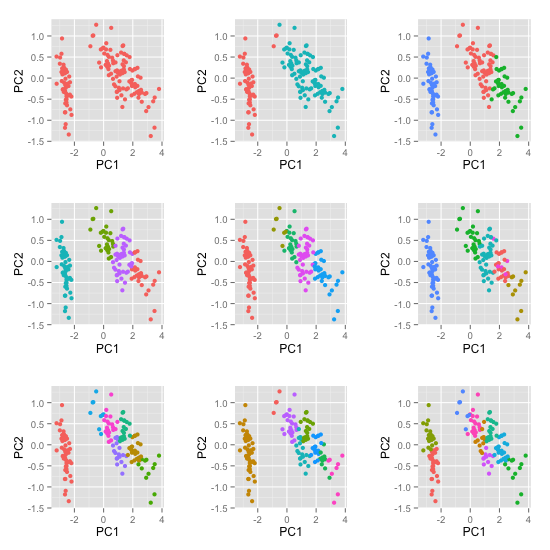
一行目は今なら {purrr} を使ったほうが簡潔でわかりやすくなると思う。
library(purrr) kclusts <- purrr::map(1:9, ~ kmeans(iris[-5], .)) autoplot(kclusts, data = iris[-5], ncol = 3) + theme(legend.position = "none") # 略
まとめ
{ggfortify} では ggmultiplot クラス、ならびに autoplot を利用してサブプロットを簡単に扱うことができる。

Rグラフィックスクックブック ―ggplot2によるグラフ作成のレシピ集
- 作者: Winston Chang,石井弓美子,河内崇,瀬戸山雅人,古畠敦
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/11/30
- メディア: 大型本
- この商品を含むブログ (2件) を見る
{purrr} でリストデータを操作する <2>
前の記事に続けて、{purrr} で {rlist} 相当の処理を行う。今回はレコードの選択とソート。
サンプルデータは前回と同じものを利用する。リストの表示も同じく Hmisc::list.tree を使う。
library(rlist) library(pipeR) library(purrr) packages <- list( list(name = 'dplyr', star = 979L, maintainer = 'hadley' , authors = c('hadley', 'romain')), list(name = 'ggplot2', star = 1546L, maintainer = 'hadley' , authors = c('hadley')), list(name = 'knitr', star = 1047L, maintainer = 'yihui' , authors = c('yihui', 'hadley', '...and all')) ) packages ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[3]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
レコードの選択
rlist Tutorial: Filtering 後半の内容。
ある条件にあてはまるレコードのうち最初の N 件だけを取得したいとき、{rlist} には専用の関数 list.find がある。{purrr} には対応する関数はないが、keep %>% head で同じ結果が得られる。
packages %>>% rlist::list.find(star >= 1000, 1) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::keep(~ .$star >= 1000) %>% head(n = 1) # 略
また、{rlist} には list.findi という インデックスを返す関数がある。purrr::keep では、引数に logical 型のベクトルを渡せば TRUE に対応する要素のみを残すことができる。
packages %>>% rlist::list.findi(star >= 1000, 2) ## [1] 2 3 purrr::keep(seq_along(packages), flatmap(packages, ~ .$star >= 1000)) %>% head(n = 2) # 略
条件にあてはまる最初のレコードだけを取得したいときは rlist::first もしくは purrr::detect。
rlist::list.first(packages, star >= 1000) ## x = list 4 (792 bytes) ## . name = character 1= ggplot2 ## . star = integer 1= 1546 ## . maintainer = character 1= hadley ## . authors = character 1= hadley purrr::detect(packages, ~ .$star >= 1000) # 略
レコード数がそこまで多くない場合は keep %>% head(n = 1) でよいと思うが、結果に 1 レベル目が入れ子として残ってしまう。上と同じ結果を得るには purrr::flatten ( unlist(recursive = FALSE) と同じ ) を通す。
purrr::keep(packages, ~ .$star >= 1000) %>% head(n = 1) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley purrr::keep(packages, ~ .$star >= 1000) %>% head(n = 1) %>% purrr::flatten() ## x = list 4 (792 bytes) ## . name = character 1= ggplot2 ## . star = integer 1= 1546 ## . maintainer = character 1= hadley ## . authors = character 1= hadley
同じく、最後のレコードを取得したいときは rlist::last もしくは purrr::detect(.right = TRUE)。これも自分としては keep %>% tail(n = 1) のほうが覚えやすい。
list.last(packages, star >= 1000) ## x = list 4 (920 bytes) ## . name = character 1= knitr ## . star = integer 1= 1047 ## . maintainer = character 1= yihui ## . authors = character 3= yihui hadley ... purrr::detect(packages, ~ .$star >= 1000, .right = TRUE) # 略 purrr::keep(packages, ~ .$star >= 1000) %>% tail(n = 1) %>% purrr::flatten() # 略
類似の処理として、{rlist} には list.take という最初の N レコードを取得する関数と list.skip という最初の N レコードを除外する関数がある。{purrr} には対応する関数はないが、組み込みの head と tail でよいのでは...。
packages %>>% rlist::list.take(2) ## x = list 2 (1696 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% head(n = 2) # 略 packages %>% rlist::list.skip(1) ## x = list 2 (1768 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... packages %>% tail(length(.) - 1) # 略
ある条件が初めて偽となるまでレコードを取得する場合は rlist::list.takeWhile もしくは purrr::head_while。
packages %>>% rlist::list.takeWhile(star <= 1500) ## x = list 1 (896 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain packages %>% purrr::head_while(~ .$star <= 1500) # 略
ある条件が初めて偽となったレコード以降を取得したい場合は rlist::list.skipWhile。{purrr} には対応する処理はないが、purrr::detect_index を使って以下のように書ける。
packages %>>% rlist::list.skipWhile(star <= 1500) ## x = list 2 (1768 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... purrr::detect_index(packages, ~ .$star > 1500) ## [1] 2 packages[purrr::detect_index(packages, ~ .$star > 1500):length(packages)] # 略
一方、{purrr} には tail_while という末尾からみて条件にあてはまるレコードだけを取得する関数がある。
packages %>% purrr::tail_while(~ .$star <= 1500) ## x = list 1 (968 bytes) ## . [[1]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
ある条件を満たす要素がいくつあるかを調べるには rlist::list.count。{purrr} なら keep %>% length でよい。
list.count(packages, star > 1000) ## [1] 2 purrr::keep(packages, ~ .$star > 1000) %>% length() # 略
{rlist} には リストの要素の名前を正規表現で抽出する list.match がある。{purrr} なら keep でできる。stringr::str_match はマッチした結果を matrix で返すため、complete.cases で logical に変換する。
data <- list(p1 = 1, p2 = 2, a1 = 3, a2 = 4) list.match(data, "p[12]") ## x = list 2 (416 bytes) ## . p1 = double 1= 1 ## . p2 = double 1= 2 complete.cases(stringr::str_match(names(data), 'p[12]')) ## [1] TRUE TRUE FALSE FALSE keep(data, complete.cases(stringr::str_match(names(data), 'p[12]'))) # 略
logical の処理
条件に当てはまるかどうかを logical ベクトルとして返すには rlist::list.is。これは rlist::list.mapv と同じなのでは...? {purrr} なら flatmap もしくは map_lgl。
packages %>>% rlist::list.is(star < 1500) ## [1] TRUE FALSE TRUE packages %>>% rlist::list.mapv(star < 1500) # 略 packages %>% purrr::flatmap(~ .$star < 1500) # 略 packages %>% purrr::map_lgl(~ .$star < 1500) # 略
また、logical のリスト全体の真偽値を以下のように取得できる
- 要素全てが
TRUEかどうか:rlist::list.allもしくはpurrr::every。 - 要素いずれかが
TRUEかどうか:rlist::list.anyもしくはpurrr::some。
自分は purrr::flatmap %>% all もしくは purrr::flatmap %>% any のほうが覚えやすい。
ただし、{purrr} v0.1.0 の every, some にはバグがあり正しく動作しない。下のスクリプトを実行するためには GitHub から最新の master をインストールする必要がある。
rlist::list.all(packages, star >= 500) ## [1] TRUE # install_github('hadley/purrr') # が必要 purrr::every(packages, ~ .$star >= 500) # 略 packages %>% purrr::flatmap(~ .$star >= 500) %>% all() # 略 rlist::list.any(packages, star >= 1500) ## [1] TRUE # install_github('hadley/purrr') # が必要 purrr::some(packages, ~ .$star >= 1500) packages %>% purrr::flatmap(~ .$star >= 1500) %>% any() # 略
要素の削除
{rlist} では list.remove で指定した名前をリストから除外できる。{purrr} には discard という条件にマッチする要素をリストから除外する関数がある ( purrr::keep とは逆 )。条件の否定をとれば keep でも書ける。
rlist::list.remove(data, c("p1", "p2")) ## x = list 2 (416 bytes) ## . a1 = double 1= 3 ## . a2 = double 1= 4 purrr::discard(data, names(data) %in% c("p1", "p2")) # 略 purrr::keep(data, !(names(data) %in% c("p1", "p2"))) # 略
{rlist} で、名前ではなく 条件式を使ってリストから除外したい場合は list.exclude。{purrr} の場合は 上と同じく discard もしくは keep。
packages %>>% rlist::list.exclude("yihui" %in% authors) ## x = list 2 (1696 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::discard(~ "yihui" %in% .$authors) # 略
また、{rlist} には 欠損などの異常値を リストから除去する list.clean という関数がある。下の例では リストの 1 レベル目にある欠損 "b" を除去している。{purrr} では同じく discard。
x <- list(a = 1, b = NULL, c = list(x = 1, y = NULL, z = logical(0L), w = c(NA, 1))) x ## x = list 3 (1040 bytes) ## . a = double 1= 1 ## . b = NULL 0 ## . c = list 4 ## . . x = double 1= 1 ## . . y = NULL 0 ## . . z = logical 0 ## . . w = double 2= NA 1 rlist::list.clean(x) ## x = list 2 (960 bytes) ## . a = double 1= 1 ## . c = list 4 ## . . x = double 1= 1 ## . . y = NULL 0 ## . . z = logical 0 ## . . w = double 2= NA 1 purrr::discard(x, ~ is.null(.)) # 略
rlist::list.clean は recursive = TRUE を指定することで 処理を再帰的に適用できる。{purrr} ではこういった再帰的な処理は (自分で関数を書かない限り) できないと思う。
rlist::list.clean(x, recursive = TRUE) ## x = list 2 (912 bytes) ## . a = double 1= 1 ## . c = list 3 ## . . x = double 1= 1 ## . . z = logical 0 ## . . w = double 2= NA 1
要素の更新
rlist Tutorial: Updating に相当する内容。
rlist::list.map や purrr::map を用いたリストの選択では、結果に含める属性を明示的に指定する必要があった。
これは対象のリストに選択したい属性が多い場合はすこし面倒だ。
rlist::list.update を使うと、もともとのリストの属性を残した上で 指定した要素を追加 / 更新できる。{purrr} で同じことをするには map + update_list。
packages %>>% rlist::list.update(lang = 'R', star = star + 1) ## x = list 3 (3160 bytes) ## . [[1]] = list 5 ## . . name = character 1= dplyr ## . . star = double 1= 980 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . . lang = character 1= R ## . [[2]] = list 5 ## . . name = character 1= ggplot2 ## . . star = double 1= 1547 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . . lang = character 1= R ## . [[3]] = list 5 ## . . name = character 1= knitr ## . . star = double 1= 1048 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . . lang = character 1= R packages %>% purrr::map(~ purrr::update_list(., lang = 'R', star = ~ .$star + 1)) # 略
一部の要素を取り除きたい場合は NULL を指定する。これは {purrr} も同じ。
packages %>>% rlist::list.update(maintainer = NULL, authors = NULL) ## x = list 3 (1464 bytes) ## . [[1]] = list 2 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . [[2]] = list 2 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . [[3]] = list 2 ## . . name = character 1= knitr ## . . star = integer 1= 1047 packages %>% purrr::map(~ purrr::update_list(., maintainer = NULL, authors = NULL)) # 略
ソート
rlist Tutorial: Sorting に相当する内容。
{rlist}, {purrr} とも 標準の sort ( 値のソート ) と order ( インデックスのソート ) それぞれに対応した関数を持つ。
x = c('a', 'c', 'd', 'b') sort(x) ## [1] "a" "b" "c" "d" order(x) ## [1] 1 4 2 3
sort のようにソートした値を得るためには rlist::list.sort もしくは purrr::sort_by。
packages %>>% rlist::list.sort(star) ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . [[3]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::sort_by('star') # 略
{rlist} では 列名を括弧でくくると逆順となる。{purrr} には対応する記法がないが、結果を rev で逆順にすればよい。
packages %>>% rlist::list.sort((star)) ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[2]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... ## . [[3]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain packages %>% purrr::sort_by('star') %>% rev() # 略
order のように結果をインデックスで得るためには rlist::list.order もしくは purrr::order_by。
rlist::list.order(packages, star) ## [1] 1 3 2 purrr::order_by(packages, 'star') ## [1] 1 3 2
まとめ
前回と同じく、{purrr} では map (flatmap)、keep (discard) でだいたいのことはできる。処理を少し変えたい場合は 組み込み関数を適当に組み合わせればよいため、追加での学習コストは低いと思う。
一方、{rlist} はパッケージの中だけで処理が完結するので、そちらにメリットを感じる方は {rlist} を使ったほうがよさそう。
さらに続きます。

- 作者: Hadley Wickham,石田基広,市川太祐,高柳慎一,福島真太朗
- 出版社/メーカー: 共立出版
- 発売日: 2016/01/23
- メディア: 単行本
- この商品を含むブログ (25件) を見る
{purrr} でリストデータを操作する <1>
R で関数型プログラミングを行うためのパッケージである {purrr}、すこし使い方がわかってきたので整理をしたい。RStudio のブログの記載をみると、とくにデータ処理フローを関数型のように記述することが目的のようだ。
The core of purrr is a set of functions for manipulating vectors (atomic vectors, lists, and data frames). The goal is similar to dplyr: help you tackle the most common 90% of data manipulation challenges.
ここでいう"関数型プログラミング言語"とは Haskell のような静的型の言語を想定しており、型チェック、ガード、リフトなど断片的に影響を受けたと思われる関数群をもつ。ただし、同時に Haskell を目指すものではない、とも明言されている。
そもそも R は Scheme の影響をうけているためか、関数型らしい言語仕様やビルトイン関数群 Map, Reduce, Filter などをすでに持っている。ただ、これらの関数群は引数の順序からパイプ演算子と組み合わせて使いにくい。{purrr} でこのあたりが使いやすくなっているとうれしい。
R: Common Higher-Order Functions in Functional Programming...
※ 上記の Map など、S のリファレンスに見当たらなかったため R 独自だと思っていますが、間違っていたら教えてください。
{purrr} によるリストの操作
{purrr} を使うとリストやベクトルの処理が書けるのかをまとめたい。リスト操作のためのパッケージである {rlist} とできること、記法をくらべてみる。
{purrr} を利用する目的は 処理を簡単に、見やすく書くことであるため、{rlist} と比べて可読性が悪いもの、複雑になるものは "できない" 処理として扱う。サンプルデータとして、R の著名パッケージに関連した情報のリストを用意した。
packages <- list( list(name = 'dplyr', star = 979L, maintainer = 'hadley' , authors = c('hadley', 'romain')), list(name = 'ggplot2', star = 1546L, maintainer = 'hadley' , authors = c('hadley')), list(name = 'knitr', star = 1047L, maintainer = 'yihui' , authors = c('yihui', 'hadley', '...and all')) ) packages ## x = list 3 (2632 bytes) ## . [[1]] = list 4 ## . . name = character 1= dplyr ## . . star = integer 1= 979 ## . . maintainer = character 1= hadley ## . . authors = character 2= hadley romain ## . [[2]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley ## . [[3]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ...
1レベル目がレコード、以降のレベルが各レコードの要素となっている。この記事では主に以下二つの操作に関連した機能について記載する。
- 要素に対する操作: 特定の要素を選択する。特定の要素を元に新しい要素をつくる (マッピングする)。
- レコードに対する操作: 特定のレコードを選択する。
準備: リストの表示
既定の print ではリストの階層構造がわかりにくく、出力も長い。わかりやすくするため Hmisc::list.tree を利用した出力を記載する。また、出力が同一のものは省略する。
library(Hmisc) l1 <- list(a = 1, b = c(3L, 4L), c = list(x = c(5, 6), y = 'AAA')) # 既定での表示 l1 ## $a ## [1] 1 ## ## $b ## [1] 3 4 ## ## $c ## $c$x ## [1] 5 6 ## ## $c$y ## [1] "AAA" # list.tree での表示 (名前 = 型 要素数 = 値 と読む) Hmisc::list.tree(l1) ## l1 = list 3 (968 bytes) ## . a = double 1= 1 ## . b = integer 2= 3 4 ## . c = list 2 ## . . x = double 2= 5 6 ## . . y = character 1= AAA
パッケージのロード
library(rlist) library(pipeR) library(purrr) library(dplyr)
要素の選択とマッピング
rlist Tutorial: Mapping に相当する内容。
要素の選択
R 標準の lapply 他 apply 系の関数に対応するのは rlist::list.map と purrr::map。purrr::map では第二引数に文字列を渡すとその要素の抽出、ラムダ式 ( ~ .$name )を渡すとその式の適用になる。
lapply(packages, function(x) { x$name }) ## x = list 3 (360 bytes) ## . [[1]] = character 1= dplyr ## . [[2]] = character 1= ggplot2 ## . [[3]] = character 1= knitr rlist::list.map(packages, name) # 略 purrr::map(packages, 'name') # 略 purrr::map(packages, ~ .$name) # 略
複数の要素を一度に選択したい場合、{rlist} には rlist::list.select という選択専用の関数がある。{purrr} では渡せる式が一つのため、ラムダ式を利用する。
rlist::list.select(packages, maintainer, star) ## x = list 3 (1488 bytes) ## . [[1]] = list 2 ## . . maintainer = character 1= hadley ## . . star = integer 1= 979 ## . [[2]] = list 2 ## . . maintainer = character 1= hadley ## . . star = integer 1= 1546 ## . [[3]] = list 2 ## . . maintainer = character 1= yihui ## . . star = integer 1= 1047 purrr::map(packages, ~ .[c('maintainer', 'star')]) # 略
補足 第二引数にベクトルを渡した場合の処理は、以下のように階層的な選択になる ( l[[c('a', 'b')]] と一緒)。
l2 <- list(list(a=list(b=1)), list(a=list(b=2))) l2 ## x = list 2 (1176 bytes) ## . [[1]] = list 1 ## . . a = list 1 ## . . . b = double 1= 1 ## . [[2]] = list 1 ## . . a = list 1 ## . . . b = double 1= 2 purrr::map(l2, c('a', 'b')) ## x = list 2 (152 bytes) ## . [[1]] = double 1= 1 ## . [[2]] = double 1= 2
{rlist}, {purrr} でのラムダ式
f <- function(.) {. + 1} に対応する無名関数を {rlist}, {purrr} それぞれの記法で書く。形式はチルダの有無以外は同じ。ドットが引数に対応する。
nums <- c(a = 3, b = 2, c = 1) rlist::list.map(nums, . + 1) ## x = list 3 (544 bytes) ## . a = double 1= 4 ## . b = double 1= 3 ## . c = double 1= 2 purrr::map(nums, ~ . + 1) # 略
{rilst} では ラムダ式中の .i で元のリストの位置 (インデックス) を、.name で名前 (names) をそれぞれ参照できる。purrr のラムダ式には対応する記法がないため、近いことをやるためには直接 map の引数として渡す必要がある。
rlist::list.map(nums, .i) ## x = list 3 (544 bytes) ## . a = integer 1= 1 ## . b = integer 1= 2 ## . c = integer 1= 3 purrr::map(seq_along(nums), ~ .) # namesは変わってしまう ## x = list 3 (216 bytes) ## . [[1]] = integer 1= 1 ## . [[2]] = integer 1= 2 ## . [[3]] = integer 1= 3
rlist::list.map(nums, paste0("Name: ", .name)) ## x = list 3 (688 bytes) ## . a = character 1= Name: a ## . b = character 1= Name: b ## . c = character 1= Name: c purrr::map(names(nums), ~ paste0("Name: ", .)) # namesは変わってしまう ## x = list 3 (360 bytes) ## . [[1]] = character 1= Name: a ## . [[2]] = character 1= Name: b ## . [[3]] = character 1= Name: c
また、内部的にはラムダ式を eval で評価をしているため、変数として扱われない位置にあるドットは評価されない。
purrr::map(nums, ~ list(. = 1)) ## x = list 3 (1312 bytes) ## . a = list 1 ## . . . = double 1= 1 ## . b = list 1 ## . . . = double 1= 1 ## . c = list 1 ## . . . = double 1= 1
要素の追加、変更
元となるリストの値から新しいリストを作りたい場合はラムダ式でリストを返す。
rlist::list.map(packages, list(star = star, had = 'hadley' %in% authors)) ## x = list 3 (1320 bytes) ## . [[1]] = list 2 ## . . star = integer 1= 979 ## . . had = logical 1= TRUE ## . [[2]] = list 2 ## . . star = integer 1= 1546 ## . . had = logical 1= TRUE ## . [[3]] = list 2 ## . . star = integer 1= 1047 ## . . had = logical 1= TRUE purrr::map(packages, ~ list(star = .$star, had = 'hadley' %in% .$authors)) # 略
結果の型の変更
結果をベクトルで取得したい場合、{rilst} では list.mapv。{purrr} では flatmap もしくは map_int ( map_xxx のように結果の型を指定できる関数群がある )。
rlist::list.mapv(packages, star) ## [1] 979 1546 1047 purrr::flatmap(packages, 'star') # 略 purrr::map_int(packages, 'star') # 略
data.frame としたい場合は rlist::list.stack。{purrr} では dplyr::bind_rows に渡す。
packages %>>% rlist::list.select(name, star) %>>% rlist::list.stack() ## name star ## 1 dplyr 979 ## 2 ggplot2 1546 ## 3 knitr 1047 packages %>% purrr::map(~ .[c('name', 'star')]) %>% dplyr::bind_rows()) # 略
関数の適用
返り値を変更せずに関数を適用するには rlist::list.iter と purrr::walk。パイプ処理の間に出力やプロットなど意図する返り値を持たない処理を挟み込む場合に利用する。
r <- rlist::list.iter(packages, cat(name, ":", star, "\n")) ## dplyr : 979 ## ggplot2 : 1546 ## knitr : 1047 r <- purrr::walk(packages, ~ cat(.$name, ":", .$star, "\n")) # 略
レコードの選択
rlist Tutorial: Filtering 前半の単純な条件での選択。
{rilst} では list.filter。{purrr} では keep。ラムダ式が使えるのは map などと同じ。
packages %>>% list.filter(star >= 1500) ## x = list 1 (840 bytes) ## . [[1]] = list 4 ## . . name = character 1= ggplot2 ## . . star = integer 1= 1546 ## . . maintainer = character 1= hadley ## . . authors = character 1= hadley packages %>% purrr::keep(~ .$star >= 1500) # 略
packages %>>% list.filter("yihui" %in% authors) ## x = list 1 (968 bytes) ## . [[1]] = list 4 ## . . name = character 1= knitr ## . . star = integer 1= 1047 ## . . maintainer = character 1= yihui ## . . authors = character 3= yihui hadley ... packages %>% purrr::keep(~ "yihui" %in% .$authors) # 略
まとめ
{purrr} によるリストデータの属性、レコードに対する操作を記載した。purrr::map と purrr::keep だけでもパイプ演算子 + ラムダ式と組み合わせて幅広い処理ができそうだ。
11/28追記 続きです。

- 作者: Hadley Wickham,石田基広,市川太祐,高柳慎一,福島真太朗
- 出版社/メーカー: 共立出版
- 発売日: 2016/01/23
- メディア: 単行本
- この商品を含むブログ (25件) を見る
Python Jupyter + pandas で DataFrame 表示をカスタマイズする
先日 pandas v0.17.1 がリリースされた。v0.17.0 に対するバグフィックスがメインだが、以下の追加機能もあるため その内容をまとめたい。
HTML 表示のカスタマイズ
Jupyer 上では pandasの DataFrame は自動的に HTML として描画される。この HTML に対して、さまざまな CSS を柔軟に設定できるようになった。
このエントリでは、添付した公式ドキュメントとは少し違う例を記載する。
重要 公式ドキュメントにも記載がされているが v0.17.1 時点で開発中 / Experimental な追加のため、今後 破壊的な変更が発生する可能性がある。ご要望やお気づきの点があれば GitHub issue か @ ください。
以降、Jupyter にて実行する。まず DataFrame を作成して表示する。
import pandas as pd import numpy as np np.__version__ # '1.10.1' pd.__version__ # u'0.17.1' np.random.seed(1) df = pd.DataFrame({'name': list('abcdefg'), 'values1': np.random.randn(7), 'values2': np.random.randn(7)}) df

v0.17.0 にて DataFrame に DataFrame.style アクセサが追加され、これを経由して種々のカスタマイズができる。例えば、各セルの値に応じてカラーマップを適用するには Styler.background_gradient()。
type(df.style) # pandas.core.style.Styler df.style.background_gradient(cmap='winter')

カラーマップの一部範囲の色のみ利用する場合は low, high キーワードで範囲を指定する。
df.style.background_gradient(cmap='winter', low=2.)

特定の列にのみ Styler を適用する場合は subset キーワード。
df.style.background_gradient(cmap='winter', low=2., subset=['values1'])

さらに特定のセルにのみ適用する場合は 対象セルを指定する pd.IndexSlice インスタンスを subset として指定する。
pd.IndexSlice[2:5, ['values1']] # (slice(2, 5, None), ['values1']) # .loc に渡した場合は対象セルのみをスライス df.loc[pd.IndexSlice[2:5, ['values1']]]

df.style.background_gradient(cmap='winter', low=2., subset=pd.IndexSlice[2:5, ['values1']])

Styler は background_gradient 以外にも 以下のようなメソッドをもつ。
Styler.highlight_max(): 列もしくは行の最大値を指定して色分け。Styler.highlight_min(): 列もしくは行の最小値を指定して色分け。Styler.highlight_null():NaNを指定して色分け。Styler.background_gradient(): 上述。Styler.bar(): 各セルの値に応じて棒グラフのように背景色表示。
df.iloc[1, 1] = np.nan df.style.highlight_null()

さらに、Styler.set_properties を利用すると任意の CSS を全セルに対して適用できる。数値以外のセルを色分けするにはこれを使えばよい。
df.style.set_properties(**{'background-color': '#00ff7f'})

また、Styler はチェインできる。
(df.style.
set_properties(**{'background-color': '#00ff7f'}).
background_gradient(cmap='winter', low=2.).
highlight_null())

より柔軟な条件分けのため、 Styler は .apply 系のメソッドも持つ。使い方は通常の DataFrame.apply や .applymap と同じだが、値そのものではなく CSS を返す関数を適用する。
Styler.applymap: 各セルに対して CSS を返す関数を適用 (関数への入力はスカラー)。Styler.apply: 各列もしくは各行に対して CSS を返す関数を適用 (関数への入力は 各列/各行の値からなるSeries)。
参考 Python pandas データのイテレーションと関数適用、pipe - StatsFragments
セルの値が 0 より小さい場合は 赤 / 太字で表示する場合は Styler.applymap。
def highlight_negative(val): if val < 0: return 'color: {0}; font-weight: bold'.format('red') else: return 'color: {0}'.format('black') df.style.applymap(highlight_negative)

各行について、values1 列の値が values2 列の値より大きいときに values1 列のみ 赤背景で表示したい。行ごとに関数を適用するため、axis=1 を指定する。highlight_values1 関数が返す リストの各要素が、各列に適用される CSS に対応している。
def highlight_values1(s): if s['values1'] > s['values2']: # return ['', 'background-color: red', ''] else: # スタイル変更しない場合は空の文字列のリストを返す return [''] * len(s) df.style.apply(highlight_values1, axis=1)
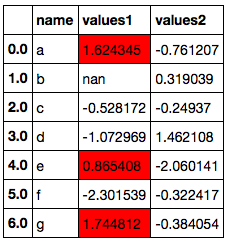
最後に、Jupyter の widget と組み合わせることで動的なスタイル変更ができる。以下ではスライドバーの位置によって フォントの大きさを変更している。
from IPython.html import widgets @widgets.interact def f(s=(5, 30)): return (df.style.background_gradient('winter', low=2.).set_properties(**{'font-size': '{0}px'.format(s)}))


Jupyter 以外に 同様の出力を HTML として埋め込みたい場合は、Styler.render() を利用する。
style = df.style.background_gradient('winter', low=2.) style.render() # 略 (HTML が文字列として出力される)
ピボットテーブル + グラフ表示
また Jupyter 上での DataFrame の扱いに関連して、データそのものを GUI でインタラクティブに操作 / グラフ表示ができるpivottablejs というパッケージがある。
まとめ
pandas v0.17.1 で追加された DataFrame の HTML 表示のカスタマイズ方法について記載した。

Pythonによるデータ分析入門 ―NumPy、pandasを使ったデータ処理
- 作者: Wes McKinney,小林儀匡,鈴木宏尚,瀬戸山雅人,滝口開資,野上大介
- 出版社/メーカー: オライリージャパン
- 発売日: 2013/12/26
- メディア: 大型本
- この商品を含むブログ (11件) を見る
R パッケージを CRAN で公開する
少し前に 自作パッケージを CRAN で公開したのだが ブログに書くのを忘れていた。CRAN 公開時の注意点に関して、日本語の説明があまりない / 情報が古いので簡単にまとめたい。
パッケージの作成
この資料を読みましょう。
www.slideshare.net
継続的インテグレーション (CI)
Travis CI は R をサポート (community supportだが) しているため、.travis.yml に2行記載するだけで利用できる。CI 上でパッケージのチェック (R CMD check) も走るので利用したほうが楽。
複数の環境でテストを実行したい場合、Travis CI では Build Matrix という仕組みがある。R では実行したいテストの数だけ env: タグ中に環境変数を記載するのがよいと思う。自分のパッケージでは {ggplot2} の公式版 / GitHub 上の開発版で 2 つのテストを実行している。
そのほかの CI ツール
ほか、R から CI を利用するツールとして、以下のようなものもある。
- r-appveyor: Windows 用の CI 環境である AppVeyor が利用できる。
- r-builder: Travis CI を R から利用する。R-devel が利用できる。
vignettes の作成
11/30追記 {knitr} で作るのがカンタン。vignettes 化したい {knitr}ドキュメント中に以下のようなタグを含めればよい。VignetteIndexEntry は vignettes へのリンクテキストとして利用される。ASCII文字以外を含める場合、DESCRIPTION ファイル中で Encoding: UTF-8 を指定する。
<!--
%\VignetteEngine{knitr::knitr}
%\VignetteIndexEntry{Introduction to ggfortify package}
-->
参考 Package vignettes: How to build package vignettes with knitr | knitr
パッケージのチェック
開発版 (R-devel) の日次ビルド を利用して、R CMD Check --as-cran package_name.tar.gz で行う。開発版のほうが警告が厳しく出るようだ。
自分は 開発版環境を AWS 上に構築している。 Amazon Linux であれば以下のスクリプトでインストールできると思う。
R CMD check では ERROR, WARNING はもちろん、NOTE も "checking CRAN incoming feasibility" 1 つを除いて全て表示されないようにするのが望ましい。
特に、{dplyr} 等で NSE を使っている場合は xxx: no visible binding for global variable 'xxx' といった NOTE が表示されるが、これらについても CRAN 管理者から指摘される場合がある。 SE を使うか、関数中に (ダミーとして) 同名の変数を作るなどで回避しておくのがよい。
パッケージのサブミット
FTP で直接あげてはダメ。Submit package to CRAN に必要事項を記載して Upload package をする。その後、確認のメールが来たら Confirmation する。
公開待ちのパッケージは 以下の FTP サーバにて確認できる。
パッケージによっては 拡張子の後に以下のようなステータス情報が付けられる場合がある。これは CRAN 公式の情報ではなく、いくつかのフォーラム文書をみて自分が推測したものなので間違っているかもしれない。
.save:UploadされたがConfirmationされていないもの。.pending: CRAN 管理者にて何らかの理由でペンディングされているもの (放っておくしかない)。.noemail:Confirmationはされているが確認時のメールが CRAN に飛んでいないもの。
参考 What are the steps in submitting an R-package to CRAN and how long does each step take? - Stack Overflow (ほぼ情報ない)
その後の対応
CRAN 管理者から指摘がきたら適切に対応してください。
パッケージの公開
11/30追記 CRAN 上でのテストとレビューに問題がなければ CRAN 公開後にメールがくる。その後 1日程度すると Windows 用のバイナリのビルドが自動的に行われ、改めて通知がくる。
Rust で階層的クラスタリング
こちらの続き。
階層的クラスタリングについてはこちらを。サンプルデータもリンク先のものを利用させていただく。
再帰的なデータ構造の作成
階層的クラスタリングでは もっとも距離の小さいクラスタ同士を順に併合していく。そのため併合元となるクラスタ (子) と 作成される新しいクラスタ (親) に親子関係をもたせたい。
こういったとき、親から子に参照を持たせればいいかな? と思うのだが、Rust でこれをやろうとすると "親のインスタンスが存在している間は子への参照が有効となっている" ということを lifetime として記述しなければならず、すこし面倒である。
そのため、参照ではなく所有権を移してしまうのが一般的なようだ。
参考 The idea behind a recursive struct - help - The Rust Programming Language Forum
階層的クラスタリング
場合分けをシンプルにするため、ほぼ同一のロジックである最近隣法、最遠隣法、群平均法を書いた。
extern crate csv; extern crate nalgebra; extern crate num; extern crate rand; use csv::Reader; use nalgebra::{DVec, DMat, RowSlice, Iterable}; use num::{Signed, Zero, Float}; use std::f64; use std::io; use std::ops::Index; use std::str; use std::str::FromStr; fn main() { let data = "名前,算数,理科,国語,英語,社会 田中,89,90,67,46,50 佐藤,57,70,80,85,90 鈴木,80,90,35,40,50 本田,40,60,50,45,55 川端,78,85,45,55,60 吉野,55,65,80,75,85 斉藤,90,85,88,92,95"; // http://burntsushi.net/rustdoc/csv/ let mut reader = csv::Reader::from_string(data).has_headers(true); // http://nalgebra.org/doc/nalgebra/struct.DMat.html let dx = read_csv_f64(&mut reader); println!("最近隣法"); let mut hclust = HClust::new(ClusterDistance::Single); hclust.fit(&dx); println!("最遠隣法"); let mut hclust = HClust::new(ClusterDistance::Complete); hclust.fit(&dx); println!("群平均法"); let mut hclust = HClust::new(ClusterDistance::Average); hclust.fit(&dx); } enum ClusterDistance { Single, // 最近隣法 Complete, // 最遠隣法 Average, // 群平均法 } struct HClust { dist_mat: DMat<f64>, // 距離行列 method: ClusterDistance, // クラスタ間距離の計算方法 clusters: Vec<Cluster> } impl HClust { fn new(method: ClusterDistance) -> HClust { HClust { // dummy dist_mat: DMat::from_elem(1, 1, 1.), method: method, clusters: vec![] } } fn fit(&mut self, data: &DMat<f64>) { self.dist_mat = self.get_dist_matrix(&data); // クラスタを初期化 for i in 0..data.nrows() { let c = Cluster::from_nodes(vec![i]); self.clusters.push(c); } while self.clusters.len() > 1 { self.fit_step(); } } /// もっとも距離の小さいクラスタを併合 fn fit_step(&mut self) { let mut tmp_i = 0; let mut tmp_j = 0; let mut current_dist = f64::MAX; for i in 0..self.clusters.len() { for j in (i + 1)..self.clusters.len() { let d = self.get_cluster_dist(&self.clusters[i], &self.clusters[j]); if d < current_dist { current_dist = d; tmp_i = i; tmp_j = j; } } } // R と比較するための出力を表示 println!("Mergeing clusters: {:?} and {:?}: Distance {:?}", &self.clusters[tmp_i].nodes, &self.clusters[tmp_j].nodes, (current_dist * 1e+6).round() / 1e+6); let mut new_clusters: Vec<Cluster> = vec![]; for (i, n) in self.clusters.iter().enumerate() { if i != tmp_i && i != tmp_j { let n2 = Cluster::from_nodes(n.nodes.clone()); new_clusters.push(n2); } } // Vec から要素を取り出し & 所有権を取得 // コンストラクタに所有権ごと渡す let new = Cluster::from_clusters(self.clusters.swap_remove(tmp_j), self.clusters.swap_remove(tmp_i)); new_clusters.push(new); self.clusters = new_clusters; } /// クラスタに含まれるノード間の距離を計算 fn get_cluster_dist(&self, c1: &Cluster, c2: &Cluster) -> f64 { let mut dists: Vec<f64> = vec![]; for i in &c1.nodes { for j in &c2.nodes { dists.push(self.get_node_dist(*i, *j)); } } return self.fold_dist_vec(dists); } /// クラスタに含まれるノード間の距離から、クラスタ間の距離を計算 fn fold_dist_vec(&self, dists: Vec<f64>) -> f64 { match self.method { ClusterDistance::Single => { return dists.iter().fold(f64::MAX, |a, b| a.min(*b)); }, ClusterDistance::Complete => { return dists.iter().fold(f64::MIN, |a, b| a.max(*b)); }, ClusterDistance::Average => { return dists.iter().fold(0., |a, b| a + b) / (dists.len() as f64); }, } } /// 距離行列を作成 fn get_dist_matrix(&self, data: &DMat<f64>) -> DMat<f64> { // 各列は 0番目 から ノード数-1 番目までのノードに対応 - // 各行は 1番目 から ノード数番目 までのノードに対応 return DMat::from_fn(data.nrows() - 1, data.nrows() - 1, |i, j| if i >= j { euc_dist(&data.row_slice(i + 1, 0, data.ncols()), &data.row_slice(j, 0, data.ncols())) } else { 0. }); } /// 距離行列を利用して ノード間の距離を求める fn get_node_dist(&self, i: usize, j: usize) -> f64 { match i > j { true => *self.dist_mat.index((i - 1, j)), false => *self.dist_mat.index((j - 1, i)) } } } struct Cluster { nodes: Vec<usize>, children: Vec<Cluster> } impl Cluster { fn from_nodes(nodes: Vec<usize>) -> Cluster { Cluster { nodes: nodes, children: vec![] } } /// 二つのクラスタを併合 fn from_clusters(left: Cluster, right: Cluster) -> Cluster { let mut id = vec![]; for i in &left.nodes { id.push(*i); } for j in &right.nodes { id.push(*j); } Cluster { nodes: id, children: vec![left, right] } } } // ユークリッド距離 fn euc_dist<T: Float + Signed>(vec1: &DVec<T>, vec2: &DVec<T>) -> T { let mut val: T = Zero::zero(); for (v1, v2) in vec1.iter().zip(vec2.iter()) { val = val + num::pow(num::abs(*v1 - *v2), 2); } return val.sqrt(); } /// csv::Reder から f64 に変換できるカラムのみ読み込み pub fn read_csv_f64<R: io::Read>(reader: &mut Reader<R>) -> DMat<f64> { let mut x:Vec<f64> = vec![]; let mut nrows: usize = 0; for record in reader.byte_records().map(|r| r.unwrap()) { // f64 に変換できる列のみ読み込み for item in record.iter().map(|i| str::from_utf8(i).unwrap()) { match f64::from_str(item) { Ok(v) => x.push(v), Err(_) => {} }; } nrows += 1; } let ncols = x.len() / nrows; // http://nalgebra.org/doc/nalgebra/struct.DMat.html return DMat::from_row_vec(nrows, ncols, &x); }
出力。
# 最近隣法 # Mergeing clusters: [1] and [5]: Distance 12.409674 # Mergeing clusters: [2] and [4]: Distance 21.307276 # Mergeing clusters: [0] and [4, 2]: Distance 28.478062 # Mergeing clusters: [6] and [5, 1]: Distance 38.105118 # Mergeing clusters: [3] and [4, 2, 0]: Distance 47.106263 # Mergeing clusters: [5, 1, 6] and [4, 2, 0, 3]: Distance 54.313902 # 最遠隣法 # Mergeing clusters: [1] and [5]: Distance 12.409674 # Mergeing clusters: [2] and [4]: Distance 21.307276 # Mergeing clusters: [0] and [4, 2]: Distance 33.778692 # Mergeing clusters: [6] and [5, 1]: Distance 45.585085 # Mergeing clusters: [3] and [4, 2, 0]: Distance 60.133186 # Mergeing clusters: [5, 1, 6] and [4, 2, 0, 3]: Distance 91.531415 # 群平均法 # Mergeing clusters: [1] and [5]: Distance 12.409674 # Mergeing clusters: [2] and [4]: Distance 21.307276 # Mergeing clusters: [0] and [4, 2]: Distance 31.128377 # Mergeing clusters: [6] and [5, 1]: Distance 41.845102 # Mergeing clusters: [3] and [4, 2, 0]: Distance 53.305906 # Mergeing clusters: [5, 1, 6] and [4, 2, 0, 3]: Distance 69.922956
R の結果。コメントは ノードが 0 から始まるとして (上の出力にあわせて ) 記載。作成されるクラスタはどの方法でも同じだが、クラスタ間の距離 (3 列目) は異なる。
# 最近隣法 hc <- hclust(seiseki.d, method = "single") cbind(hc$merge, hc$height) # [,1] [,2] [,3] # [1,] -2 -6 12.40967 # ノード 1 と ノード 5 を併合 # [2,] -3 -5 21.30728 # ノード 2 と ノード 4 を併合 # [3,] -1 2 28.47806 # ノード 0 と ノード [2, 4] からなるクラスタを併合 # [4,] -7 1 38.10512 # ノード 6 と ノード [1, 5] からなるクラスタを併合 # [5,] -4 3 47.10626 # ノード 3 と ノード [0, 2, 4] からなるクラスタを併合 # [6,] 4 5 54.31390 # ノード [1, 5, 6] と ノード [0, 2, 3, 4] からなるクラスタを併合 # 最遠隣法 hc <- hclust(seiseki.d, method = "complete") cbind(hc$merge, hc$height) # [,1] [,2] [,3] # [1,] -2 -6 12.40967 # ノード 1 と ノード 5 を併合 # [2,] -3 -5 21.30728 # ノード 2 と ノード 4 を併合 # [3,] -1 2 33.77869 # ノード 0 と ノード [2, 4] からなるクラスタを併合 # [4,] -7 1 45.58509 # ノード 6 と ノード [1, 5] からなるクラスタを併合 # [5,] -4 3 60.13319 # ノード 3 と ノード [0, 2, 4] からなるクラスタを併合 # [6,] 4 5 91.53142 # ノード [1, 5, 6] と ノード [0, 2, 3, 4] からなるクラスタを併合 # 群平均法 hc <- hclust(seiseki.d, method = "average") cbind(hc$merge, hc$height) # [,1] [,2] [,3] # [1,] -2 -6 12.40967 # ノード 1 と ノード 5 を併合 # [2,] -3 -5 21.30728 # ノード 2 と ノード 4 を併合 # [3,] -1 2 31.12838 # ノード 0 と ノード [2, 4] からなるクラスタを併合 # [4,] -7 1 41.84510 # ノード 6 と ノード [1, 5] からなるクラスタを併合 # [5,] -4 3 53.30591 # ノード 3 と ノード [0, 2, 4] からなるクラスタを併合 # [6,] 4 5 69.92296 # ノード [1, 5, 6] と ノード [0, 2, 3, 4] からなるクラスタを併合
2016/12/20 追記 上記以外の手法を追加 / Lance-Williams formulation で書き直した。
Rust で主成分分析
Rust で重回帰に続き、今日は 主成分分析をやりたい。
- Rust(Nightly) rustc 1.6.0-nightly (d5fde83ae 2015-11-12)
- rust-csv 0.14.3 : CSV の読み込みに利用
- nalgebra 0.3.2 : 行列/ベクトルの処理に利用
rust-csv での CSV ファイルの読み込み
今回は ローカルのCSV ファイル (iris.csv) を読みとる処理を加える。CSV が単一の型しか含まない場合、 前の記事 のように読み取った値をそのまま Vec に追加していけばよい。
iris のように複数の型を含むデータから特定の型のみを抜き出すには以下のような処理を書く必要がある。
まず Reader.byte_records() で各行のエントリをバイト列として読み取る。
let mut reader = csv::Reader::from_file(path).unwrap().has_headers(false); for record in reader.byte_records().map(|r| r.unwrap()) { println!("{:?}", record); } // [[53, 46, 49], [51, 46, 53], [49, 46, 52], [48, 46, 50], [73, 114, 105, 115, 45, 115, 101, 116, 111, 115, 97]] // [[52, 46, 57], [51, 46, 48], [49, 46, 52], [48, 46, 50], [73, 114, 105, 115, 45, 115, 101, 116, 111, 115, 97]] // ...
読み取ったバイト列を文字列に変換。
for record in reader.byte_records().map(|r| r.unwrap()) { for item in record.iter().map(|i| str::from_utf8(i).unwrap()) { println!("{:?}", item); } } // "5.1" // "3.5" // "1.4" // "0.2" // "Iris-setosa" // "4.9" // ...
文字列を f64 に変換し、成功したもののみ残す。
for record in reader.byte_records().map(|r| r.unwrap()) { // f64 に変換できる列のみ読み込み for item in record.iter().map(|i| str::from_utf8(i).unwrap()) { match f64::from_str(item) { Ok(v) => println!("{}", v), Err(e) => {} }; } } // 5.1 // 3.5 // 1.4 // 0.2 // 4.9 // ...
主成分分析
nalgebra では 行列の固有ベクトル/固有値を nalgebra::eigen_qr で計算できるが、この関数は 動的サイズの行列 DMat では利用できない。そのため、データの入力次元にあわせた行列 Mat4 を使った。
現状の nalgebra::DMat で任意の入力次元に対応した処理を書くためには固有値計算を自力でやる必要がある...と思う。
extern crate csv; extern crate nalgebra; extern crate num; use std::f64; use std::str; use std::str::FromStr; use std::vec::Vec; use nalgebra::{DVec, DMat, Mat4, Mean, ColSlice, Iterable, Transpose}; fn main() { // "iris" データを使用 // http://aima.cs.berkeley.edu/data/iris.csv let path = "./data/iris.csv"; // http://burntsushi.net/rustdoc/csv/ let mut reader = csv::Reader::from_file(path).unwrap().has_headers(false); let mut x:Vec<f64> = vec![]; let mut nrows: usize = 0; for record in reader.byte_records().map(|r| r.unwrap()) { // f64 に変換できる列のみ読み込み for item in record.iter().map(|i| str::from_utf8(i).unwrap()) { match f64::from_str(item) { Ok(v) => x.push(v), Err(e) => {} }; } nrows += 1; } let ncols = x.len() / nrows; // http://nalgebra.org/doc/nalgebra/struct.DMat.html let dx = DMat::from_row_vec(nrows, ncols, &x); // 主成分分析 let mut pca = PCA::new(ncols, true); pca.fit(&dx); // 結果は小数点以下 5 桁に丸めて表示 println!("主成分 (center=true)\n{:?}", &round(&mut pca.rotation, 5)); println!("主成分得点\n{:?}", &round(&mut pca.transform(&dx), 5)); } struct PCA { center: bool, // センタリングするかどうか nfeatures: usize, // 入力の次元 rotation: DMat<f64> // 主成分 } impl PCA { fn new(nfeatures: usize, center: bool) -> PCA { PCA { center: center, // 固有値計算に Mat4 を使うため、次元は 4 に固定 nfeatures: match nfeatures { 4 => nfeatures, _ => panic!("Number of features must be 4") }, rotation: DMat::new_ones(nfeatures, nfeatures) } } fn get_centers(&self, data: &DMat<f64>) -> DVec<f64> { // センタリングに用いるベクトルを計算 return match self.center { true => data.mean(), false => DVec::from_elem(self.nfeatures, 0.) }; } fn fit(&mut self, data: &DMat<f64>) { let nrows = data.nrows(); let centers = self.get_centers(&data); // 偏差平方和積和行列 // Dmat::from_fn で、行番号 i, 列番号 j を引数とする関数から各要素の値を生成できる let smx = DMat::from_fn(self.nfeatures, self.nfeatures, |i, j| sum_square(&data.col_slice(i, 0, nrows), &data.col_slice(j, 0, nrows), centers[i], centers[j])); // DMat では eigen_qr が使えないため Mat4 に変換 let smxv = smx.to_vec(); let smx4 = Mat4::new(smxv[0], smxv[1], smxv[2], smxv[3], smxv[4], smxv[5], smxv[6], smxv[7], smxv[8], smxv[9], smxv[10], smxv[11], smxv[12], smxv[13], smxv[14], smxv[15]); // 固有ベクトル、固有値を計算 let (evec, eval) = nalgebra::eigen_qr(&smx4, &1e-8, 10000); let mut vals: Vec<f64> = vec![]; for row in evec.transpose().as_array() { vals.extend(row); } self.rotation = DMat::from_row_vec(self.nfeatures, self.nfeatures, &vals); } fn transform(&mut self, data: &DMat<f64>) -> DMat<f64> { // 主成分得点を計算 let centers = self.get_centers(&data); let cdata = DMat::from_fn(data.nrows(), data.ncols(), |i, j| data[(i, j)] - centers[j]); return cdata * &self.rotation; } } fn sum_square(vec1: &DVec<f64>, vec2: &DVec<f64>, m1: f64, m2: f64) -> f64 { // 平方和 let mut val: f64 = 0.; for (v1, v2) in vec1.iter().zip(vec2.iter()) { val += (v1 - m1) * (v2 - m2); } return val; } fn round(data: &mut DMat<f64>, decimals: usize) -> DMat<f64> { // DMat の各要素を指定された有効数字で丸め let nrows = data.nrows(); let ncols = data.ncols(); let d: f64 = num::pow(10., decimals); // round() は常に整数で丸めるため、有効数字の処理を別途行う let vals: Vec<f64> = data.as_mut_vec().iter().map(|x| (x * d).round() / d).collect(); return DMat::from_col_vec(nrows, ncols, &vals); }
実行結果。
# 主成分 (center=true) # 0.36159 -0.65654 0.581 0.31725 # -0.08227 -0.72971 -0.59642 -0.32409 # 0.85657 0.17577 -0.07252 -0.47972 # 0.35884 0.07471 -0.54906 0.75112 # 主成分得点 # -2.68421 -0.32661 0.02151 0.00101 # -2.71539 0.16956 0.20352 0.0996 # -2.88982 0.13735 -0.02471 0.0193 # -2.74644 0.31112 -0.03767 -0.07596 # -2.72859 -0.33392 -0.09623 -0.06313 # ...
R の結果。
biris = read.csv("iris.csv", header=FALSE) # 主成分 prcomp(biris[-5]) # Standard deviations: # [1] 2.0554417 0.4921825 0.2802212 0.1538929 # # Rotation: # PC1 PC2 PC3 PC4 # V1 0.36158968 -0.65653988 0.58099728 0.3172545 # V2 -0.08226889 -0.72971237 -0.59641809 -0.3240944 # V3 0.85657211 0.17576740 -0.07252408 -0.4797190 # V4 0.35884393 0.07470647 -0.54906091 0.7511206 # 主成分得点 head(prcomp(biris[-5])$x, n = 5) # PC1 PC2 PC3 PC4 # [1,] -2.684207 -0.3266073 0.02151184 0.001006157 # [2,] -2.715391 0.1695568 0.20352143 0.099602424 # [3,] -2.889820 0.1373456 -0.02470924 0.019304543 # [4,] -2.746437 0.3111243 -0.03767198 -0.075955274 # [5,] -2.728593 -0.3339246 -0.09622970 -0.063128733
補足 R 組み込みの iris データと berkeley.edu からダウンロードできる iris データは一部のレコードが異なる。そのため、R の iris データを利用した場合は上と同じ結果にはならない。
iris[apply(iris[-5] != biris[-5], 1, any), ] # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # 35 4.9 3.1 1.5 0.2 setosa # 38 4.9 3.6 1.4 0.1 setosa biris[apply(iris[-5] != biris[-5], 1, any), ] # V1 V2 V3 V4 V5 # 35 4.9 3.1 1.5 0.1 Iris-setosa # 38 4.9 3.1 1.5 0.1 Iris-setosa
2016/12/20追記 rulinalg の SVD を使って書き直した。また、上のプログラムでは .transform 時のセンタリングを予測データから行なってしまっているが、これもおかしいので修正。