R で 状態空間モデル: {dlm} の最尤推定を可視化する
{dlm} において、状態空間モデルが最尤推定される過程がみたい。以下内容の補足的なエントリ。
「状態空間時系列分析入門」では 引き続き 第8章 に相当。

- 作者: J.J.F.コマンダー,S.J.クープマン,Jacques J.F. Commandeur,Sime Jan Koopman,和合肇
- 出版社/メーカー: シーエーピー出版
- 発売日: 2008/09
- メディア: 単行本
- 購入: 2人 クリック: 4回
- この商品を含むブログを見る
データの準備
データは著者のサポートサイト、An Introduction to State Space Time Series Analysis から入手できる。
library(dlm) # install_github('sinhrks/ggfortify') library(ggfortify) ukdrivers <- read.table('UKdriversKSI.txt', skip = 1) ukdrivers <- ts(ukdrivers[[1]], start = c(1969, 1), frequency = 12) ukdrivers <- log(ukdrivers) y <- ukdrivers
モデルの作成と状態推定値のプロット
サンプルとして、第3章3節 確率的レベルと確定的傾きをもつローカル線形トレンドモデル を作成する。
parm <- log(c(var(y), 0.001)) buildFun <- function(parm){ dlmModPoly(order = 2, dV = exp(parm[1]), dW = c(exp(parm[2]), 0)) } dlmFit <- dlmMLE(y, parm = parm, build = buildFun) # カルマンフィルタ dlmFiltered <- dlmFilter(y, buildFun(dlmFit$par)) # 平滑化 dlmSmoothed <- dlmSmooth(y, buildFun(dlmFit$par))
第8章ではモデルの推定値として 3 つの値が出てくるため、その差異がわかるようにプロットする。用語は状態空間時系列分析入門のものを利用。
p <- autoplot(y) p <- autoplot(dlmFiltered$a[, 1], colour = 'red', linetype = 'dashed', p = p) p <- autoplot(dlmFiltered$m[, 1], colour = 'red', p = p) p <- autoplot(dlmSmoothed, colour = 'blue', p = p) p + ylim(c(6.8, 8))
- 黒実線 (
y): 観測時系列 - 赤破線 (
dlmFiltered$a): カルマンフィルタの1期先予測 (予測ステップでの予測値) - 赤実線 (
dlmFiltered$m): カルマンフィルタのフィルタ化状態 (更新ステップでの予測値) - 青実線 (
dlmSmoothed): 平滑化状態
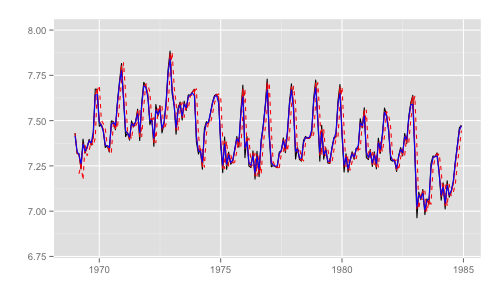
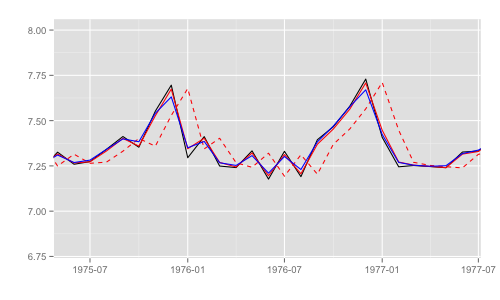
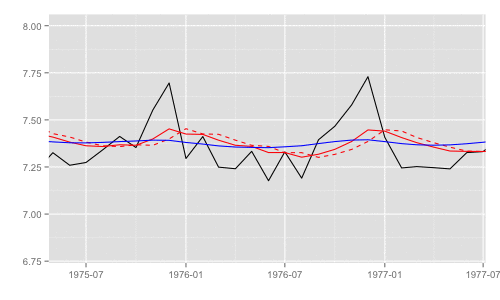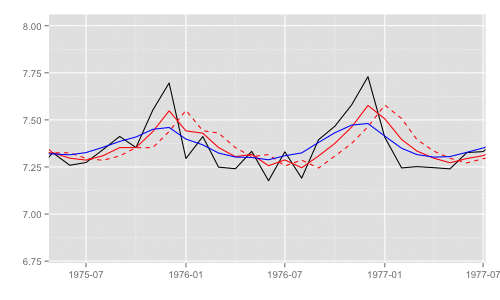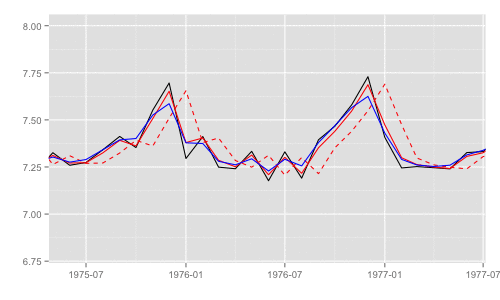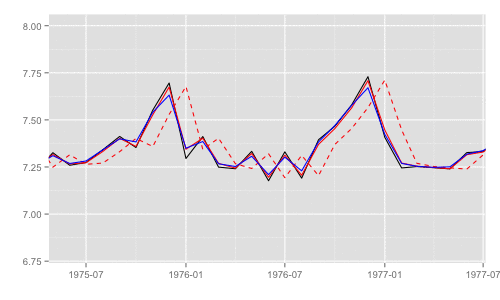
一部期間を拡大するとそれぞれの差異がわかる。これらのうち、最尤推定時の尤度計算式中で直接 利用されるのは "1期先予測" 時の予測誤差 (赤破線) とその標準偏差。
p + xlim(as.Date('1975', '%Y'), as.Date('1977', '%Y')) + ylim(c(6.8, 8))
補足 上のモデルでは FF = [1, 0] のため、観測値の推定値 == 状態推定値。
dlmFiltered$mod$FF # [,1] [,2] # [1,] 1 0 all((dlmFiltered$a %*% t(dlmFiltered$mod$FF)) == dlmFiltered$a[, 1]) # [1] TRUE
補足 カルマンフィルタの動きについては以前こんなエントリを書いた。2つ目のエントリにカルマンフィルタの予測 / フィルタ化状態がどういった感じで動いているか記載がある (平滑化は記載ない)。
予測誤差と予測誤差分散
最尤推定の尤度計算式は先のエントリに記載。計算に必要な (1期先) 予測誤差と予測誤差標準偏差は dlm:::residuals.dlmFiltered で求めることができる。
# 既定では標準化されるため、 type = 'raw' を指定して実値をとる resid <- residuals(dlmFiltered, type = 'raw') # 予測誤差 resid$res[1:12] # [1] 7.430707083 -3.827523556 0.111504168 -0.021616238 0.225665269 -0.044983932 # [7] 0.045295381 0.061233329 -0.021088564 0.049603994 0.270853650 0.007138991 # 予測誤差標準偏差 resid$sd[1:12] # [1] 4472.1359566 2236.0679824 0.1922632 0.1596384 0.1484746 0.1429560 # [7] 0.1396797 0.1375109 0.1359691 0.1348166 0.1339224 0.1332084
ふつうに計算して求めるなら、dlm::dlmFiltered インスタンスの各プロパティを利用して、
# 予測誤差 (y - (matrix(as.numeric(dlmFiltered$a), 192, 2) %*% t(dlmFiltered$mod$FF)))[1:12] # [1] 7.430707083 -3.827523556 0.111504168 -0.021616238 0.225665269 -0.044983932 # [7] 0.045295381 0.061233329 -0.021088564 0.049603994 0.270853650 0.007138991 # もしくは (y - dlmFiltered$f)[1:12] # [1] 7.430707083 -3.827523556 0.111504168 -0.021616238 0.225665269 -0.044983932 # [7] 0.045295381 0.061233329 -0.021088564 0.049603994 0.270853650 0.007138991 # 予測誤差標準偏差 f <- function(i) { FF <- dlmFiltered$mod$FF V <- dlmFiltered$mod$V diag(crossprod(dlmFiltered$D.R[i, ] * t(FF %*% dlmFiltered$U.R[[i]])) + V) } drop(t(sqrt(sapply(seq(along = dlmFiltered$U.R), f))))[1:12] # [1] 4472.1359566 2236.0679824 0.1922632 0.1596384 0.1484746 0.1429560 # [7] 0.1396797 0.1375109 0.1359691 0.1348166 0.1339224 0.1332084
最尤推定の可視化
最尤推定時のパラメータの変化を記録するため、dlm::dlmMLE を書き換えた関数を定義する。LL_ckbookcompat はテキスト記載の対数尤度 (に近いもの) を計算するための関数 ( 詳細はこちら )。
# optim 1 ステップごとに作成されたモデルを保存 models <<- list() dlmMLE_wrapper <- function (y, parm, build, method = "L-BFGS-B", ..., debug = FALSE) { logLik <- function(parm, ...) { mod <- build(parm, ...) # グローバル変数に追加 models[[length(models) + 1]] <<- mod return(dlmLL(y = y, mod = mod, debug = debug)) } return(optim(parm, logLik, method = method, ...)) } # 推定するパラメータ数が 2 のため、総和をとる範囲を変更 LL_ckbookcompat <- function(y, mod) { n <- length(y) filtered <- dlmFilter(y, mod) resid <- residuals_dlmcompat(filtered) F <- (as.numeric(resid$sd)^2)[3:n] v <- as.numeric(resid$res)[3:n] return(-0.5 * (n * log(2 * pi) + sum(log(F) + (v^2/F)))) }
続けて、アニメーション用の処理を定義して実行。
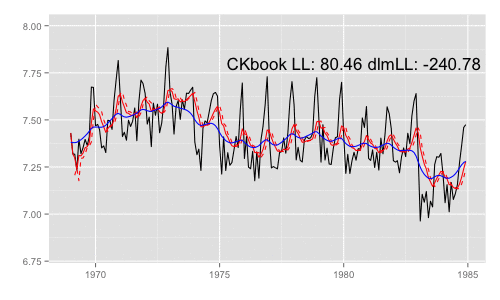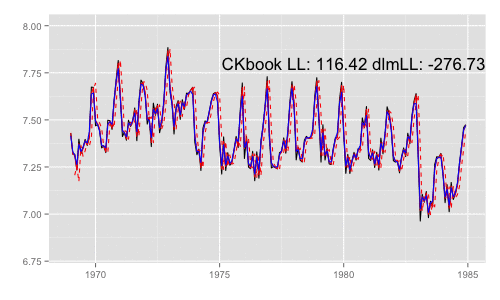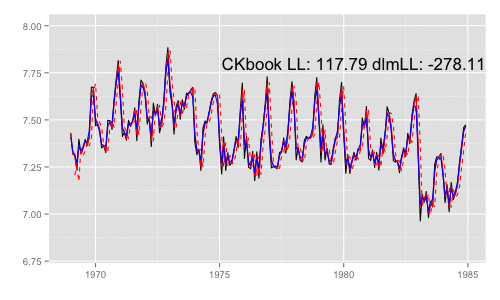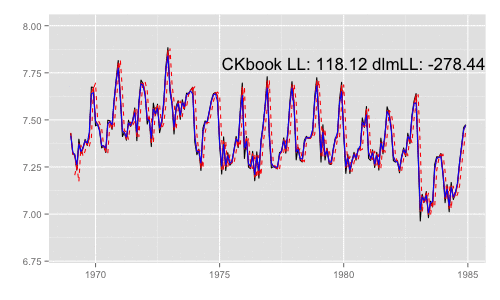
library(animation) models <<- list() dlmMLE_wrapper(y, parm = parm, build = buildFun) saveGIF({ for (mod in models[1:65]) { filtered <- dlmFilter(y, mod) p <- autoplot(filtered, fitted.colour = 'red', fitted.linetype = 'dashed') p <- autoplot(dlmSmooth(y, mod), colour = 'blue', p = p) label <- paste('CKbook LL:', round(LL_ckbookcompat(y, mod), 2), 'dlmLL:', round(dlmLL(y, mod), 2)) p <- p + annotate('text', label = label, x = as.Date('1980', '%Y'), y = 7.8, size = 6) print(p) Sys.sleep(0.01) } }, interval = 0.3, movie.name = "dlm.gif", ani.width = 500, ani.height = 300)
最尤推定時に各状態推定値がどのように変化していくかをみる。左側 "CKbookLL" はテキスト記載の対数尤度計算式 LL_ckbookcompat の値。右側 "dlmLL" は dlm::dlmLL の値 ( optim で最適化される値 )。
テキストに記載された数値より、収束時には左側の値が 0.6247935 x 観測時系列の長さ (192) = 119.9604 程度になるはずだ。
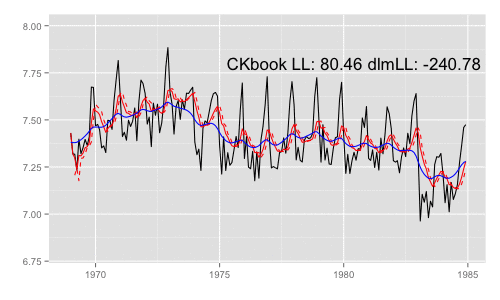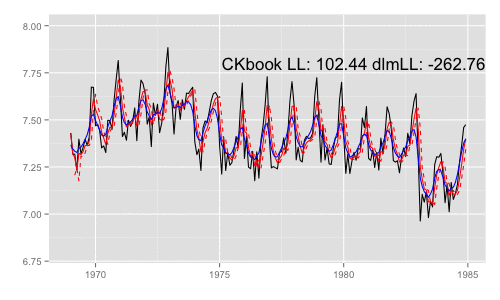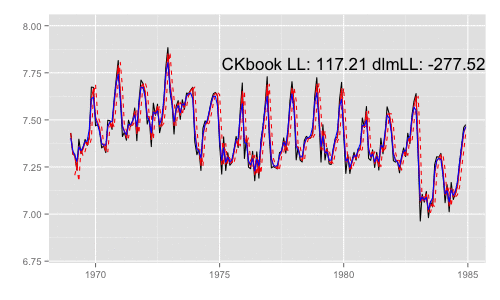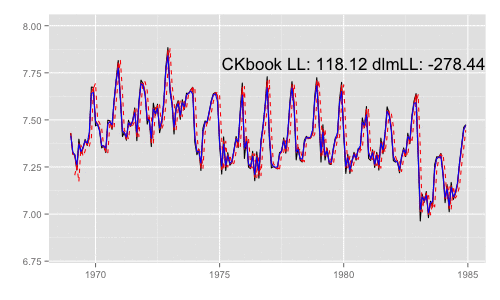
補足 {dlm} は既定では optim の際に method = 'L-BFGS-B' を利用する。可視化目的であれば Nelder-Mead 法なんかを使ったほうが 素人目には "最適化らしい" 動きになる気がする。
models <<- list() dlmMLE_wrapper(y, parm = parm, build = buildFun, method = 'Nelder-Mead') # 以降略